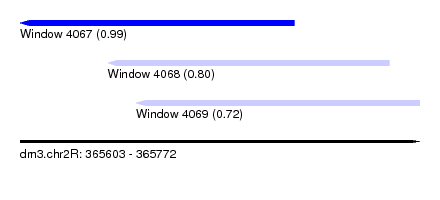
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 365,603 – 365,772 |
| Length | 169 |
| Max. P | 0.990410 |

| Location | 365,603 – 365,719 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 75.43 |
| Shannon entropy | 0.47067 |
| G+C content | 0.56493 |
| Mean single sequence MFE | -41.10 |
| Consensus MFE | -26.93 |
| Energy contribution | -29.95 |
| Covariance contribution | 3.02 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.66 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.990410 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 365603 116 - 21146708 AGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGUG-ACAACGAGGUGACUGUGUUGUGGACAGCAACUCGCCGCAACA-CACUUCA ((((((((((((.(((((.....))))).)))))(((.(((((((.(........).))))))))))-.......)))))))(((((((((.(........)))))))))-)...... ( -48.80, z-score = -3.66, R) >droSim1.chr3h_random 192768 116 + 1452968 AGUCACCGAUCGUGGCCACCACCUGGUCCAGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCG-ACAACGAGGUGACUGUGUUGUGGGCAGCAACUCGCCGCAACA-CACUUCA (((((((((((.((((((.....))).)))))))(((.(((((((.(........).))))))))))-.......)))))))(((((((((.(........)))))))))-)...... ( -49.70, z-score = -3.82, R) >droSec1.super_28 808529 116 + 873875 AUUCACCGAUCGUGGACACCACCUGAUCCCGAUCCGCACUCUGCACCACACCGCAGCUACAGAGGCG-UCAACGAGGUGACUGUGUUGUGGGCAGCAACUCGCCGCAACA-CACUUCA ..((((((((((.(((((.....)).))))))))(((.(((((................))))))))-.......))))).((((((((((.(........)))))))))-))..... ( -40.59, z-score = -2.05, R) >droYak2.chrU 2674141 117 - 28119190 AGUCACCUAUCGGGGCCACCACCUCGACCCGAUCCGCACUCAGUACCACACCAGAGCCACGGAUACG-ACAACAAGGUGGCUGUGUUGUGGGCAGAAACGUAUCUUAACACCAAUGCA ((((((((.((((((......))))))..(((((((..(((............)))...))))).))-......))))))))((((((((...(((......)))...)).)))))). ( -34.80, z-score = -0.67, R) >droEre2.scaffold_4784 25245384 117 - 25762168 AGUCACCGAUCGUGGCCACCACCUGGUCUCGAUCCGCGCUCUGUACCACACCAUAGCUACAGAGGUG-AUAUCGAGGUGACUGUGUUGUGGGCAGCAACGUGCCGCUACACCCAUGUA ((((((((((((.(((((.....))))).)))))(((.(((((((.(........).))))))))))-.......)))))))((((.((((.((......)))))).))))....... ( -47.20, z-score = -2.23, R) >droAna3.scaffold_13250 2521647 100 + 3535662 CACCAUUGAUCGUGGCCACCACCUGAACCUGAUCCGCACUCUGCAUCACUCUACAGUUGCAGAGGCAUUCAACAUAGUGACCG-AUCGUAG-UGACCGUGUA---------------- (((...(((((((((...)))).......(((...((.(((((((..(((....))))))))))))..)))...........)-))))..)-))........---------------- ( -25.50, z-score = -0.35, R) >consensus AGUCACCGAUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCG_ACAACGAGGUGACUGUGUUGUGGGCAGCAACGCGCCGCAACA_CACUUCA ((((((((((((.(((((.....))))).)))))(((.(((((((.(........).))))))))))........))))))).((((((((...........))))))))........ (-26.93 = -29.95 + 3.02)
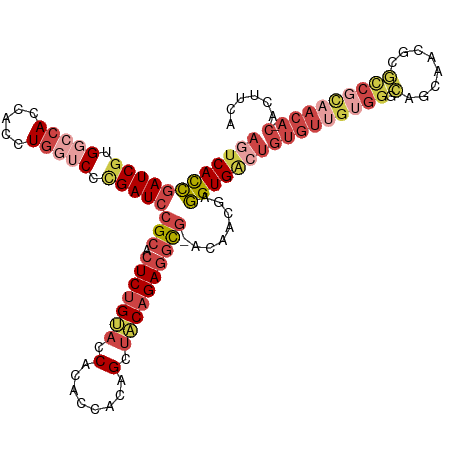
| Location | 365,640 – 365,759 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.42 |
| Shannon entropy | 0.45283 |
| G+C content | 0.55225 |
| Mean single sequence MFE | -34.20 |
| Consensus MFE | -20.67 |
| Energy contribution | -21.87 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.74 |
| SVM RNA-class probability | 0.803771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 365640 119 - 21146708 ACCCUGCUACCGGCUUAUUCCACUUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGUG-ACAACGAGGUGA .......((((((((....((....)).......))))...(((((((((((.(((((.....))))).))))).....((((((.(........).))))))))))-)).....)))). ( -35.70, z-score = -1.09, R) >droSim1.chr3h_random 192805 119 + 1452968 ACCCUGCUACCGGCUUAUUCCAGUUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCAGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCG-ACAACGAGGUGA .....((((((((((......)))))))......))).....(((((((((.((((((.....))).)))))))(((.(((((((.(........).))))))))))-.......))))) ( -39.00, z-score = -2.03, R) >droSec1.super_28 808566 119 + 873875 ACCCUGCUACCGACUUAUUCCAGUUGGUCACUUCAGCUAUAUUCACCGAUCGUGGACACCACCUGAUCCCGAUCCGCACUCUGCACCACACCGCAGCUACAGAGGCG-UCAACGAGGUGA .....((((((((((......)))))))......))).....((((((((((.(((((.....)).))))))))(((.(((((................))))))))-.......))))) ( -34.19, z-score = -2.06, R) >droYak2.chrU 2674179 119 - 28119190 ACCUUGCUACGGGCUUAUUCCAAUUGGUCACCUGAGCUACAGUCACCUAUCGGGGCCACCACCUCGACCCGAUCCGCACUCAGUACCACACCAGAGCCACGGAUACG-ACAACAAGGUGG ((((((...((((............(((...(((.....)))..)))..((((((......))))))))))(((((..(((............)))...)))))...-....)))))).. ( -31.90, z-score = -0.55, R) >droEre2.scaffold_4784 25245422 119 - 25762168 ACCCUGCUACCGGCUUAUUCCAGUUGGACACCUGAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCUCGAUCCGCGCUCUGUACCACACCAUAGCUACAGAGGUG-AUAUCGAGGUGA .....((((..((((((.(((....)))....)))))).))))(((((((((.(((((.....))))).)))))(((.(((((((.(........).))))))))))-.......)))). ( -41.20, z-score = -2.02, R) >droAna3.scaffold_13250 2521667 99 + 3535662 ---------------------ACCCUGCUAUCAGCACCAUCACCAUUGAUCGUGGCCACCACCUGAACCUGAUCCGCACUCUGCAUCACUCUACAGUUGCAGAGGCAUUCAACAUAGUGA ---------------------.....((((((((....((((....)))).((((...))))))))...(((...((.(((((((..(((....))))))))))))..)))...)))).. ( -23.20, z-score = -1.06, R) >consensus ACCCUGCUACCGGCUUAUUCCAGUUGGUCACCUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCG_ACAACGAGGUGA ..........................................((((((((((.(((((.....))))).)))))(((.(((((((.(........).))))))))))........))))) (-20.67 = -21.87 + 1.20)


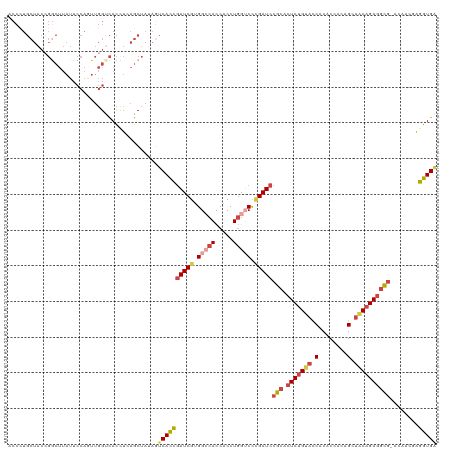
| Location | 365,652 – 365,772 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.22 |
| Shannon entropy | 0.40583 |
| G+C content | 0.56586 |
| Mean single sequence MFE | -33.54 |
| Consensus MFE | -22.91 |
| Energy contribution | -24.78 |
| Covariance contribution | 1.88 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.68 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.717390 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
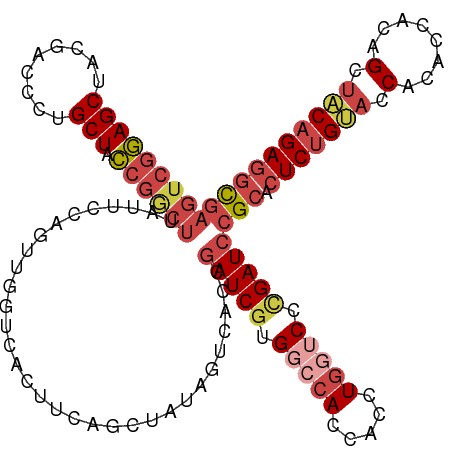
>dm3.chr2R 365652 120 - 21146708 AGUCGGAGCUACGACCCUGCUACCGGCUUAUUCCACUUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCUGAUCCGCACUCUGUACCACACCACAGCUACAGAGGUG (((((((((.........))).))))))..........(((.(((........))).)))(((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))) ( -38.10, z-score = -1.88, R) >droSim1.chr3h_random 192817 120 + 1452968 AGUCGGAGCUACGACCCUGCUACCGGCUUAUUCCAGUUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCAGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCG (((((((((.........))).))))))..........(((.(((........))).)))((((.((((((.....))).)))))))(((.(((((((.(........).)))))))))) ( -39.00, z-score = -2.08, R) >droSec1.super_28 808578 120 + 873875 AGUCGGAGCUACGACCCUGCUACCGACUUAUUCCAGUUGGUCACUUCAGCUAUAUUCACCGAUCGUGGACACCACCUGAUCCCGAUCCGCACUCUGCACCACACCGCAGCUACAGAGGCG (((((((((.........))).))))))......((((((.....)))))).........(((((.(((((.....)).))))))))(((.(((((................)))))))) ( -34.59, z-score = -2.23, R) >droYak2.chrU 2674191 120 - 28119190 AGUCAGAGCUACGACCUUGCUACGGGCUUAUUCCAAUUGGUCACCUGAGCUACAGUCACCUAUCGGGGCCACCACCUCGACCCGAUCCGCACUCAGUACCACACCAGAGCCACGGAUACG .(((..(((.........))).(((((((........((((...(((((.....(((.....((((((......))))))...))).....))))).)))).....))))).)))))... ( -28.12, z-score = 0.38, R) >droEre2.scaffold_4784 25245434 120 - 25762168 AGUCGGAGCUACGACCCUGCUACCGGCUUAUUCCAGUUGGACACCUGAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCUCGAUCCGCGCUCUGUACCACACCAUAGCUACAGAGGUG .((((......))))...((((..((((((.(((....)))....)))))).))))....(((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))) ( -39.00, z-score = -1.22, R) >droAna3.scaffold_13250 2521680 95 + 3535662 ----GAAGCUAAGACCCUGCUAUCAGC---------------------ACCAUCACCAUUGAUCGUGGCCACCACCUGAACCUGAUCCGCACUCUGCAUCACUCUACAGUUGCAGAGGCA ----..(((.........)))(((((.---------------------...((((....)))).((((...))))......)))))..((.(((((((..(((....)))))))))))). ( -22.40, z-score = -0.82, R) >consensus AGUCGGAGCUACGACCCUGCUACCGGCUUAUUCCAGUUGGUCACUUCAGCUAUAGUCACCGAUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCG (((((((((.........))).))))))................................(((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))) (-22.91 = -24.78 + 1.88)

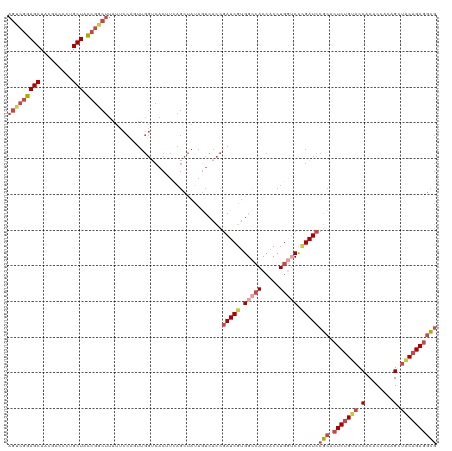
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:01:06 2011