
| Sequence ID | dm3.chr2RHet |
|---|---|
| Location | 627,499 – 627,613 |
| Length | 114 |
| Max. P | 0.994744 |

| Location | 627,499 – 627,613 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 79.54 |
| Shannon entropy | 0.39078 |
| G+C content | 0.58552 |
| Mean single sequence MFE | -46.67 |
| Consensus MFE | -31.52 |
| Energy contribution | -32.50 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.16 |
| Structure conservation index | 0.68 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.519783 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2RHet 627499 114 + 3288761 ---GUGUGUUGCGACACGUUGCUGCCCACAACA--CAGUCACCUCGUUGUCGCCUCUGUAGCUGUGGUGUGGUGCAGAGUGCGGAUCGGGAUCAGGUGGUGUCCACGAUCGGUGACUAU ---(((.((.((((....)))).)).)))....--.(((((((.......((((((((((.(........).))))))).)))(((((((((........)))).)))))))))))).. ( -47.90, z-score = -1.36, R) >droSim1.chrU 5586784 114 + 15797150 ---GUGUGUUGCGGCGAGUUGCUGCCCACAACA--CAGUCACCUCGUUGUCGCCUCUGUAGCUGUGGUGUGGUACAGAGUGCGGAUCUGGACCAGGUGGUGGCCACGAUCGGUGACUAU ---..((((((.((((......))))..)))))--)(((((((.......((((((((((.(........).))))))).)))(((((((.(((.....)))))).))))))))))).. ( -51.10, z-score = -2.09, R) >droSec1.super_6 4341880 114 - 4358794 ---GUGUGUUGCGGCACGUUGCUGCUCACAACA--CAGUCACCUCGUUGUCGCCUAUUUAGCUGUGGUGUGGUGCAGAGUGCGGAUCGUGAUCAGGUGGUGUCCACGAUCGGUGACUAU ---((((((.(((((.....)))))..)).)))--)(((((((.(((..((((((((...((....)))))).)).))..)))(((((((..((.....))..)))))))))))))).. ( -42.20, z-score = -0.32, R) >droYak2.chrU 2863716 110 + 28119190 ------UACCGCGGCACGUUGCUGCCCCCAGCA--CAGUCACCUCGUUGUCGCUUCUGUAGCUCUG-UACCACACAGAGUGCGGAUCGGGACCAGGUGGUGGCCACGAACGGUGACUAU ------....(((((.....)))))........--.(((((((((((.((((((((((((.(((((-(.....))))))))))))..(....)....)))))).))))..))))))).. ( -43.20, z-score = -0.89, R) >droEre2.scaffold_4784 23976750 114 + 25762168 ---GGGUGUUGCGGCACGUUGCUGCCCACAACA--CAGUCAACUGGAUGUCGCCUCUGUAGCUGUGGUGUGGUACAGAGUGCGGAUCGGGACCAGGUGGUGGCCACGAUCGGUGACUAU ---..((((((.((((......))))..)))))--)(((((.((......((((((((((.(........).))))))).)))(((((((.(((.....))))).)))))))))))).. ( -47.00, z-score = -0.74, R) >droAna3.scaffold_13417 3479180 119 + 6960332 GUGUACCGUGGCAGCAAGUGGCUGCCCACAACGGCCGGUCACUCGGCAGGUGCCUCUACAGUCAUGUAGCACUGCAGAGCACCGAUCAAGACCAGGUGGUUGCCACGAUCAGUGAUGAU ......((((((.((.((((((((.((.....)).))))))))..)).(((((.(((.((((........)))).))))))))......((((....))))))))))((((....)))) ( -48.60, z-score = -1.57, R) >consensus ___GUGUGUUGCGGCACGUUGCUGCCCACAACA__CAGUCACCUCGUUGUCGCCUCUGUAGCUGUGGUGUGGUACAGAGUGCGGAUCGGGACCAGGUGGUGGCCACGAUCGGUGACUAU ..........(((((.....)))))...........(((((((.......((((((((((.(........).))))))).)))((((((((((....)))..)).)))))))))))).. (-31.52 = -32.50 + 0.98)

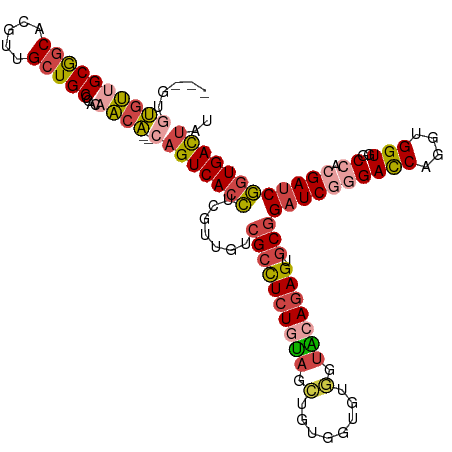
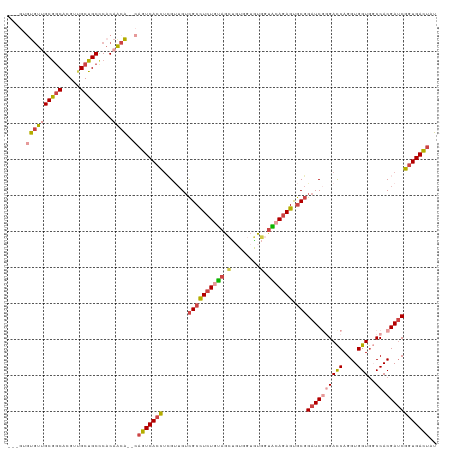
| Location | 627,499 – 627,613 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.54 |
| Shannon entropy | 0.39078 |
| G+C content | 0.58552 |
| Mean single sequence MFE | -45.18 |
| Consensus MFE | -35.77 |
| Energy contribution | -36.53 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.73 |
| SVM RNA-class probability | 0.994744 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2RHet 627499 114 - 3288761 AUAGUCACCGAUCGUGGACACCACCUGAUCCCGAUCCGCACUCUGCACCACACCACAGCUACAGAGGCGACAACGAGGUGACUG--UGUUGUGGGCAGCAACGUGUCGCAACACAC--- ..((((((((((((.(((((.....)).))))))))(((.(((((................)))))))).......)))))))(--((((((((.((......)))))))))))..--- ( -43.39, z-score = -2.43, R) >droSim1.chrU 5586784 114 - 15797150 AUAGUCACCGAUCGUGGCCACCACCUGGUCCAGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGACAACGAGGUGACUG--UGUUGUGGGCAGCAACUCGCCGCAACACAC--- ..(((((((((((.((((((.....))).)))))))(((.(((((((.(........).)))))))))).......)))))))(--((((((((.(........))))))))))..--- ( -49.90, z-score = -4.25, R) >droSec1.super_6 4341880 114 + 4358794 AUAGUCACCGAUCGUGGACACCACCUGAUCACGAUCCGCACUCUGCACCACACCACAGCUAAAUAGGCGACAACGAGGUGACUG--UGUUGUGAGCAGCAACGUGCCGCAACACAC--- ..(((((((((((((((.((.....)).))))))))(((.....((...........)).......))).......)))))))(--(((((((.(((......)))))))))))..--- ( -41.30, z-score = -3.18, R) >droYak2.chrU 2863716 110 - 28119190 AUAGUCACCGUUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUGUGGUA-CAGAGCUACAGAAGCGACAACGAGGUGACUG--UGCUGGGGGCAGCAACGUGCCGCGGUA------ (((((((((..(((.(((((.....))))).)))..(((..(((((((....-....)).))))).))).......))))))))--)((((((.((......)).)).)))).------ ( -40.60, z-score = 0.37, R) >droEre2.scaffold_4784 23976750 114 - 25762168 AUAGUCACCGAUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGUACCACACCACAGCUACAGAGGCGACAUCCAGUUGACUG--UGUUGUGGGCAGCAACGUGCCGCAACACCC--- ..(((((.((((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))).......).)))))(--((((((((.((......)))))))))))..--- ( -46.00, z-score = -3.37, R) >droAna3.scaffold_13417 3479180 119 - 6960332 AUCAUCACUGAUCGUGGCAACCACCUGGUCUUGAUCGGUGCUCUGCAGUGCUACAUGACUGUAGAGGCACCUGCCGAGUGACCGGCCGUUGUGGGCAGCCACUUGCUGCCACGGUACAC ........(((((((((((.(((((((((((((...(((((((((((((........)))))))).)))))...)))..)))))).....))))((((....))))))))))))).)). ( -49.90, z-score = -1.90, R) >consensus AUAGUCACCGAUCGUGGCCACCACCUGGUCCCGAUCCGCACUCUGCACCACACCACAGCUACAGAGGCGACAACGAGGUGACUG__UGUUGUGGGCAGCAACGUGCCGCAACACAC___ .(((((((((((((.(((((.....))))).)))))(((.(((((((.(........).)))))))))).......))))))))..(((((((.(((......))))))))))...... (-35.77 = -36.53 + 0.76)

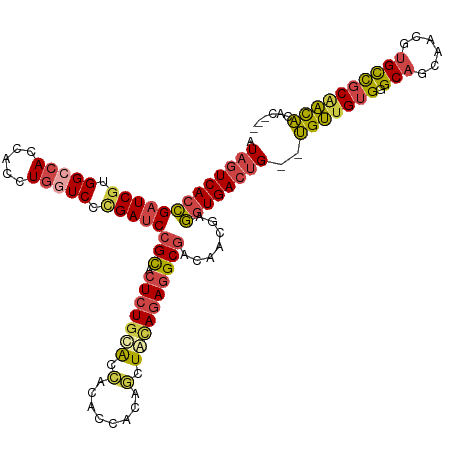
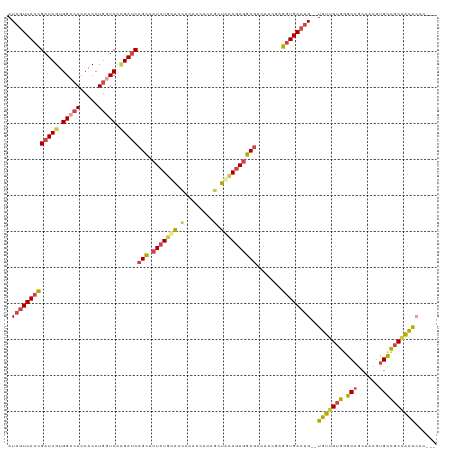
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:59:17 2011