| Sequence ID | dm3.chr2L |
|---|---|
| Location | 22,427,527 – 22,427,677 |
| Length | 150 |
| Max. P | 0.961551 |

| Location | 22,427,527 – 22,427,677 |
|---|---|
| Length | 150 |
| Sequences | 13 |
| Columns | 172 |
| Reading direction | reverse |
| Mean pairwise identity | 65.44 |
| Shannon entropy | 0.71656 |
| G+C content | 0.42756 |
| Mean single sequence MFE | -44.79 |
| Consensus MFE | -12.05 |
| Energy contribution | -12.29 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.27 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.961551 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 22427527 150 - 23011544 ACGAGCAACGGAAGACUAAGAUUGCAGCUCAUAAGGCAGCAUAUGUUGCCAAGCUGCAGUCUG---AAACUGAGGCUUAAA--AGUGUU--CGUCACCAG--CUUCA--GUUUAUAUU---GGUGUAAAAAUAAAUAAACAGUUCUAAUGACACCAGCGACAAU-------- ....((..(....)....(((((((((((.....((((((....)))))).))))))))))).---(((((((((((....--.(((..--...))).))--)))))--))))....(---(((((.......(((.....)))......))))))))......-------- ( -49.52, z-score = -4.29, R) >droEre2.scaffold_4845 20752459 150 + 22589142 ACGAGCAACGGAAGACUAAGAUUGCAGCUCAUAAGGCAGCUUAUGUUGCUAAGCUGCAAUCUG---AAACUGAGGCAUAAA--AGAAAU--CGUCACCCG--CUUCA--GUUUACAUU---GGUGUAAUAAUAAAUAAACAGUUGUAAUGACGCCAGCGGCAAU-------- ....((..(....)....(((((((((((.....((((((....)))))).))))))))))).---((((((((((.....--.(....--).......)--)))))--)))).(.((---(((((.((.((((........)))).)).))))))).)))...-------- ( -46.82, z-score = -3.37, R) >droYak2.chr2h_random 3597856 144 + 3774259 ACGAGCAACGGAAGACUAAGAUUGCAGCUCAUAAGGCAGCUUAUGUUGCCAAGCUCCAGUCUG---AAACUGAGGCCUAAA--AGUGAU--CGACACCCG--CUUUA--GUUGAGAUU---GGUGUAAAAAUAAAUAAACAGUUGUAAUGACACCAGC-------------- ....((..(....)......(((((((((.....((((((....))))))..((.(((((((.---.(((((((((.....--.(((..--...)))..)--)))))--))).)))))---)).))..............))))))))).......))-------------- ( -43.00, z-score = -3.06, R) >droSim1.chr2h_random 1245529 147 - 3178526 ACGAGCAACGGAAGACUAAGAUUGCAGCUCAUAAGGCAGCUUAUGUUGCAAAGCUGCAGUCUG---AAACUGAGGCUUGAA--AGUGAU--CGUCAUCAG--CUUCA--GUUUAGAUU---GGUGUAAAAAUAAAUAAACAGUUGUAAUGACACCAGCAAU----------- ....((..(....)....(((((((((((......(((((....)))))..))))))))))).---(((((((((((.((.--..((..--...))))))--)))))--))))....(---(((((....((((........))))....))))))))...----------- ( -48.60, z-score = -3.79, R) >droSec1.super_42 17277 147 - 298440 ACGAGCAACGGAAGACUAAGAUUGCAGCUCAUAAGGCAGCUUAUGUUGCAAAGCUGCAGUCUG---AAACUGAGGCUUGAA--AGUGAU--CGUCAUCAG--CUUCA--GUUUAGAUU---GGUGUAAAAAUAAAUAAACAGUUGUAAUGACACCAGCAAU----------- ....((..(....)....(((((((((((......(((((....)))))..))))))))))).---(((((((((((.((.--..((..--...))))))--)))))--))))....(---(((((....((((........))))....))))))))...----------- ( -48.60, z-score = -3.79, R) >droVir3.scaffold_12963 17769998 152 + 20206255 AUGAGGAGCGCAAGACCAAGAUCGCCGCCCACAAGGCUGCCUAUGUGGCUAAGCUUCAGUCUG---AGACUGAGGCCUAAG--GGCAUU--UGACUACCGU-UUGCA--GUGUGUAUUGGGGGUAAAA-AAGAAAUAAA--CAAACAAUUGUAAUGAGAAACAAU------- ....((..(....).))......(((((((....(((..(....)..)))..(((((((((..---.)))))))))....)--)))...--......(((.-.(((.--....))).))).)))....-..........--......(((((........)))))------- ( -38.60, z-score = 0.11, R) >droMoj3.scaffold_6500 19757651 157 + 32352404 AUGAGGAACGCAAGGCCAAGGUCGCCGCCCACAAGGCUGCUUAUGUGGCUAAGCUUCAGUCCG---AGACUGAGGCAUAAG--UGCAUU--UGACUAUCGU-UUGGAGUGUGUGUAUUGGGGGUGAAAGAAGAAAUAAACACAAACAGUUGUAAUAAAAAACAAU------- ....((..(....).))....(((((.((((...(((..(....)..)))..(((((((((..---.)))))))))(((.(--..((((--((((....))-)..)))))..).)))))))))))).....................(((((........)))))------- ( -43.20, z-score = -0.81, R) >droGri2.scaffold_15126 2575655 152 - 8399593 AUGAGGAUCGCAAGAUCAAGAUCGCCGCCCACAAGGCAGCCUAUGUGGCUAAGCUUCAGUCGG---AGACUGAGGCCUAAG--UGCAUUUCUGACUACCGU-UUGCA--GUGUGUAUU---GGUGAAAGAAAAAAUAAA--CAAACAAUUGUAAAGAAAAACAAU------- .....((((....))))....((((((((.....)))((((.....))))..(((((((((..---.)))))))))...((--(((((..(((.(......-..)))--).)))))))---))))).............--......(((((........)))))------- ( -44.80, z-score = -2.70, R) >droWil1.scaffold_180772 5641972 151 + 8906247 ACGAGCAGCGCAAGACCAAGAUUGCAGCCCACAAGGCCGCCUAUGUGGCCAAGCUUCAGUCGG---AGACUGAUGCCUAAGUCUGCAUUUAAGGGAAUUAUGCUGCUCUGUUUGUAUUGGGUGUGAAAAGAAGAGAAAA--UAAAUAACAA-AACAA--------------- ..((((((((.((..((..((.((((((......((((((....))))))..((.((((((..---.)))))).))....).))))).))...))..)).))))))))(((((...(((..(((..............)--))..)))..)-)))).--------------- ( -46.14, z-score = -1.88, R) >droAna3.scaffold_13258 769778 145 + 1584923 AUGAGCAGCGCAAGACCAAGAUUGCCGCUCACAAGGCGGCGUAUGUUGCAAAGCUUCAGUCUG---AAACUGAGGCUUAAU--AAUUUU--UUUCAUCC----------GUUUAUGU--AAAGCGUGAAGAAAAAUAAACAAUUCUAAUGACAAUGUCAAAUAU-------- .(((((.(.((((........))))))))))......(((((.((((...((((((((((...---..))))))))))...--..((((--(((((..(----------((((....--.))))))))))))))...............)))))))))......-------- ( -39.20, z-score = -2.33, R) >droPer1.super_16 1593390 145 + 1990440 ACGAGCAGCGCAAGACUAAGAUUGCCGCCCACAAGGCCGCCUAUGUGGCCAAGCUUCAGUCCG---AGACUGAGGCCUAGG--UGCGCU--UAUCACCUG--UUGCA--GUUCGUAUU-GGUGUGAAA-AAGAAAUAAA-------AAUUGUAGUGAGAAACAAU------- ..((((.(.((((........)))))((((....((((((....))))))..(((((((((..---.)))))))))...))--.)))))--).((((...--.((((--(((..((((-....(....-)...))))..-------)))))))))))........------- ( -46.40, z-score = -2.31, R) >dp4.chr4_group4 6464911 145 - 6586962 ACGAGCAGCGCAAGACUAAGAUUGCCGCCCACAAGGCCGCCUAUGUGGCCAAGCUUCAGUCCG---AGACUGAGGCCUAGG--UGCGCU--UAUCACCUG--UUGCA--GUUCGUAUU-GGUGUGAAA-AAGAAAUAAA-------AAUUGUAGUGAGAAACAAU------- ..((((.(.((((........)))))((((....((((((....))))))..(((((((((..---.)))))))))...))--.)))))--).((((...--.((((--(((..((((-....(....-)...))))..-------)))))))))))........------- ( -46.40, z-score = -2.31, R) >triCas2.ChLG10 8798610 170 + 8806720 UGGCCGAACGCAAGAACCGUGUCGCCCAAAAGAAAAAAUCUUUCAUUGCAAAGAUCCAAGAAGGACAGGCUGAGCCUUAGAUUUUUAUUUCUUUGUAUUA--UAUAAAAGAUUGAGUGUGAUCAAUAAAUGUUGGUGCACGGAGGGAGUGUUUUUAUUACACCAUCCAAAGU (((.(((.(((.......)))))).)))........(((((((.((((((((((...(((((....((((...))))....)))))...))))))))...--.)).)))))))..((((.((((((....))))))))))(((.((.(((.......))).)).)))..... ( -41.00, z-score = -1.18, R) >consensus ACGAGCAACGCAAGACUAAGAUUGCCGCCCACAAGGCAGCUUAUGUUGCCAAGCUUCAGUCUG___AGACUGAGGCCUAAA__AGCAUU__UGUCAACCG__CUGCA__GUUUGUAUU___GGUGUAAAAAUAAAUAAACAGUUGUAAUGACAACAACAAACAA________ ........(....)...............(((...(((((....)))))...(((.((((........)))).)))..............................................)))............................................... (-12.05 = -12.29 + 0.24)

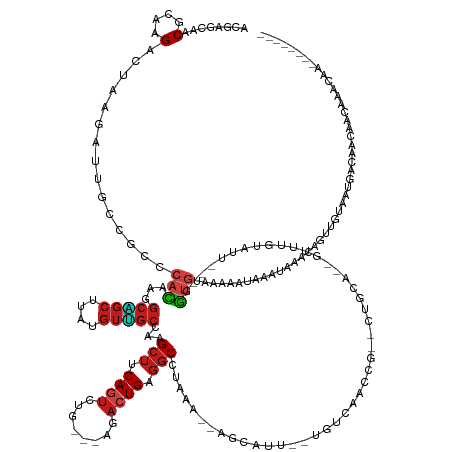
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:58:08 2011