
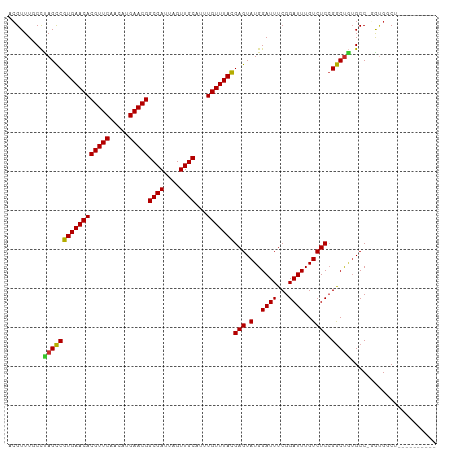
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 22,070,070 – 22,070,194 |
| Length | 124 |
| Max. P | 0.613682 |

| Location | 22,070,070 – 22,070,169 |
|---|---|
| Length | 99 |
| Sequences | 9 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 88.55 |
| Shannon entropy | 0.22568 |
| G+C content | 0.49178 |
| Mean single sequence MFE | -31.39 |
| Consensus MFE | -26.66 |
| Energy contribution | -26.17 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.21 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.529501 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 22070070 99 - 23011544 ACGUUU-CCCAGCCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCAGGUGGCU---------- ......-..(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).(((....))).---------- ( -30.60, z-score = -1.06, R) >droSim1.chr2L 21628764 99 - 22036055 UCGUUU-CCCAGCCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCAGGUGGCC---------- ......-..(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).(((....))).---------- ( -30.20, z-score = -0.94, R) >droSec1.super_28 668838 99 - 873875 UCGUUU-CCCAGCCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCAGGUGGCC---------- ......-..(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).(((....))).---------- ( -30.20, z-score = -0.94, R) >droYak2.chr2R 20410963 99 + 21139217 ACGUUU-CUCAGUCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCAGACUCUGCCAGGUGGCU---------- ......-...((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))..(((....))).---------- ( -26.20, z-score = -0.39, R) >droAna3.scaffold_12943 2270143 100 + 5039921 ACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCC--AGGUCGCU-------- .(((((((.(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).).)--))).))..-------- ( -31.10, z-score = -1.12, R) >dp4.chr4_group1 1470506 108 - 5278887 ACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCC--AUGCCAGUGGAUCGCA .((((..(.....(((((((((((((.....)))))((((....))))...)))))))).((((((((.(((((......)))))..)).))--))))..)..)).)).. ( -33.80, z-score = -0.98, R) >droPer1.super_5 3076391 105 + 6813705 ACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCC--AUGCCAGUGGAGA--- .((....((..(((((((((((((((.....)))))((((....))))...)))))))).))..))....))......((((((.(((.((.--..)))))))))))--- ( -34.60, z-score = -1.81, R) >droWil1.scaffold_181071 524450 98 - 1211509 ACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCCA-GCC-GGCGGCU---------- .((....((..(((((((((((((((.....)))))((((....))))...)))))))).))..))....)).....(((.((((...-.))-)).))).---------- ( -34.20, z-score = -2.01, R) >droVir3.scaffold_12963 15009712 98 + 20206255 AGGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCC-UGUGGC----------- ((((..(((..(((((((((((((((.....)))))((((....))))...)))))))).)).........(((......))))))...)))-).....----------- ( -31.60, z-score = -1.61, R) >consensus ACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCC_GGUGGCU__________ .........(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).)))))...................... (-26.66 = -26.17 + -0.49)


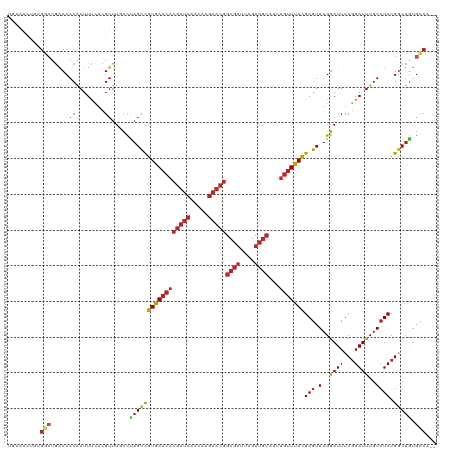
| Location | 22,070,077 – 22,070,194 |
|---|---|
| Length | 117 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.55 |
| Shannon entropy | 0.35794 |
| G+C content | 0.47212 |
| Mean single sequence MFE | -34.01 |
| Consensus MFE | -23.02 |
| Energy contribution | -23.34 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.68 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.613682 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 22070077 117 - 23011544 UGAAACUCUGGCUGACCACGAUUCCACGUUU-CCCAGCCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCA-- ....((((.(((((...(((......)))..-..)))))((((((((((((.....)))))((((....))))...))))))))))).(((((.(((((......)))))..)).)))-- ( -33.60, z-score = -0.94, R) >droSim1.chr2L 21628771 117 - 22036055 UGAAGCUCUGGCUGACCACGAUUCCUCGUUU-CCCAGCCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCA-- ....((((.(((((...((((....))))..-..)))))((((((((((((.....)))))((((....))))...))))))))))).(((((.(((((......)))))..)).)))-- ( -35.30, z-score = -1.19, R) >droSec1.super_28 668845 117 - 873875 UGAAACUCUGGCUGACCACGAUUCCUCGUUU-CCCAGCCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCA-- ....((((.(((((...((((....))))..-..)))))((((((((((((.....)))))((((....))))...))))))))))).(((((.(((((......)))))..)).)))-- ( -35.00, z-score = -1.40, R) >droYak2.chr2R 20410970 117 + 21139217 UGAAGCUGUGGCUGGCCACGAUUCCACGUUU-CUCAGUCGUGAACACGUUCAACACGAACGGCGAUUAGUUGCAUUUGUUUAUGAGUAUGGAUUUCGGAUUUGUCUCAGACUCUGCCA-- (((..(.((((....))))((.(((....((-(..(.((((((((((((((.....)))))((((....))))...))))))))).)..)))....))).)))..)))..........-- ( -31.40, z-score = 0.04, R) >droEre2.scaffold_4845 20188143 89 + 22589142 UGAAACUGUGGCUGACCACGAUUCCACGUUU-UCCAGCCGUAAA----------------------------CAUUUGCUUAUGAGUAUGAAUUUCGGAUUUGUAUCCGACUGUGCCA-- .........(((((...(((......)))..-..)))))((((.----------------------------...))))...((.(((((....((((((....)))))).)))))))-- ( -18.20, z-score = -0.28, R) >droAna3.scaffold_12943 2270150 115 + 5039921 ---CCGUGUGGCUGACCACAAUUUCACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCA-- ---..(((..((((...((((..((.((....((..(((((((((((((((.....)))))((((....))))...)))))))).))..))....)))).))))...))))..)))..-- ( -36.40, z-score = -1.56, R) >dp4.chr4_group1 1470519 120 - 5278887 UGGAACUGUGGCUGACCACAAUUUCACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCAUG (((((.(((((....))))).))))).....((.(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).)).... ( -39.80, z-score = -2.37, R) >droPer1.super_5 3076401 120 + 6813705 UGGAACUGUGGCUGACCACAAUUUCACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCAUG (((((.(((((....))))).))))).....((.(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).)).... ( -39.80, z-score = -2.37, R) >droWil1.scaffold_181071 524457 116 - 1211509 UGAGAUUGAAACUGGCCACAAUUUUACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCCA-GCC--- ...........((((((..........(....((..(((((((((((((((.....)))))((((....))))...)))))))).))..))....)(((......))))))))-)..--- ( -36.10, z-score = -2.53, R) >droVir3.scaffold_12963 15009718 114 + 20206255 ---GAUUUGGUGAAGCCACAAUUUCAGGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCC--- ---.....(((.(((((.........))))))))(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).)))))....--- ( -34.20, z-score = -1.46, R) >droGri2.scaffold_15252 9342563 114 + 17193109 ---GAUUUGGUGUAGCCACAAUUUCAGGUUUGCCUAGCUGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGCGAC--- ---.......(((((((.....((((((....))).(((((((((((((((.....)))))((((....))))...))))))).)))..)))....(((......))))))))))..--- ( -34.30, z-score = -1.59, R) >consensus UGAAACUGUGGCUGACCACAAUUUCACGUUUGCCUAGCCGUGAACACGUUCAACAUGAACGGCGAUUAGUUGCAUUUGUUUACGAGUAUGGAUUUCGGAUUUGUCUCCGGCUGUGCCA__ .........(((.....((........)).....(((((((((((((((((.....)))))((((....))))...)))))))(((.(..((((...))))..)))).))))).)))... (-23.02 = -23.34 + 0.32)

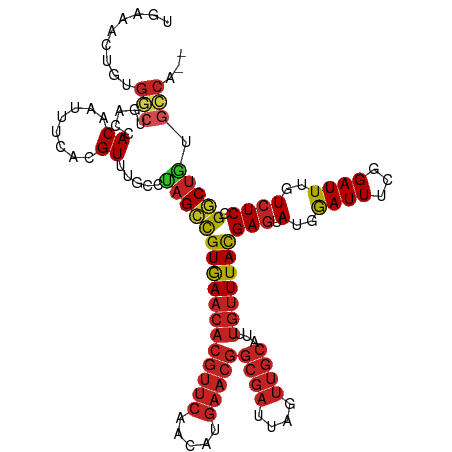
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:57:37 2011