
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 21,680,617 – 21,680,756 |
| Length | 139 |
| Max. P | 0.996443 |

| Location | 21,680,617 – 21,680,756 |
|---|---|
| Length | 139 |
| Sequences | 12 |
| Columns | 163 |
| Reading direction | forward |
| Mean pairwise identity | 61.65 |
| Shannon entropy | 0.79756 |
| G+C content | 0.48768 |
| Mean single sequence MFE | -43.50 |
| Consensus MFE | -12.66 |
| Energy contribution | -12.05 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.48 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.29 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.792778 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
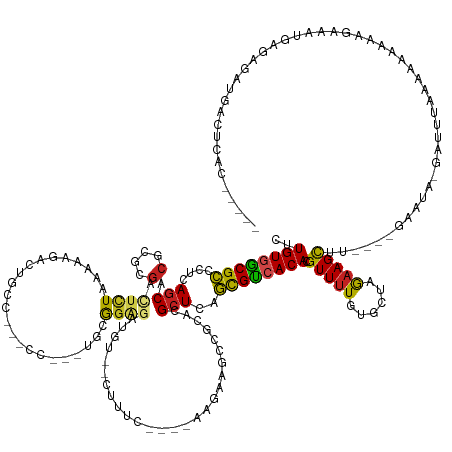
>dm3.chr2L 21680617 139 + 23011544 CUUGUGGCGCCGUCAGCACGAGAGACUCUAAAAGGACUUUC---CU---UGCGGAGAUGU--CUUUC----AAGAAGCCGCCCGCUAAGCGCCACACGUUUUGUGGUAGAAGUUU----GAAAA-CAUUUACAAACAAA--CUUGAGACAUGACUCAC----- ..((((((((.........((((((((((..((((.....)---))---)....))).))--)))))----....(((.....)))..))))))))...(((((.(((((.(((.----...))-).)))))..)))))--..((((......)))).----- ( -41.60, z-score = -1.94, R) >droGri2.scaffold_15252 15550467 129 - 17193109 CUUGUGGCGCUUUCAGCACGCGCGGCUCUUGAGA---------------UUCGAGAA--------UC--GGAAGAGUUCGCACGCUCAGCGUCACACGUUUUGUGCUAAAAGCAA----AGGGAUGCGGAGCGGAGUGGCGGAUUGGUUAAAACUUAC----- ....((((.(..((.((.((((((((((((..((---------------((....))--------))--..))))).)))).(((((.(((((....(((((......)))))..----...))))).)))))..))))).))..)))))........----- ( -44.50, z-score = -0.80, R) >droMoj3.scaffold_6500 11528054 156 + 32352404 CUUGUGGCGCUAUCAGCACGCGCGGUUCUAGCUGAACAGCCACGCCCCUUGCAGGUUUGUGACUGUCCAAAAAGAAGUCGCACGCUCAGCGUCACACGUUUUGUGCUAAAAGCGU------AUA-GAUUGAUAUAGAUAUAUACAAGUAUUAGUUUAGCUUAC ...((((((((...(((..(.((((((((...((.((((.((((..((.....))..)))).)))).))...)))).)))).)))).))))))))(((((((......)))))))------.((-(((((((((............)))))))))))...... ( -47.00, z-score = -1.21, R) >droVir3.scaffold_12963 83588 153 + 20206255 CUUGUGGCGCCGUCAGCACGCGCGGUUCUAGCGAAGCUGUCGC-CCUUUGAAGGGGAGGCAGCGAUC--AAAAGAAGUCGCACGCUCAGCGUCACACGUUUUGUGCUAAAAGCAA----GCAAAUGGGUUGCAGUUAUGUUAAUACGCGGCGAUUCUUAC--- .......((((((.(((((..((((((((...((.((((((.(-(((.....)))).))))))..))--...)))).))))((((...))))..........)))))....((((----.(.....).))))..............))))))........--- ( -50.90, z-score = 0.28, R) >droWil1.scaffold_180772 250330 127 - 8906247 CUUGUGACGUCUUCAUCGCGCAUAGUUCUCUCUC-------------UUAAAAGAGA--------------GAGAAACUGCACGCUCAACGUCACACGUUUUGUGCUAAAAGCAU----GGAAAUACUAUACGUAUUAGGGAGGGAAGUGGGACUUAC----- ..((((((((......((.(((...(((((((((-------------(....)))))--------------)))))..))).))....))))))))...((..(((.....))).----.))(((((.....))))).....................----- ( -38.60, z-score = -2.53, R) >dp4.chr4_group5 2168528 147 - 2436548 CUUGUAGCGCCCUCAGCACGCGCGACUCUAAAAAAGGGGCC----CCCGUACGGGGGAAC---UUUC----AAGAUGUCGCACGCUCAGCGUCACACGUUUUGUGCUAGAAGCGUGUUAGUGUAAGGAAUGGAGUGGGCGACACGAGGGAUGACUCAC----- (((((..(((((.((((((((((((((((....((((..((----(((....)))))..)---))).----.))).))))).(((..((((.....))))..)))......))))))).((.......))...).)))))..)))))...........----- ( -57.30, z-score = -2.01, R) >droPer1.super_5 2040130 146 - 6813705 CUUGUAGCGCCCUCAGCACGCGCGACUCUAAAAAAGGGGCC----CCCGUACGGAGGAAC---UUUC----AAGAUGUCGCACGCUCAGCGUCACACGUUUUGUGCUAGAAGCGUGUUAGUGUAAGGAAUGGAGUGG-CGACACGAGGGAUGACUCAC----- ..((.((((((((((((((((((((((((....((((..((----.((....)).))..)---))).----.))).))))).(((..((((.....))))..)))......)))))))...............(((.-...)))))))).)).)))).----- ( -51.10, z-score = -0.81, R) >droSim1.chr2L 21236453 141 + 22036055 CUUGUGGCGCCCUCAGCACGAGAGACUCUUAAAGGACUUUC---CU---UGCGGAGAUGU--CUUUC----AAGAAGCCGACCGCUCAGCGCCACACGUUUUGUGGUAGAAGUUU----GAAUA-CAUUUAAAAAAAAAACUUUGAAAUAUAACUCAC----- ..((((((((.........(((((((((((.((((.....)---))---)...)))).))--)))))----..((.((.....)))).)))))))).(((.(((....(((((((----.....-............)))))))...))).)))....----- ( -41.03, z-score = -2.74, R) >droSec1.super_28 223096 141 + 873875 CUUGUGGCGCCCUCAGCACGAGAGACUCUUAAAGGACUUUC---CU---UGCGGAGAUGU--CUUUC----AAGAAGCCGACCGCUCAGCGCCACACGUUUUGUGGUAGAAGUUU----GAAUA-CAUUUAAAAAAAAAACUUUGAAAUAUAACUCAC----- ..((((((((.........(((((((((((.((((.....)---))---)...)))).))--)))))----..((.((.....)))).)))))))).(((.(((....(((((((----.....-............)))))))...))).)))....----- ( -41.03, z-score = -2.74, R) >droYak2.chr2R 19977017 144 - 21139217 CUUGUGGCGCUUUCAGCACGAGCGACUUUAAAAAGAAUGCC---CCCCUUGGGGAGAUGU--CUUUC----AAGAAGCCGCCCGCUCAGUGCCACACGUUUUGUGGUAGAAGUUU----AAAUA-GAUUUAAAACAUUCCCUAUAAGACACGACUCAC----- ..(((((((((...(((..(.(((.((((..(((((...((---((....)))).....)--)))).----..)))).)))).))).))))))))).(((((((((..(((.(((----(((..-..))))))...))).))))))))).........----- ( -44.60, z-score = -3.24, R) >droEre2.scaffold_4845 19744386 136 - 22589142 CUUGUGGCGCCCUCAGCACGAGCGACACUAAAAAGACUGCC---CC---UGGGGAGAAGU--CUUUC----AAGAAGCCGCCCGCUCAGCGCCACACGUUAUGUGGUAGAAGUUU----GAAUA-UAUUUAAAAAA----CAAUGAGA-ACGACUUAC----- ..((((((((....(((.((.((....((..(((((((.((---(.---..)))...)))--)))).----.))..))))...)))..))))))))((((.(((.....((((..----.....-.)))).....)----)).....)-)))......----- ( -35.20, z-score = -0.92, R) >droAna3.scaffold_12916 9277409 128 + 16180835 CUUGUGGCGCCCUCAGCACGCGCGACUCUAAAGA-------------CAAUUAG----------UCC----AAGAUGCCGCACGCUCAGCGUCACACGUUUUGUGCUAGAAGCGU-UAACUGCAAACUAACUAACC--UUAAGUAUAUGAUAACUUAC----- ..((((((((....(((..(((.(..(((...((-------------(.....)----------)).----.)))..))))..)))..)))))))).(((.((((((..(((.((-((.............)))))--)).)))))).))).......----- ( -29.12, z-score = 0.06, R) >consensus CUUGUGGCGCCCUCAGCACGCGCGACUCUAAAAAGACUGCC___CC___UGCGGAGAUGU__CUUUC____AAGAAGCCGCACGCUCAGCGUCACACGUUUUGUGCUAGAAGCUU____GAAUA_GAUUUAAAAAAAAAGAAAUGAGAGAUGACUCAC_____ ..((((((((....(((.(....).((((.......................))))...........................)))..)))))))).(((((......))))).................................................. (-12.66 = -12.05 + -0.61)

| Location | 21,680,617 – 21,680,756 |
|---|---|
| Length | 139 |
| Sequences | 12 |
| Columns | 163 |
| Reading direction | reverse |
| Mean pairwise identity | 61.65 |
| Shannon entropy | 0.79756 |
| G+C content | 0.48768 |
| Mean single sequence MFE | -45.70 |
| Consensus MFE | -14.64 |
| Energy contribution | -14.04 |
| Covariance contribution | -0.60 |
| Combinations/Pair | 1.53 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.32 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.93 |
| SVM RNA-class probability | 0.996443 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
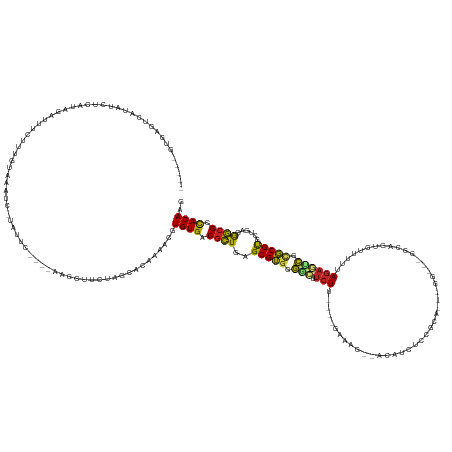
>dm3.chr2L 21680617 139 - 23011544 -----GUGAGUCAUGUCUCAAG--UUUGUUUGUAAAUG-UUUUC----AAACUUCUACCACAAAACGUGUGGCGCUUAGCGGGCGGCUUCUU----GAAAG--ACAUCUCCGCA---AG---GAAAGUCCUUUUAGAGUCUCUCGUGCUGACGGCGCCACAAG -----.((((......)))).(--(((....(((...(-((...----.)))...)))....)))).(((((((((..(((((.((((.((.----(((.(--((...(((...---.)---))..))).))).)))))).)))))......))))))))).. ( -45.00, z-score = -2.40, R) >droGri2.scaffold_15252 15550467 129 + 17193109 -----GUAAGUUUUAACCAAUCCGCCACUCCGCUCCGCAUCCCU----UUGCUUUUAGCACAAAACGUGUGACGCUGAGCGUGCGAACUCUUCC--GA--------UUCUCGAA---------------UCUCAAGAGCCGCGCGUGCUGAAAGCGCCACAAG -----....(((((.........((......))...(((.....----.))).........))))).((((.((((..(((((((..(((((..--((--------((....))---------------))..))))).)))))))......)))).)))).. ( -34.40, z-score = -1.25, R) >droMoj3.scaffold_6500 11528054 156 - 32352404 GUAAGCUAAACUAAUACUUGUAUAUAUCUAUAUCAAUC-UAU------ACGCUUUUAGCACAAAACGUGUGACGCUGAGCGUGCGACUUCUUUUUGGACAGUCACAAACCUGCAAGGGGCGUGGCUGUUCAGCUAGAACCGCGCGUGCUGAUAGCGCCACAAG ................(((((.......((((......-)))------)((((.(((((((..........((((...))))(((..((((..((((((((((((...(((....)))..))))))))))))..))))...)))))))))).))))..))))) ( -51.10, z-score = -2.47, R) >droVir3.scaffold_12963 83588 153 - 20206255 ---GUAAGAAUCGCCGCGUAUUAACAUAACUGCAACCCAUUUGC----UUGCUUUUAGCACAAAACGUGUGACGCUGAGCGUGCGACUUCUUUU--GAUCGCUGCCUCCCCUUCAAAGG-GCGACAGCUUCGCUAGAACCGCGCGUGCUGACGGCGCCACAAG ---........((((((((....((((....((((.....))))----.(((.....)))......)))).))))...(((((((..((((..(--((..((((..((((((....)))-).)))))).)))..)))).)))))))......))))....... ( -46.40, z-score = -0.60, R) >droWil1.scaffold_180772 250330 127 + 8906247 -----GUAAGUCCCACUUCCCUCCCUAAUACGUAUAGUAUUUCC----AUGCUUUUAGCACAAAACGUGUGACGUUGAGCGUGCAGUUUCUC--------------UCUCUUUUAA-------------GAGAGAGAACUAUGCGCGAUGAAGACGUCACAAG -----.....................(((((.....)))))...----.(((.....))).......(((((((((...((((((..(((((--------------(((((....)-------------)))))))))...)))))).....))))))))).. ( -42.20, z-score = -5.22, R) >dp4.chr4_group5 2168528 147 + 2436548 -----GUGAGUCAUCCCUCGUGUCGCCCACUCCAUUCCUUACACUAACACGCUUCUAGCACAAAACGUGUGACGCUGAGCGUGCGACAUCUU----GAAA---GUUCCCCCGUACGGG----GGCCCCUUUUUUAGAGUCGCGCGUGCUGAGGGCGCUACAAG -----..(((......)))(((.(((((...................((.((.((..((((.....)))))).)))).(((((((((.(((.----((((---(..(((((....)))----))...)))))..)))))))))))).....))))).)))... ( -54.00, z-score = -3.02, R) >droPer1.super_5 2040130 146 + 6813705 -----GUGAGUCAUCCCUCGUGUCG-CCACUCCAUUCCUUACACUAACACGCUUCUAGCACAAAACGUGUGACGCUGAGCGUGCGACAUCUU----GAAA---GUUCCUCCGUACGGG----GGCCCCUUUUUUAGAGUCGCGCGUGCUGAGGGCGCUACAAG -----..(((......)))(((.((-((.(((...............((.((.((..((((.....)))))).)))).(((((((((.(((.----((((---(..(((((....)))----))...)))))..))))))))))))...))))))).)))... ( -50.60, z-score = -1.99, R) >droSim1.chr2L 21236453 141 - 22036055 -----GUGAGUUAUAUUUCAAAGUUUUUUUUUUAAAUG-UAUUC----AAACUUCUACCACAAAACGUGUGGCGCUGAGCGGUCGGCUUCUU----GAAAG--ACAUCUCCGCA---AG---GAAAGUCCUUUAAGAGUCUCUCGUGCUGAGGGCGCCACAAG -----.(((((.((((((.((((.....)))).)))))-)))))----)..................(((((((((..((((..((((.(((----(((.(--((...(((...---.)---))..))).))))))))))..))))......))))))))).. ( -44.20, z-score = -2.96, R) >droSec1.super_28 223096 141 - 873875 -----GUGAGUUAUAUUUCAAAGUUUUUUUUUUAAAUG-UAUUC----AAACUUCUACCACAAAACGUGUGGCGCUGAGCGGUCGGCUUCUU----GAAAG--ACAUCUCCGCA---AG---GAAAGUCCUUUAAGAGUCUCUCGUGCUGAGGGCGCCACAAG -----.(((((.((((((.((((.....)))).)))))-)))))----)..................(((((((((..((((..((((.(((----(((.(--((...(((...---.)---))..))).))))))))))..))))......))))))))).. ( -44.20, z-score = -2.96, R) >droYak2.chr2R 19977017 144 + 21139217 -----GUGAGUCGUGUCUUAUAGGGAAUGUUUUAAAUC-UAUUU----AAACUUCUACCACAAAACGUGUGGCACUGAGCGGGCGGCUUCUU----GAAAG--ACAUCUCCCCAAGGGG---GGCAUUCUUUUUAAAGUCGCUCGUGCUGAAAGCGCCACAAG -----(((.((.(.((...(((((.((....))...))-)))..----..))..).)))))......((((((.....(((((((((((...----(((((--(...(((((....)))---))...))))))..)))))))))))((.....)))))))).. ( -48.10, z-score = -2.86, R) >droEre2.scaffold_4845 19744386 136 + 22589142 -----GUAAGUCGU-UCUCAUUG----UUUUUUAAAUA-UAUUC----AAACUUCUACCACAUAACGUGUGGCGCUGAGCGGGCGGCUUCUU----GAAAG--ACUUCUCCCCA---GG---GGCAGUCUUUUUAGUGUCGCUCGUGCUGAGGGCGCCACAAG -----....(.(((-(((((...----...........-.....----.........((((((...)))))).((...(((((((((..((.----(((((--(((.((((...---))---)).)))))))).)).))))))))))))))))))).)..... ( -47.10, z-score = -3.18, R) >droAna3.scaffold_12916 9277409 128 - 16180835 -----GUAAGUUAUCAUAUACUUAA--GGUUAGUUAGUUUGCAGUUA-ACGCUUCUAGCACAAAACGUGUGACGCUGAGCGUGCGGCAUCUU----GGA----------CUAAUUG-------------UCUUUAGAGUCGCGCGUGCUGAGGGCGCCACAAG -----.(((((........))))).--(((..(((((.......)))-))(((..((((((.....(((((((((((......)))).(((.----(((----------(.....)-------------)))..))))))))))))))))..))))))..... ( -41.10, z-score = -1.11, R) >consensus _____GUGAGUCAUAUCUCAUAGAUUUCUUUGUAAAUC_UAUUC____AAGCUUCUAGCACAAAACGUGUGACGCUGAGCGUGCGGCUUCUU____GAAAG__ACAUCUCCGCA___GG___GGCAGUCUUUUUAGAGUCGCGCGUGCUGAGGGCGCCACAAG ...................................................................((((.((((..(((((.(((.(((...........................................)))))).)))))......)))).)))).. (-14.64 = -14.04 + -0.60)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:56:46 2011