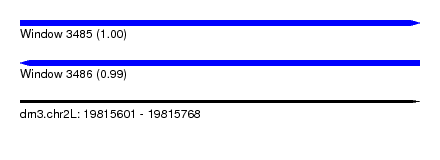
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 19,815,601 – 19,815,768 |
| Length | 167 |
| Max. P | 0.999122 |

| Location | 19,815,601 – 19,815,768 |
|---|---|
| Length | 167 |
| Sequences | 13 |
| Columns | 173 |
| Reading direction | forward |
| Mean pairwise identity | 90.17 |
| Shannon entropy | 0.23326 |
| G+C content | 0.45678 |
| Mean single sequence MFE | -57.74 |
| Consensus MFE | -48.05 |
| Energy contribution | -48.15 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.16 |
| Mean z-score | -4.18 |
| Structure conservation index | 0.83 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.66 |
| SVM RNA-class probability | 0.999122 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
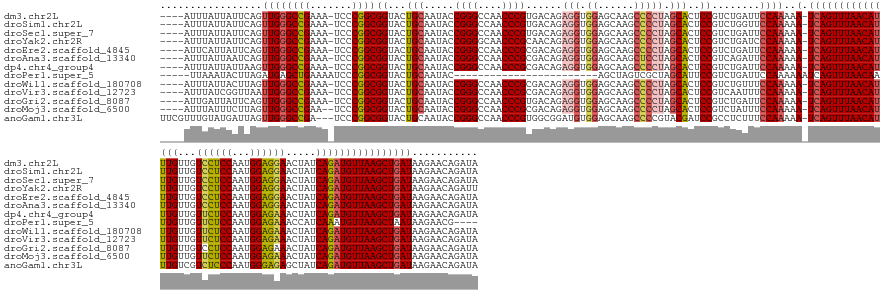
>dm3.chr2L 19815601 167 + 23011544 ----AUUUAUUAUUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----............(((((((((...-...))))((((......))))...)))))..(((....(((.((......)))))..)))...(((((.(((.....(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))). ( -59.90, z-score = -4.33, R) >droSim1.chr2L 19474822 167 + 22036055 ----AUUUAUUAUUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGGUUCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----............(((((((((...-...))))((((......))))...)))))..(((....(((.((......)))))..)))...(((((.(((.....(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))). ( -59.90, z-score = -3.91, R) >droSec1.super_7 3421298 167 + 3727775 ----AUUUAUUAUUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----............(((((((((...-...))))((((......))))...)))))..(((....(((.((......)))))..)))...(((((.(((.....(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))). ( -59.90, z-score = -4.33, R) >droYak2.chr2R 6280986 167 + 21139217 ----AUUUAUUAUUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGGCAACCCGCAACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUCCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUU ----............(((((((((...-...))))(((..(((..(((((((....))).(....).))))..))).))).))))).....(((((.((......(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)).))))). ( -61.90, z-score = -4.66, R) >droEre2.scaffold_4845 4509315 167 + 22589142 ----AUUCAUUAUUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----............(((((((((...-...))))((((......))))((((...(((((.......)))).)...))))))))).....(((((.(((.....(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))). ( -61.30, z-score = -4.59, R) >droAna3.scaffold_13340 22989600 167 + 23697760 ----AUUUAUUAAUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCUCCUAGCACUCCGUCAGAUUCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----........(((..((((((((...-...))))((((......))))(((....)))..(((.(((((((((....))))..)).))).))).....))))..(-(((((((((((((((...((((((...)))))).....)))))))))))))))).......))). ( -62.00, z-score = -5.09, R) >dp4.chr4_group4 4126373 167 + 6586962 ----AUUUAUUAUUAAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAA-UCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----............(((((((((...-...))))((((......))))((((...(((((.......)))).)...))))))))).....(((((.(((.....(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))). ( -58.90, z-score = -4.67, R) >droPer1.super_5 2257859 140 - 6813705 -----UUAAAUACUUAGAUGAGCUGAAAAUCCCGGCGGUACUGCAAUAC------------------------AGCUAGUCGCUAGCAUUCCGUCUGAUUCCAAAAAAUCAGUUUAACAAUUGUUGUUCUCCAAUGGAGAAACCAUCAAAUGUUAAGCUAAUAAGAACG---- -----........(((((((.((((.......)))).............------------------------.(((((...)))))....)))))))............(((((((((.(((.((((((((...))))))..)).))).)))))))))..........---- ( -33.70, z-score = -2.79, R) >droWil1.scaffold_180708 409889 167 - 12563649 ----AUUUAUUACUUAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGUUUCCAAAAA-UCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----............(((((((((...-...))))((((......))))((((...(((((.......)))).)...))))))))).....((((((((......(-(((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))). ( -60.30, z-score = -4.82, R) >droVir3.scaffold_12723 3580068 167 + 5802038 ----AUUUAUCGGUUAAUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCAAUUUCCAAAAA-UCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----...((((((..((((((((((...-...))))((...(((....(((((....)))).)....(((.((......))))).)))..)).)))))).))....(-(((((((((((((((...((((((...)))))).....)))))))))))))))).......)))) ( -57.50, z-score = -4.11, R) >droGri2.scaffold_8087 2783 167 + 4350 ----AUUGAUUAUUCAGUUGGGCCGAAA-UCCCGGCGGUACUGCAAUACCGGGCCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAA-UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----..(((....)))(((((((((...-...))))((((......))))...)))))..(((....(((.((......)))))..)))...(((((.(((.....(-(((((((((((((((..((.((((...))))..))...))))))))))))))))..)))))))). ( -55.10, z-score = -3.31, R) >droMoj3.scaffold_6500 17118281 166 - 32352404 ----AUUUAUUUCUUAGUUGGGCCGAA--UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUAUUUCCAAAAA-UCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ----......(((((((((((((((..--...))))((((......))))((((...(((((.......)))).)...)))))))))....................-(((((((((((((((...((((((...)))))).....)))))))))))))))))))))...... ( -56.00, z-score = -3.89, R) >anoGam1.chr3L 4337747 169 + 41284009 UUCGUUUGUAUGAUUAGUUGGGCCGA---UCCCGGCGGUACUGCAAUACCGGGCCAACCCGUGGCGGAUGUGGAGCAAGCCCCGUACGAUCCGCCUCUUUCCAAAAA-UCAGUUUAACAUUUGUCGUCUCCCAAUGGGAGAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUA ...((((((........((((((((.---...))))((((......))))(((....)))(.(((((((((((.(....).))..)).))))))).)...))))..(-(((((((((((((((..(((((((...)))))).)...))))))))))))))))....)))))). ( -64.20, z-score = -3.90, R) >consensus ____AUUUAUUAUUCAGUUGGGCCGAAA_UCCCGGCGGUACUGCAAUACCGGGCCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAA_UCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUA .................((((((((.......))))((...(((.....((((....))))......(((.((......))))).)))..))........))))....(((((((((((((((...((((((...)))))).....)))))))))))))))............ (-48.05 = -48.15 + 0.10)

| Location | 19,815,601 – 19,815,768 |
|---|---|
| Length | 167 |
| Sequences | 13 |
| Columns | 173 |
| Reading direction | reverse |
| Mean pairwise identity | 90.17 |
| Shannon entropy | 0.23326 |
| G+C content | 0.45678 |
| Mean single sequence MFE | -55.44 |
| Consensus MFE | -47.35 |
| Energy contribution | -47.82 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.55 |
| SVM RNA-class probability | 0.992569 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 19815601 167 - 23011544 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAAUAAUAAAU---- ..((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))-)....)))).))))(((.(((.((((((.........)))))).)))(((((((((((((...........)))))).-..)))))))..)))..........---- ( -58.00, z-score = -3.12, R) >droSim1.chr2L 19474822 167 - 22036055 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGAACCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAAUAAUAAAU---- .....(((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))-)....)))))..(((((((.(...((((....)))).))))))))..(((((((((((((...........)))))).-..)))))))...............---- ( -58.80, z-score = -3.21, R) >droSec1.super_7 3421298 167 - 3727775 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAAUAAUAAAU---- ..((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))-)....)))).))))(((.(((.((((((.........)))))).)))(((((((((((((...........)))))).-..)))))))..)))..........---- ( -58.00, z-score = -3.12, R) >droYak2.chr2R 6280986 167 - 21139217 AAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGGAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUUGCGGGUUGCCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAAUAAUAAAU---- ....(((((..(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))-)..(((((......((((((.((.(((((..((((((.......)).)))).)))))(((((....)))))...)).)-))))).)))))..)))))......---- ( -55.80, z-score = -2.20, R) >droEre2.scaffold_4845 4509315 167 - 22589142 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAAUAAUGAAU---- ..((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))-)....)))).))))(((.(((.((((((.........)))))).)))(((((((((((((...........)))))).-..)))))))..)))..........---- ( -57.60, z-score = -2.55, R) >droAna3.scaffold_13340 22989600 167 - 23697760 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGAAUCUGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAUUAAUAAAU---- .....(((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))-)....)))))..(((((((.(...((((....)))).))))))))..(((((((((((((...........)))))).-..)))))))...............---- ( -57.70, z-score = -3.25, R) >dp4.chr4_group4 4126373 167 - 6586962 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGA-UUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUUAAUAAUAAAU---- .....(((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))-)....)))))..(((((((.(...((((....)))).))))))))..(((((((((((((...........)))))).-..)))))))...............---- ( -54.60, z-score = -2.68, R) >droPer1.super_5 2257859 140 + 6813705 ----CGUUCUUAUUAGCUUAACAUUUGAUGGUUUCUCCAUUGGAGAACAACAAUUGUUAAACUGAUUUUUUGGAAUCAGACGGAAUGCUAGCGACUAGCU------------------------GUAUUGCAGUACCGCCGGGAUUUUCAGCUCAUCUAAGUAUUUAA----- ----(((..........((((((.(((.((...(((((...))))).)).))).)))))).(((((((....))))))))))(((((((...((..((((------------------------(.(((.(.(.....).).)))...)))))..))..)))))))..----- ( -34.60, z-score = -1.53, R) >droWil1.scaffold_180708 409889 167 + 12563649 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGA-UUUUUGGAAACAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUAAGUAAUAAAU---- ....((((((((((((.(((((((.((...(((.((((...)))))))..)).))))))).))))-....((....))(((((((.(...((((....)))).))))))))..(((((((((((((...........)))))).-..)))))))...)))).))))...---- ( -54.40, z-score = -2.41, R) >droVir3.scaffold_12723 3580068 167 - 5802038 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGA-UUUUUGGAAAUUGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAAUUAACCGAUAAAU---- ((((..((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))-)....))))...(((((((.(...((((....)))).))))))))..(((((((((((((...........)))))).-..)))))))........))))...---- ( -55.30, z-score = -2.62, R) >droGri2.scaffold_8087 2783 167 - 4350 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGGACAACAAAUGUUAAACUGA-UUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA-UUUCGGCCCAACUGAAUAAUCAAU---- ..((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))-)....)))).))))(((.(((.((((((.........)))))).)))(((((((((((((...........)))))).-..)))))))..)))..........---- ( -55.10, z-score = -2.43, R) >droMoj3.scaffold_6500 17118281 166 + 32352404 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGA-UUUUUGGAAAUAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA--UUCGGCCCAACUAAGAAAUAAAU---- ............((((.(((((((.((...(((.((((...)))))))..)).))))))).))))-(((((((.....(((((((.(...((((....)))).))))))))..((((((.((((((...........)))))).--..))))))..)))))))......---- ( -55.60, z-score = -2.80, R) >anoGam1.chr3L 4337747 169 - 41284009 UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUCUCCCAUUGGGAGACGACAAAUGUUAAACUGA-UUUUUGGAAAGAGGCGGAUCGUACGGGGCUUGCUCCACAUCCGCCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA---UCGGCCCAACUAAUCAUACAAACGAA ..(((.((((.(((((.(((((((.((...(.((((((...)))))))..)).))))))).))))-)....)))))))(((((((.((..((((....)))))))))))))..(((((((((((((...........)))))).---)))))))................... ( -65.20, z-score = -4.23, R) >consensus UAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGA_UUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGA_UUUCGGCCCAACUGAAUAAUAAAU____ ......((((..((((.(((((((.((...(.((((((...)))))))..)).))))))).))))......))))...(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))....)))))))................... (-47.35 = -47.82 + 0.48)

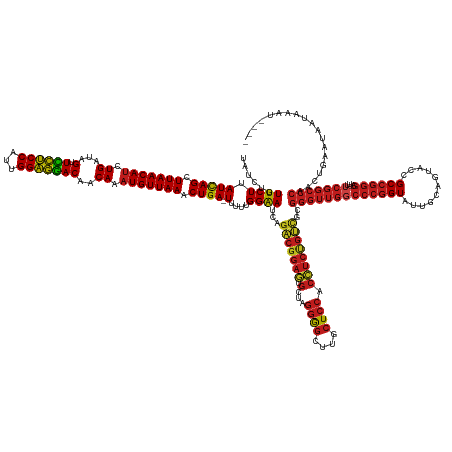
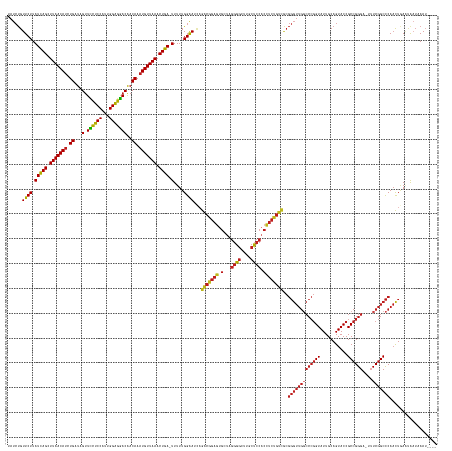
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:53:07 2011