
| Sequence ID | dm3.chr2L |
|---|---|
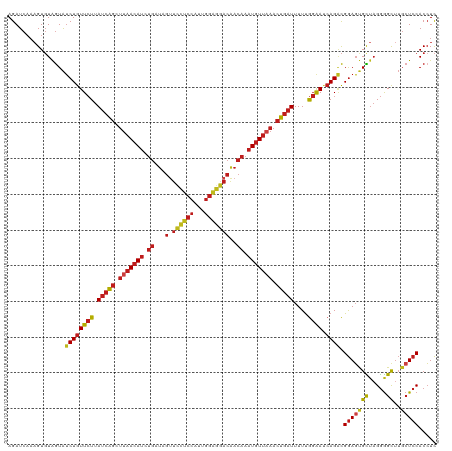
| Location | 19,812,668 – 19,812,787 |
| Length | 119 |
| Max. P | 0.999221 |

| Location | 19,812,668 – 19,812,787 |
|---|---|
| Length | 119 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.95 |
| Shannon entropy | 0.24937 |
| G+C content | 0.40640 |
| Mean single sequence MFE | -32.68 |
| Consensus MFE | -27.99 |
| Energy contribution | -28.54 |
| Covariance contribution | 0.55 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.855905 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
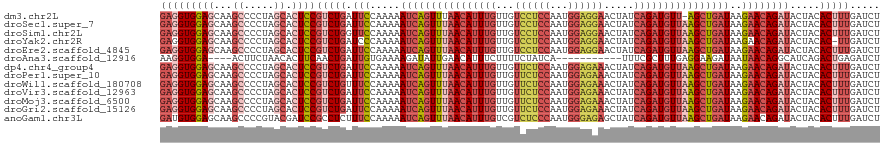
>dm3.chr2L 19812668 119 + 23011544 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCU-AACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(-(((((.((...((.(((((...)))))))..)).))))))..)))))....)))).)))).(((((((.....))..)))))..... ( -32.50, z-score = -1.08, R) >droSec1.super_7 3418139 120 + 3727775 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -35.40, z-score = -1.92, R) >droSim1.chr2L 19471789 120 + 22036055 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAACCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -35.40, z-score = -1.81, R) >droYak2.chr2R 6275398 119 + 21139217 AGAUCAA-GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGGAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC .......-........((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -34.90, z-score = -1.41, R) >droEre2.scaffold_4845 18079646 120 - 22589142 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -35.40, z-score = -1.92, R) >droAna3.scaffold_12916 7183671 105 - 16180835 AGAUCUCAGUCUGAUGCCUGUUAUUAUCUUCCUCAAAGCGAAA-----------UGAUAGAAAAGAAAUGUUCAAUAUCUUUUCACAAUCAGUUGAAGUGUUAGAAGU----UCCACCUU .....((((.(((((......((((((.((((.....).))))-----------)))))(((((((.((.....)).)))))))...)))))))))............----........ ( -16.80, z-score = 0.08, R) >dp4.chr4_group4 2583281 120 - 6586962 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -33.00, z-score = -1.67, R) >droPer1.super_10 406312 120 - 3432795 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -33.00, z-score = -1.67, R) >droWil1.scaffold_180708 407193 120 - 12563649 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAACAGACGGAGUGCUAGGGGCUUGCUCCACCUC ...........(((((((((((...(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))...((....)))))))).)))))((((....))))..... ( -34.20, z-score = -2.07, R) >droVir3.scaffold_12963 18754608 120 - 20206255 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -33.00, z-score = -1.67, R) >droMoj3.scaffold_6500 4054925 120 - 32352404 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -33.00, z-score = -1.67, R) >droGri2.scaffold_15126 7053743 120 + 8399593 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))).(((((((.....))..)))))..... ( -33.00, z-score = -1.67, R) >anoGam1.chr3L 4334947 120 + 41284009 AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUCUCCCAUUGGGAGACGACAAAUGUUAAACUGAUUUUUGGAAAGAGGCGGAUCGUACGGGGCUUGCUCCACAUC .((((........((.(((.((((.(((((.(((((((.((...(.((((((...)))))))..)).))))))).)))))....))))))).)).))))....((((....))))..... ( -35.30, z-score = -1.89, R) >consensus AGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUC ................((((((((.(((((.(((((((.((.....((((((...))))))...)).))))))).)))))....)))).)))).(((((((.....))..)))))..... (-27.99 = -28.54 + 0.55)


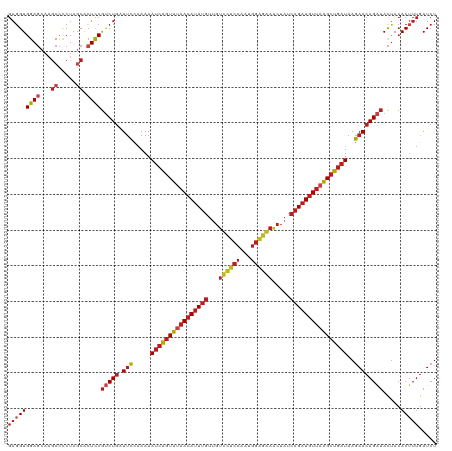
| Location | 19,812,668 – 19,812,787 |
|---|---|
| Length | 119 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.95 |
| Shannon entropy | 0.24937 |
| G+C content | 0.40640 |
| Mean single sequence MFE | -38.72 |
| Consensus MFE | -33.68 |
| Energy contribution | -34.88 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.17 |
| Mean z-score | -4.25 |
| Structure conservation index | 0.87 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.72 |
| SVM RNA-class probability | 0.999221 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
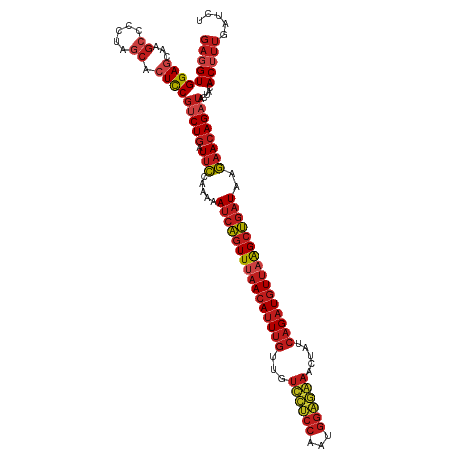
>dm3.chr2L 19812668 119 - 23011544 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUU-AGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....(((((((.((((((((...((((((...)))))).....))))))))-)))))))..)))))))).....)))))..... ( -37.90, z-score = -3.58, R) >droSec1.super_7 3418139 120 - 3727775 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -42.60, z-score = -5.05, R) >droSim1.chr2L 19471789 120 - 22036055 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGGUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -42.60, z-score = -4.53, R) >droYak2.chr2R 6275398 119 - 21139217 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUCCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC-UUGAUCU .((((((((...((.....)).))))(((((.((......((((((((((((((((...((((((...)))))).....))))))))))))))))..)).))))).....))-))..... ( -40.70, z-score = -4.52, R) >droEre2.scaffold_4845 18079646 120 + 22589142 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -42.60, z-score = -5.05, R) >droAna3.scaffold_12916 7183671 105 + 16180835 AAGGUGGA----ACUUCUAACACUUCAACUGAUUGUGAAAAGAUAUUGAACAUUUCUUUUCUAUCA-----------UUUCGCUUUGAGGAAGAUAAUAACAGGCAUCAGACUGAGAUCU .(((((..----........)))))...(((((.(((((((((...........)))))))((((.-----------((((.......)))))))).......))))))).......... ( -17.70, z-score = 0.64, R) >dp4.chr4_group4 2583281 120 + 6586962 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -40.20, z-score = -4.85, R) >droPer1.super_10 406312 120 + 3432795 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -40.20, z-score = -4.85, R) >droWil1.scaffold_180708 407193 120 + 12563649 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGUUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))((((((((......((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -41.60, z-score = -5.19, R) >droVir3.scaffold_12963 18754608 120 + 20206255 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -40.20, z-score = -4.85, R) >droMoj3.scaffold_6500 4054925 120 + 32352404 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -40.20, z-score = -4.85, R) >droGri2.scaffold_15126 7053743 120 - 8399593 GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... ( -40.20, z-score = -4.85, R) >anoGam1.chr3L 4334947 120 - 41284009 GAUGUGGAGCAAGCCCCGUACGAUCCGCCUCUUUCCAAAAAUCAGUUUAACAUUUGUCGUCUCCCAAUGGGAGAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU ((.(((((....((...))....))))).)).........((((((((((((((((..(((((((...)))))).)...)))))))))))))))).(((.((((........)))).))) ( -36.60, z-score = -3.73, R) >consensus GAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCU (((((((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).....)))))..... (-33.68 = -34.88 + 1.20)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:53:04 2011