| Sequence ID | dm3.chr2L |
|---|---|
| Location | 19,810,730 – 19,810,905 |
| Length | 175 |
| Max. P | 0.947258 |

| Location | 19,810,730 – 19,810,905 |
|---|---|
| Length | 175 |
| Sequences | 13 |
| Columns | 194 |
| Reading direction | forward |
| Mean pairwise identity | 86.37 |
| Shannon entropy | 0.29797 |
| G+C content | 0.42778 |
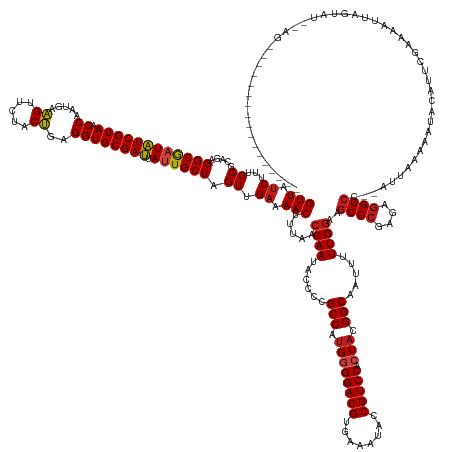
| Mean single sequence MFE | -47.48 |
| Consensus MFE | -40.40 |
| Energy contribution | -41.05 |
| Covariance contribution | 0.66 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.53 |
| SVM RNA-class probability | 0.947258 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
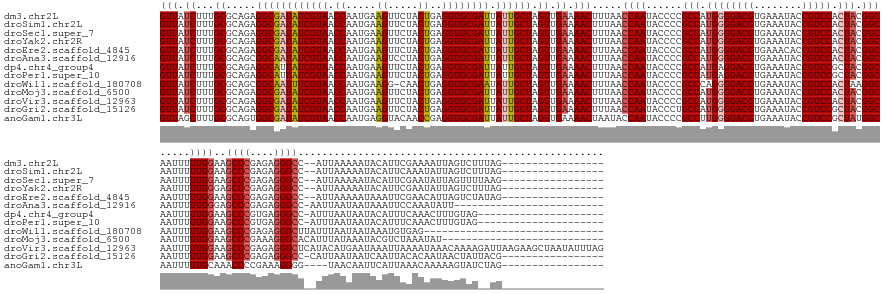
>dm3.chr2L 19810730 175 + 23011544 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGAAGCCCGAGAGGGCC--AUUAAAAAUACAUUCGAAAAUUAGUCUUUAG----------------- (((((.....(((....))).....)))))...(((((..(((.((((((.((((((....))))))...))))))...........(((.((((((((........))))).))).))).((((((((..((((....)))).--.))))))))............))).))))).----------------- ( -45.80, z-score = -1.25, R) >droSim1.chr2L 19469875 175 + 22036055 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGAAGCCCGAGAGGGCC--AUUAAAAAUACAUUCAAAUAUUAGUCUUUAG----------------- .............(((((((((((((((.((.....((.....))..))))))).)))))))..(((((..................(((.((((((((........))))).))).))).((((((((..((((....)))).--.))))))))...))))).......)))....----------------- ( -45.90, z-score = -1.42, R) >droSec1.super_7 3416151 175 + 3727775 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGAAGCCCGAGAGGGCC--AUUAAAAAUACAUUCGAAUAUUAGUUUUAAG----------------- ............(((.((((((((((((.((.....((.....))..))))))).)))))))..)))((((((..............(((.((((((((........))))).))).))).((((((((..((((....)))).--.)))))))).............))))))...----------------- ( -46.00, z-score = -1.43, R) >droYak2.chr2R 6273401 175 + 21139217 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGGAGCCCGAGAGGGCC--AUUAAAAAUACAUUCGAAUAUUAGUCUUUAG----------------- (((((.....(((....))).....)))))...(((((..(((.((((((.((((((....))))))...))))))...........(((.((((((((........))))).))).))).((((((((..((((....)))).--.))))))))............))).))))).----------------- ( -47.00, z-score = -1.25, R) >droEre2.scaffold_4845 18075118 175 - 22589142 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAACACCGUCCACUACGGCAAUUUUUGGAAGCCCGAGAGGGCC--AUUAAAAAUAAAUUCGAACAUUAGUCUAUAG----------------- (((((.....(((....))).....))))).......((((...((((((.((((((....))))))...))))))...........(((.((((((((........))))).))).))).((((((((..((((....)))).--.))))))))......))))............----------------- ( -46.40, z-score = -1.43, R) >droAna3.scaffold_12916 7182070 168 - 16180835 GUUAUCUUUGCGCAGCGGCAAUAUCGUAACCAAUGAAGUCCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGGAGCCCGAGAGGGCC-AAUUAAUAAUAAAUUCCAAAUAUU------------------------- ((((........((((((((((((((((.((.....((.....))..))))))).))))))).)))).......)))).........(((.((((((((........))))).))).)))....(((((((((((....)))).-..(((....))).)))))))....------------------------- ( -48.26, z-score = -1.99, R) >dp4.chr4_group4 2580534 172 - 6586962 GUUAUCUUUGCGCAGAGGCAUUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGAGGACGUGAAAUACCGUCCGCUACGGCAAUUUUUGGAAGCCCGUGAGGGCC-AUUUAAUAAUACAUUUCAAACUUUGUAG--------------------- ...........(((((((((..((((((.((.....((.....))..))))))))...)))...((((((.......((((......(((.((.(((((........)))))..)).))).....))))..((((....)))).-.............)))))).))))))..--------------------- ( -46.90, z-score = -2.02, R) >droPer1.super_10 403889 172 - 3432795 GUUAUCUUUGCGCAGAGGCAUUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGAGGACGUGAAAUACCGUCCGCUACGGCAAUUUUUGGAAGCCCGUGAGGGCC-AUUUAAUAAUACAUUUCAAACUUUGUAG--------------------- ...........(((((((((..((((((.((.....((.....))..))))))))...)))...((((((.......((((......(((.((.(((((........)))))..)).))).....))))..((((....)))).-.............)))))).))))))..--------------------- ( -46.90, z-score = -2.02, R) >droWil1.scaffold_180708 8629342 163 - 12563649 GUUAUCUUUGCGCAGCGGCAAUUUCGUAACCAAUGAAGG-CAACUGAGGUGCGAAUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCCAGGGGACGUGAAAUACCGUCCACUAAGGCAAUUUUUGGAAGCCCGAGAGGGCUUAUUUAAUAAUAAAUGUGAG------------------------------ ..........((((..((((((((((((.((.....((.-...))..)))))))).)))))).((((((........((((......(((..(((((((........))))).))..))).....))))((((((....)))))).))))))......))))..------------------------------ ( -50.60, z-score = -2.64, R) >droMoj3.scaffold_6500 4015815 167 - 32352404 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGAAGCCCGAAAGGGCACAUUUAUAAAUACGUCUAAAUAU--------------------------- (((.((...((.....((((((((((((.((.....((.....))..))))))).))))))).)).)).))).....((((......(((.((((((((........))))).))).))).....))))..((((....))))........................--------------------------- ( -45.10, z-score = -1.66, R) >droVir3.scaffold_12963 18752679 194 - 20206255 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGAAGCCCGAGAGGGCUCAUACAUGAAUAAAUUAAAAUAAACAAAAGAUUAAGAAGCUAAUAUUUAG ((((.((((...(((.((((((((((((.((.....((.....))..))))))).)))))))..)))....((((..((((......(((.((((((((........))))).))).))).....)))).(((((....)))))..........................)))).....)))).))))...... ( -47.50, z-score = -1.59, R) >droGri2.scaffold_15126 7051060 176 + 8399593 GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCUGCCAUGGGGACGUGAAAUACCGUCCGCUACGGCAAUUUUUGGAAGCCCGAGAGGGCC-CAUUAAUAAUCAAUUACACAAUAACUAUUACG----------------- (((((((((....))))(((((((((((.((.....((.....))..))))))).))))))(((((((.........((((.....((((.((((((((........))))).))).))))....))))..((((....)))).-.........)))))))....))))).......----------------- ( -49.40, z-score = -2.36, R) >anoGam1.chr3L 36084616 173 - 41284009 GUUAGCUUUGCGCAGUGGCGAUAUCGUAACCAAUGAGGUACAACCGAGGUGCGAUUAUUGCUAGUUGAAAACUAAUACCAAUACCCCGCCUUGGGGACGUGAAAUACCGUCCGCUAUGGCAAUUUUUGCAAACCCCGAAAGGGG----UAACAAUUCAUUAAACAAAAAGUAUCUAG----------------- ....(((((..((.(((((......(((.((.....)))))..((((((((.(..(((((.((((.....))))....)))))..)))))))))(((((........)))))))))).))...........(((((....))))----).................)))))......----------------- ( -51.50, z-score = -2.44, R) >consensus GUUAUCUUUGCGCAGAGGCGAUAUCGUAACCAAUGAAGUUCUACUGAGGUGCGAUUAUUGCUAGUUGAAAACUUUAACCAAUACCCCGCCAUGGGGACGUGAAAUACCGUCCACUACGGCAAUUUUUGGAAGCCCGAGAGGGCC__AUUAAAAAUACAUUCGAAAAUUAGUAU__AG_________________ (((.((...((.....((((((((((((.((.....((.....))..)))))))).)))))).)).)).))).....((((......(((.((((((((........))))).))).))).....))))..((((....))))................................................... (-40.40 = -41.05 + 0.66)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:53:01 2011