
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 19,717,048 – 19,717,235 |
| Length | 187 |
| Max. P | 0.869022 |

| Location | 19,717,048 – 19,717,235 |
|---|---|
| Length | 187 |
| Sequences | 15 |
| Columns | 188 |
| Reading direction | forward |
| Mean pairwise identity | 75.96 |
| Shannon entropy | 0.56867 |
| G+C content | 0.51857 |
| Mean single sequence MFE | -52.12 |
| Consensus MFE | -27.22 |
| Energy contribution | -27.81 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.52 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.99 |
| SVM RNA-class probability | 0.869022 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 19717048 187 + 23011544 GUCUACAAA-GGUGUGCCAUCGCAACCGAGCAAUUUCCGGGCAACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGACCAAGCCGACGCAUUCCAGCGAGAAUAUCGUGCACUACGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGCCCAUCACAAGUGAGUCCUUAGUCUCGGAGUUAACC (((((....-(((.((((...((......))......(((....))).....))))..)))((((.....))))..)))))....((((.(((((((((.(((....(((((...))))).)))..)))))))(((......(((.....)))...(((....))))))....)).))))........ ( -57.90, z-score = -1.72, R) >anoGam1.chr3R 3729639 179 - 53272125 ---------AGGCGUUCCCUCACAACCAAGCAACUUUCGGGCUACCGAUGUCGGUGAAACAGCUGUCACCCUGCAAUGGUCCCGGCCAACUCACUCGGGCGAGAACAUCGUACACUACGAGCUGUACUGGAAUGACACUUAUGCAAACGAGCAGCAUCAUCAGUAUGUAUAUGUUUCCGCAACCACCC ---------.(((((......))..............((((..((((.(((.(((((........)))))..))).))))))))))).........(((((..(((((((((...)))))((.((((((..((((......(((......)))...))))))))))))...))))..)))..)).... ( -45.60, z-score = 0.85, R) >droSim1.chr2L 19375980 187 + 22036055 GUCUACAAA-GGUGUGCCAUCGCAACCGAGCAAUUUCCGGGCAACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGACCAAGCCGACGCAUUCCAGCGAGAAUAUCGUGCACUACGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGCCCAUCACAAGUGAGUCCUUAGCCCCGGAGUUUACC (((((....-(((.((((...((......))......(((....))).....))))..)))((((.....))))..)))))....(((..(((((((((.(((....(((((...))))).)))..)))))))(((......(((.....)))...(((....))))))....))..)))........ ( -56.20, z-score = -1.32, R) >droSec1.super_7 3327435 187 + 3727775 GUCUACAAA-GGUGUGCCAUCGCAACCGAGCAAUUUCCGGGCAACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGACCAAGCCGACGCAUUCCAGCGAGAAUAUCGUGCACUACGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGCCCAUCACAAGUGAGUCCUUAGCCCCGGAGUUUACC (((((....-(((.((((...((......))......(((....))).....))))..)))((((.....))))..)))))....(((..(((((((((.(((....(((((...))))).)))..)))))))(((......(((.....)))...(((....))))))....))..)))........ ( -56.20, z-score = -1.32, R) >droYak2.chr2R 6181090 168 + 21139217 GUCUACAAA-GGUGUGCCAUCGCAACCGAGCAAUUUCCGGGCAACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGACCAAGCCGACGCAUUCCAGCGAGAACAUCGUGCACUACGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGCCCAUCACAAGUGAGUC------------------- (((((....-(((.((((...((......))......(((....))).....))))..)))((((.....))))..)))))......((((((((((((.(((....(((((...))))).)))..)))))))).)......(((.....)))...(((....))))))------------------- ( -52.70, z-score = -1.74, R) >droEre2.scaffold_4845 17984436 168 - 22589142 AUCUACAAA-GGUGUGCCAUCGCAACCGAGCAAUUUCCGGGCAACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGACCAAGCCGACGCAUUCCAGCGAGAAUAUCGUGCACUACGAGCUCUACUGGAAUGACACAUACGGCAACACGGUCCAUCACAAGUGAGUU------------------- ...(((...-(((.((((...((......))......(((....))).....))))..)))((((.....)))).(((((((..((((...((((((((.(((....(((((...))))).)))..)))))))).......)))).....))))))).....)))....------------------- ( -57.20, z-score = -2.96, R) >droAna3.scaffold_12916 7090979 174 - 16180835 GUCUACAAAAGGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACCGAUAUCGGCGAGACCGCAGUCACAUUGCAAUGGUCGAAGCCGACGCAUUCCAGCGAGAAUAUCGUACACUACGAGCUCUACUGGUAUGACACAUACGCCAAUCAGACCCAUCACAAGUGAGUUCUAGA-------------- ...(((....((((.(((.((......))..........)))))))(((((((((..(((((((((...)))))...))))...)))))(((......)))....)))))))..(((.((((((.(((.(((((...)))))........((....))...)))))))))))).-------------- ( -48.20, z-score = -1.13, R) >dp4.chr4_group5 1804477 172 + 2436548 GCCUACAAAAGGUGUGCCAUCGCAACCGAGCAAUUUCCGGGCCACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGUCGAAGCCUACGCAUUCCAGCGAGAACAUUGUUCACUACGAGCUCUACUGGAAUGACACAUAUGCCAAUCAGGCACAUCACAAGUGAGUAUUU---------------- ((.(((....((((((((.(((....)))(((.....(((....))).....(((..((((((((.....))))...))))...)))...(((((((((.(((....((((.....)))).)))..)))))))).).....)))......))))))))....))).))....---------------- ( -54.90, z-score = -2.61, R) >droPer1.super_5 6210120 172 + 6813705 GCCUACAAAAGGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGUCGAAGCCUACGCAUUCCAGCGAGAACAUUGUACACUACGAGCUCUACUGGAAUGACACAUAUGCCAAUCAGGCACAUCACAAGUGAGUAUUU---------------- ((.(((....((((((((.((......))(((.....(((....))).....(((..((((((((.....))))...))))...)))...(((((((((.(((....(((((...))))).)))..)))))))).).....)))......))))))))....))).))....---------------- ( -53.40, z-score = -2.71, R) >droWil1.scaffold_180708 8462170 178 - 12563649 -----CACA-GGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACUGAUAUCGGCGAGACCGCAGUCACAUUGCAAUGGUCGAAGCCCACGCAUUCCAGUGAGAAUAUCGUUCACUAUGAACUCUAUUGGAAUGACACAUAUGCCAAUCAGGCACAUCAUAAGUAAGUAUUGACCGAAAGUAAU---- -----....-((((((((.((......))(((......(((((.(((....))).).(((((((((...)))))...))))...))))..(((((((((((((....((((.....)))).))).))))))))).).....)))......))))))))................(....)....---- ( -50.10, z-score = -1.62, R) >droVir3.scaffold_12963 18651145 182 - 20206255 -----CCCA-GGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACUGAUAUCGGCGAGACCGCAGUCACAUUGCAAUGGUCCAAGCCGACGCAUUCCAGCGAGAAUAUUGUACAUUAUGAGCUCUACUGGAAUGACACAUAUGCCAAUCAGGAUCAUCACAAGUGAGUUGGAGCCCCCUUCCCCUCCC -----....-((((((....))).)))(((........((((..(((((.(((((..(((((((((...)))))...))))...))))).(((((((((.(((.(((........)))...)))..)))))))).)..........)))))..((.(((....)))....)))))).......))).. ( -51.36, z-score = -0.64, R) >droMoj3.scaffold_6500 3895926 167 - 32352404 CUGUGUGCA-GGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACUGAUAUCGGCGAGACCGCAGUCACAUUGCAGUGGUCCAAGCCGACUCAUUCCAGCGAGAACAUUGUGCACUACGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGAUCAUCACAAGUGAGU-------------------- .(((((((.-((((((....))).)))..)))...(((.(((........(((((..((((((.((......)).))))))...))))).(((((((((.(((....(((((...))))).)))..))))))))).......))).....)))....)))).......-------------------- ( -53.10, z-score = -0.74, R) >droGri2.scaffold_15126 6948331 168 + 8399593 GUUUGUACA-GGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACUGAUAUCGGCGAGACCGCAGUCACAUUGCAGUGGUCCAAGCCGACGCAUUCCAGCGAGAAUAUUGUACAUUAUGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGAUCAUCACAAGUGAGUU------------------- ..((((...-((((((....))).)))..))))..(((.(((........(((((..((((((.((......)).))))))...))))).(((((((((.(((.(((........)))...)))..)))))))).)......))).....)))((((.....))))...------------------- ( -48.10, z-score = -0.68, R) >apiMel3.Group14 7928388 166 - 8318479 ----------------------CAACCGGAGAUGUUGACCGCGAUAGAUAUCGGGGAGACCACGGUAACCCUCCAGUGGAAUAAACCUAUUCAUAGCGCGGAAAAUAUUCUCAGCUAUGAAUUGUAUUGGAACGAUACGUACGCUCAGGUGGGUGAACACGUCUUACUUCCCUCCUCUUCUUCUUUUU ----------------------.....(((((((((..........))))))((((((...(((((.((((.((((((..........(((((((((..((((....))))..))))))))).((((((...))))))...))))..)).))))..)).)))....)))))))))............. ( -47.70, z-score = -1.23, R) >triCas2.ChLG3 1927338 175 + 32080666 GUCUGUUUA-GGCGUCCCAAGCCAACCGAGCAACCUGAUGGCGUCGGAUAUCGGCGAGACCUCCGUCACCCUACAGUGGAGCAAACCGACGCAUUCCGGGGAAAACAUCGUCAAUUACGAGCUCUACUGGAAUGACACGUACGCCAAAGAGAAACACCACCGUCGCAUACCUAUCA------------ .(((.(((.-((((((.((..(((...((((..((((...(((((((.....((((.(....)))))..((......))......)))))))....)))).......((((.....))))))))...)))..))....).)))))))).)))........................------------ ( -49.20, z-score = -0.25, R) >consensus GUCUACAAA_GGUGUGCCAUCACAACCGAGCAAUUUCCGGGCCACCGAUAUCGGCGAGACCGCAGUCACACUGCAAUGGUCCAAGCCGACGCAUUCCAGCGAGAAUAUCGUGCACUACGAGCUCUACUGGAAUGACACAUACGCCAAUCAGGCCCAUCACAAGUGAGUACUU________________ ..........(((((.((((.....((((........(((....)))...)))).......((((.....)))).))))............((((((((.(((....((((.....)))).)))..))))))))......)))))........................................... (-27.22 = -27.81 + 0.60)

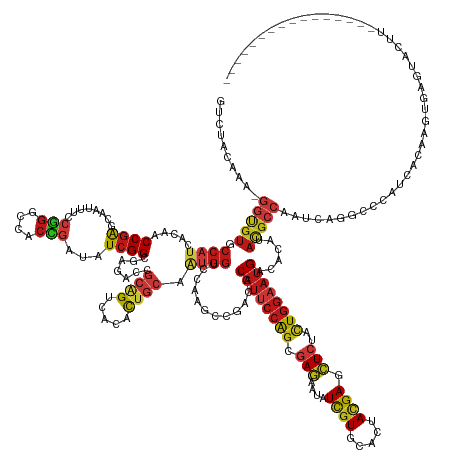
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:52:50 2011