
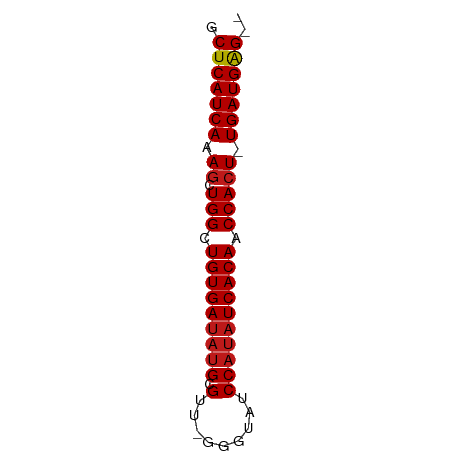
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 19,569,495 – 19,569,553 |
| Length | 58 |
| Max. P | 0.999091 |

| Location | 19,569,495 – 19,569,553 |
|---|---|
| Length | 58 |
| Sequences | 14 |
| Columns | 62 |
| Reading direction | forward |
| Mean pairwise identity | 93.58 |
| Shannon entropy | 0.14994 |
| G+C content | 0.47057 |
| Mean single sequence MFE | -23.99 |
| Consensus MFE | -23.77 |
| Energy contribution | -24.28 |
| Covariance contribution | 0.51 |
| Combinations/Pair | 1.04 |
| Mean z-score | -4.47 |
| Structure conservation index | 0.99 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.64 |
| SVM RNA-class probability | 0.999091 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
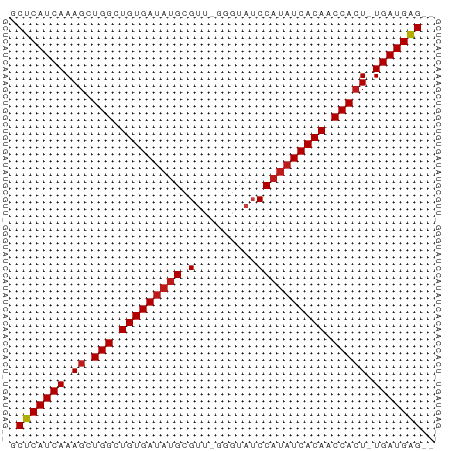
>dm3.chr2L 19569495 58 + 23011544 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droGri2.scaffold_15126 7877571 58 - 8399593 GCUCAUCAAAGCUGGCUGUGAUAUGCGUC-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.(..-......)))))))))).)))))-)))))))-- ( -24.10, z-score = -4.54, R) >droMoj3.scaffold_6500 25443428 58 - 32352404 GCUCAUCAAAGCUGGCUGUGAUAUGCGUC-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.(..-......)))))))))).)))))-)))))))-- ( -24.10, z-score = -4.54, R) >droVir3.scaffold_12723 2960521 58 + 5802038 GCUCAUCAAAGCUGGCUGUGAUAUGCGUC-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.(..-......)))))))))).)))))-)))))))-- ( -24.10, z-score = -4.54, R) >droWil1.scaffold_180708 8278524 58 - 12563649 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droPer1.super_5 1253060 58 - 6813705 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >dp4.chr4_group5 1283396 58 - 2436548 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droAna3.scaffold_12916 4166784 58 - 16180835 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droEre2.scaffold_4845 17868874 58 - 22589142 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droYak2.chr2R 6066569 58 + 21139217 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droSec1.super_7 3183858 58 + 3727775 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >droSim1.chr2L 19257547 58 + 22036055 GCUCAUCAAAGCUGGCUGUGAUAUGCGUU-GGGUAUCCAUAUCACAACCACU-UGAUGAG-- .((((((((.(.(((.(((((((((.((.-....)).))))))))).)))))-)))))))-- ( -24.20, z-score = -4.50, R) >apiMel3.Group1 134966 60 + 25854376 GCUCAUCAAAGCUGGCUGUGAUACGAGUCCAAGUUACCACAUCACAACCACUCUGAUGGG-- .(((((((.((.(((.((((((((........))......)))))).))))).)))))))-- ( -17.90, z-score = -2.39, R) >triCas2.ChLG3 31519414 60 - 32080666 GCUCAUCAAAGCUGGCUGUGAUAUGAGCUACAAAU-CCAUUUCACAUCCAGU-UGAUGAGCG ((((((((..(((((.(((((.(((..........-.))).))))).)))))-)))))))). ( -27.80, z-score = -6.08, R) >consensus GCUCAUCAAAGCUGGCUGUGAUAUGCGUU_GGGUAUCCAUAUCACAACCACU_UGAUGAG__ .(((((((.((.(((.(((((((((.((......)).))))))))).))))).))))))).. (-23.77 = -24.28 + 0.51)

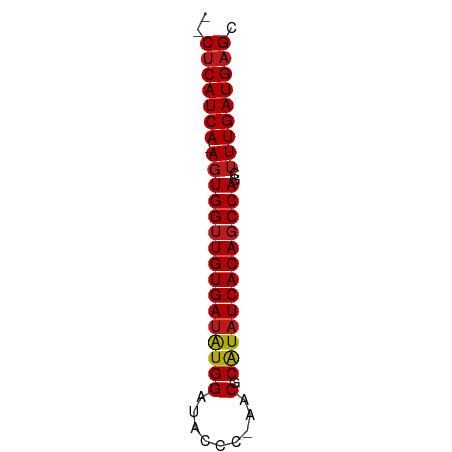
| Location | 19,569,495 – 19,569,553 |
|---|---|
| Length | 58 |
| Sequences | 14 |
| Columns | 62 |
| Reading direction | reverse |
| Mean pairwise identity | 93.58 |
| Shannon entropy | 0.14994 |
| G+C content | 0.47057 |
| Mean single sequence MFE | -27.99 |
| Consensus MFE | -27.57 |
| Energy contribution | -27.72 |
| Covariance contribution | 0.15 |
| Combinations/Pair | 1.09 |
| Mean z-score | -6.02 |
| Structure conservation index | 0.98 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.11 |
| SVM RNA-class probability | 0.997456 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
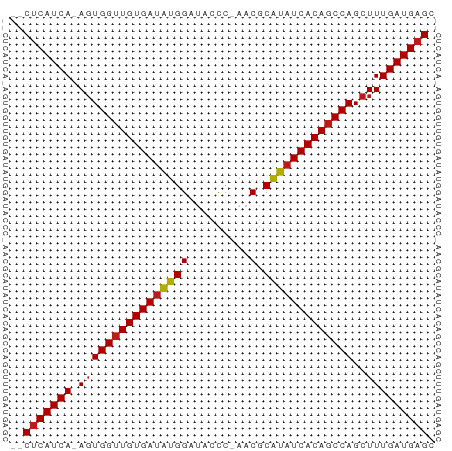
>dm3.chr2L 19569495 58 - 23011544 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droGri2.scaffold_15126 7877571 58 + 8399593 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-GACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.09, R) >droMoj3.scaffold_6500 25443428 58 + 32352404 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-GACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.09, R) >droVir3.scaffold_12723 2960521 58 - 5802038 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-GACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.09, R) >droWil1.scaffold_180708 8278524 58 + 12563649 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droPer1.super_5 1253060 58 + 6813705 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >dp4.chr4_group5 1283396 58 + 2436548 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droAna3.scaffold_12916 4166784 58 + 16180835 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droEre2.scaffold_4845 17868874 58 + 22589142 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droYak2.chr2R 6066569 58 - 21139217 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droSec1.super_7 3183858 58 - 3727775 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >droSim1.chr2L 19257547 58 - 22036055 --CUCAUCA-AGUGGUUGUGAUAUGGAUACCC-AACGCAUAUCACAGCCAGCUUUGAUGAGC --(((((((-((((((((((((((((......-..).)))))))))))))..))))))))). ( -28.20, z-score = -6.29, R) >apiMel3.Group1 134966 60 - 25854376 --CCCAUCAGAGUGGUUGUGAUGUGGUAACUUGGACUCGUAUCACAGCCAGCUUUGAUGAGC --..(((((((((((((((((((((((.......)).))))))))))))).))))))))... ( -27.40, z-score = -4.70, R) >triCas2.ChLG3 31519414 60 + 32080666 CGCUCAUCA-ACUGGAUGUGAAAUGG-AUUUGUAGCUCAUAUCACAGCCAGCUUUGAUGAGC .((((((((-(((((.(((((.(((.-.(.....)..))).))))).))))..))))))))) ( -26.10, z-score = -4.78, R) >consensus __CUCAUCA_AGUGGUUGUGAUAUGGAUACCC_AACGCAUAUCACAGCCAGCUUUGAUGAGC ..(((((((.(((((((((((((((............))))))))))))).)).))))))). (-27.57 = -27.72 + 0.15)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:52:30 2011