
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 18,133,376 – 18,133,494 |
| Length | 118 |
| Max. P | 0.951824 |

| Location | 18,133,376 – 18,133,481 |
|---|---|
| Length | 105 |
| Sequences | 10 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 72.16 |
| Shannon entropy | 0.59807 |
| G+C content | 0.66427 |
| Mean single sequence MFE | -52.65 |
| Consensus MFE | -20.45 |
| Energy contribution | -19.68 |
| Covariance contribution | -0.77 |
| Combinations/Pair | 1.57 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.951824 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
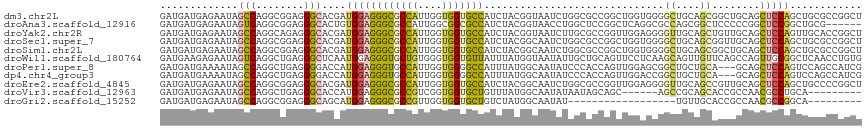
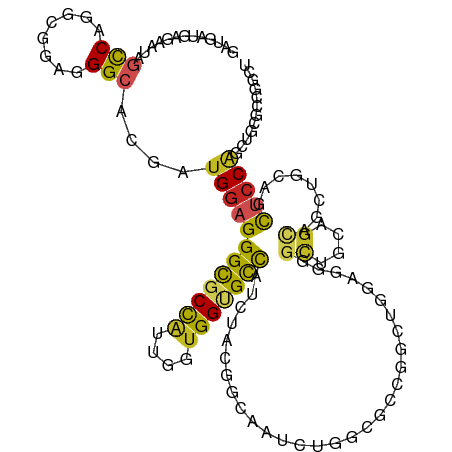
>dm3.chr2L 18133376 105 - 23011544 GCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGCUGCAGCUCCAGCUGCGCCGGCUC (((((((.....)).((.((((.(((((((....))))))).)))))).....)))))((((((.(.(((((((((((...))))))))))).)...)))))).. ( -63.30, z-score = -3.28, R) >droAna3.scaffold_12916 13397294 98 + 16180835 GUCAGGCGGAGGGCACUGUGGAGGGCGCCAUUGGCGGCGCCAUCUACGGUAACCUGGCUCCGGCUCAGGCGCCAGCGGCUCCCCCGGCUCCGGCUGCG------- (.(((.((((((((((((((((.((((((......)))))).))))))))..((((((....)).)))).)))..(((.....))).))))).)))).------- ( -55.00, z-score = -1.67, R) >droYak2.chr2R 4637979 105 - 21139217 GCCAGGCAGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUUGCGCCGGUUGGAGGGGUUGCAGCUGUUGCAGCUCCAGUUGCACCGGCUC .....(((((..((.((.((((.(((((((....))))))).))))))))..)).)))((((((....((((((((((...))))))))))......)))))).. ( -49.50, z-score = -1.86, R) >droSec1.super_7 1759181 105 - 3727775 GCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGUUGCAGCUCCAGCUGCGCCGGCUC ((((((......((.((.((((.(((((((....))))))).))))))))..))))))((((((.(.(((((((((((...))))))))))).)...)))))).. ( -62.10, z-score = -3.04, R) >droSim1.chr2L 17809598 105 - 22036055 GCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGCUGCAGCUCCAGCUGCGCCGGCUC ((((((......((.((.((((.(((((((....))))))).))))))))..))))))((((((.(.(((((((((((...))))))))))).)...)))))).. ( -64.10, z-score = -3.20, R) >droWil1.scaffold_180764 3733477 105 + 3949147 GUCAGGCUGAGGGCUCAAUGGAGGGUGCUGUGGGUGGUGUUAUUUAUGGUAAUAUUGCUGCAGUUCCUCAAGCAGUUGUUCAGCCAGUUGCGGCUCAACCUGUGG ..((((.((((.........(((((.((((((.(..(((((((.....)))))))..))))))))))))..(((..((......))..)))..)))).))))... ( -37.00, z-score = -1.07, R) >droPer1.super_8 2503540 102 + 3966273 GCCAGGCUGAGGGGACCAUGGAGGGUGCCAUUGGUGGGGCCAUUUAUGGCAAUAUCCCACCAGU---UGGAGCGGCUGCUGCAGCAGCUCCAGUCCAGCCAUCGG ....(((((..((.(((......))).))(((((((((((((....)))).....)))))))))---((((((.((((...)))).))))))...)))))..... ( -49.70, z-score = -1.98, R) >dp4.chr4_group3 1439371 102 + 11692001 GCCAGGCUGAGGGGACCAUGGAGGGUGCCAUUGGUGGGGCCAUUUAUGGCAAUAUCCCACCAGU---UGGACCGGCUGCUGCAGCAGCUCCAGUCCAGCCAUCGG ....((((....((((..((((.(((.(((((((((((((((....)))).....)))))))).---)))))).(((((....)))))))))))))))))..... ( -48.30, z-score = -1.60, R) >droEre2.scaffold_4845 16452910 105 + 22589142 GCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGUUGGAGGGGUUGCAGCCGUUGCAGCUCCAGCUGCCCCGGCUC ((((((......((.((.((((.(((((((....))))))).))))))))..))))))(((((..((.((((((((((...)))))))))).....))))))).. ( -52.80, z-score = -0.89, R) >droVir3.scaffold_12963 17225710 90 - 20206255 GCCAGGCUGAGGGCACCAUGGAGGGCGCCGUCGGUGGUGCUGUUUAUGGCAAUAUAAUAGCAGC---AGCCGCAGCACCGCCAACGCCUGCAC------------ ((.((((.((.(((.((......)).))).))(((((((((((....(((..............---.))))))))))))))...))))))..------------ ( -44.66, z-score = -2.62, R) >consensus GCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCUGCUGCAGCUCCAGCUGCGCCGGCUC (((........)))....((((.(((((((....))))))).))))(((..........)))......(((((.(.........).))))).............. (-20.45 = -19.68 + -0.77)



| Location | 18,133,377 – 18,133,494 |
|---|---|
| Length | 117 |
| Sequences | 11 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 73.22 |
| Shannon entropy | 0.59564 |
| G+C content | 0.61745 |
| Mean single sequence MFE | -51.97 |
| Consensus MFE | -17.69 |
| Energy contribution | -17.38 |
| Covariance contribution | -0.31 |
| Combinations/Pair | 1.65 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.795529 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 18133377 117 - 23011544 GAUGAUGAGAAUAGCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGCUGCAGCUCCAGCUGCGCCGGCU .............(((((((.....)).((.((((.(((((((....))))))).)))))).....)))))((((((.(.(((((((((((...))))))))))).)...)))))). ( -63.50, z-score = -2.98, R) >droAna3.scaffold_12916 13397294 111 + 16180835 GAUGAUGAGAAUAGUCAGGCGGAGGGCACUGUGGAGGGCGCCAUUGGCGGCGCCAUCUACGGUAACCUGGCUCCGGCUCAGGCGCCAGCGGCUCCCCCGGCUCCGGCUGCG------ .............(.(((.((((((((((((((((.((((((......)))))).))))))))..((((((....)).)))).)))..(((.....))).))))).)))).------ ( -55.20, z-score = -1.06, R) >droYak2.chr2R 4637980 117 - 21139217 GAUGAUGAGAAUAGCCAGGCAGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUUGCGCCGGUUGGAGGGGUUGCAGCUGUUGCAGCUCCAGUUGCACCGGCU ..................(((((..((.((.((((.(((((((....))))))).))))))))..)).)))((((((....((((((((((...))))))))))......)))))). ( -49.50, z-score = -1.50, R) >droSec1.super_7 1759182 117 - 3727775 GAUGAUGAGAAUAGCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGUUGCAGCUCCAGCUGCGCCGGCU .............((((((......((.((.((((.(((((((....))))))).))))))))..))))))((((((.(.(((((((((((...))))))))))).)...)))))). ( -62.30, z-score = -2.72, R) >droSim1.chr2L 17809599 117 - 22036055 GAUGAUGAGAAUAGCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGCUGCAGCUCCAGCUGCGCCGGCU .............((((((......((.((.((((.(((((((....))))))).))))))))..))))))((((((.(.(((((((((((...))))))))))).)...)))))). ( -64.30, z-score = -2.88, R) >droWil1.scaffold_180764 3733478 117 + 3949147 GAUGAAGAGAAUAGUCAGGCUGAGGGCUCAAUGGAGGGUGCUGUGGGUGGUGUUAUUUAUGGUAAUAUUGCUGCAGUUCCUCAAGCAGUUGUUCAGCCAGUUGCGGCUCAACCUGUG ......(((........(((((((((((.....(((((.((((((.(..(((((((.....)))))))..))))))))))))....)))).)))))))........)))........ ( -37.19, z-score = -0.62, R) >droPer1.super_8 2503541 114 + 3966273 GAUGAUGAAAAUAGCCAGGCUGAGGGGACCAUGGAGGGUGCCAUUGGUGGGGCCAUUUAUGGCAAUAUCCCACCAGUUGGAGCGGCUGCUGCA---GCAGCUCCAGUCCAGCCAUCG (((.((....)).....(((((..((.(((......))).))(((((((((((((....)))).....)))))))))((((((.((((...))---)).))))))...)))))))). ( -49.80, z-score = -1.97, R) >dp4.chr4_group3 1439372 114 + 11692001 GAUGAUGAAAAUAGCCAGGCUGAGGGGACCAUGGAGGGUGCCAUUGGUGGGGCCAUUUAUGGCAAUAUCCCACCAGUUGGACCGGCUGCUGCA---GCAGCUCCAGUCCAGCCAUCG ...((((....((((...))))...((((..((((.(((.(((((((((((((((....)))).....)))))))).)))))).(((((....---)))))))))))))...)))). ( -48.90, z-score = -1.78, R) >droEre2.scaffold_4845 16452911 117 + 22589142 GAUGAUGAGAAUAGCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGUUGGAGGGGUUGCAGCCGUUGCAGCUCCAGCUGCCCCGGCU .............((((((......((.((.((((.(((((((....))))))).))))))))..))))))(((((..((.((((((((((...)))))))))).....))))))). ( -53.00, z-score = -0.72, R) >droVir3.scaffold_12963 17225711 102 - 20206255 GAUGAUGAGAAUAGCCAGGCUGAGGGCACCAUGGAGGGCGCCGUCGGUGGUGCUGUUUAUGGCAAUAUAAUAGCAGC------AGCCGCAGCACCGCCAACGCCUGCA--------- .............((.((((.((.(((.((......)).))).))(((((((((((....(((..............------.))))))))))))))...)))))).--------- ( -44.86, z-score = -2.27, R) >droGri2.scaffold_15252 14652839 90 - 17193109 GAUGAUGAGAAUAGCCAGGCGGAGGGCAGCAUGGAGGGCGCCGUUGGUGGUGCUGUCUAUGGCAAUAU------------------UGUUGCACCGCCAACGCCGGCA--------- .............(((.((((...(((..((((((.(((((((....))))))).))))))(((((..------------------.)))))...)))..))))))).--------- ( -43.10, z-score = -3.26, R) >consensus GAUGAUGAGAAUAGCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGAGGGGCUGCAGCAGCUGCAGCUCCAGCUGCGCCGGCU .............(((........)))....((((((((((((....)))))))..............................((....))........)))))............ (-17.69 = -17.38 + -0.31)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:49:23 2011