
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 17,877,516 – 17,877,622 |
| Length | 106 |
| Max. P | 0.841500 |

| Location | 17,877,516 – 17,877,622 |
|---|---|
| Length | 106 |
| Sequences | 10 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 93.61 |
| Shannon entropy | 0.13412 |
| G+C content | 0.32678 |
| Mean single sequence MFE | -31.68 |
| Consensus MFE | -25.56 |
| Energy contribution | -26.07 |
| Covariance contribution | 0.51 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.81 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.841500 |
| Prediction | RNA |
| WARNING | Out of training range. z-scores are NOT reliable. |
Download alignment: ClustalW | MAF
>dm3.chr2L 17877516 106 - 23011544 --UGUUUGUCAUGUGCAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CACACAAAAAUUUAUGUUUGCAUGCGACAAAA --..((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -33.80, z-score = -2.71, R) >droAna3.scaffold_12916 13165188 105 + 16180835 ---GUUUGUCGUGUGUAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CACACAAAAAUUUAUGUUUGCAUGCGACAAAA ---.((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -32.10, z-score = -2.46, R) >droEre2.scaffold_4845 16204140 106 + 22589142 --UGUUUGUCAUGUGCAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CACACAAAAAUUUAUGUUUGCAUGCGACAAAA --..((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -33.80, z-score = -2.71, R) >droYak2.chr2R 17914244 106 + 21139217 --UGUUUGUCAUGUGCAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CACACAAAAAUUUAUGUUUGCAUGCGACAAAA --..((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -33.80, z-score = -2.71, R) >droSec1.super_7 1515883 106 - 3727775 --UGUUUGUCAUGUGCAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CACACAAAAAUUUAUGUUUGCAUGCGACAAAA --..((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -33.80, z-score = -2.71, R) >droSim1.chr2L 17545895 106 - 22036055 --UGUUUGUCAUGUGCAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CACACAAAAAUUUAUGUUUGCAUGCGACAAAA --..((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -33.80, z-score = -2.71, R) >droGri2.scaffold_15252 14345672 104 - 17193109 --UGCUUGUCAUGUGUAAAUGC-AUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGGCACACACAAAAAUUUAUGAUUGCAUUCAAAC--- --((..((((((((((((((((-.((.....))....)))))))).....((((((....))))))....))))))))..))..........(((......)))...--- ( -23.30, z-score = -0.12, R) >droMoj3.scaffold_6500 21908654 104 - 32352404 --UGUUUGCUAUGUGUAAAUGC-AUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGCCGCACACAAAAAUUUAUGAUUGCAUAUGGAA--- --((((.(((((((......))-))))).))))...((((..((((....((((((....))))))...))))))))(((..((........))..)))........--- ( -28.40, z-score = -1.38, R) >droPer1.super_8 2231203 108 + 3966273 UGUGUUUGUCAUGUGUAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CAUACAAAAAUUUAUGUUUGCAUGCGACAAAA ....((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -32.00, z-score = -2.15, R) >dp4.chr4_group3 1170418 108 + 11692001 UGUGUUUGUCAUGUGUAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG--CAUACAAAAAUUUAUGUUUGCAUGCGACAAAA ....((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((.--((.(((........))).))))))))))))). ( -32.00, z-score = -2.15, R) >consensus __UGUUUGUCAUGUGCAAAUGCAAUGGCUGACAAAAGGCAUUUGCAUAUUUAAUGUUAUUACGUUAUUUUGCAUGG__CACACAAAAAUUUAUGUUUGCAUGCGACAAAA ....((((((((((((((((((..((.....))....)))))))))))).((((((....))))))....(((((...((.(((........))).))))))))))))). (-25.56 = -26.07 + 0.51)

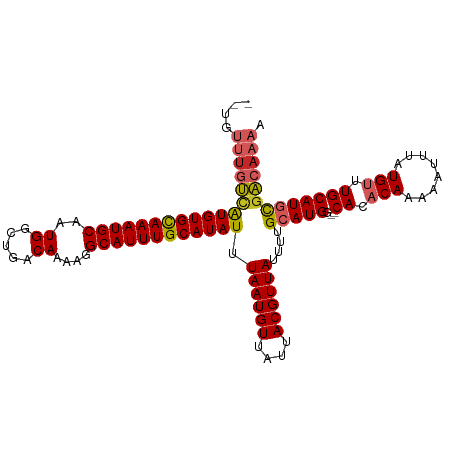
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:48:55 2011