
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 16,893,335 – 16,893,455 |
| Length | 120 |
| Max. P | 0.603624 |

| Location | 16,893,335 – 16,893,455 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.58 |
| Shannon entropy | 0.54912 |
| G+C content | 0.56690 |
| Mean single sequence MFE | -45.13 |
| Consensus MFE | -20.19 |
| Energy contribution | -19.47 |
| Covariance contribution | -0.71 |
| Combinations/Pair | 1.68 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.45 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.603624 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 16893335 120 + 23011544 GCGCAAGAUUUGCCAGCGCUCUGCCGGUUGGAUCGACUGUUGGGCAGUGCCCAUUCUGGCACUGUCAAUUCAGUAUGUUUAUCAUCCAGUUCCACCACUAGGACCUGAUCCUCAAGCGGA ((((...........))))(((((..(..((((((((((...(((((((((......)))))))))....))))..........((((((......))).)))..)))))).)..))))) ( -43.20, z-score = -2.13, R) >droEre2.scaffold_4845 15248423 120 - 22589142 GCGCAAGAUUUGCCAGCGCUCUGCCGGUUGGAUCGACUGCUGGGCAGUGCCUAUUCUGGCACUGUCGAUUCAGUAUGUCUAUCAUCCUGUUCCACCAUUAGGACCAGAUCCUCAAGCGGA ((((...........))))(((((..(..((((((((((((((((((((((......)))))))))....))))).))).....(((((.........)))))...))))).)..))))) ( -47.40, z-score = -2.88, R) >droYak2.chr2R 16928263 120 - 21139217 GCGCAAGAUUUGUCAGCGCUCUGCCGGUUGGAUUGACUGCUGGGCAGUGCCUAUUCUGGCACUGUCAAUUCAGUACGUCUAUCAUCCUGUUCCACCAUUAGGACCAGAUCCUCAAGCGGA ((((..((....)).))))....(((.((((((.(((((((((((((((((......)))))))))....))))).))).)))...(((.(((.......))).))).....))).))). ( -45.90, z-score = -2.95, R) >droSim1.chr2L 16577214 120 + 22036055 GCGGAAGAUUUGCCAGCGCUCUGCCGGUUGGAUCGACUGUUGGGCAGUGCCCAUUCUGGCACUGUCAAUUCAGUAUGUUUAUCAUCCAGUUCCACCACUAGGACCAGAUCCUCAAGCGGA ((((........)).))..(((((..(..(((((.((((...(((((((((......)))))))))....))))..........((((((......))).)))...))))).)..))))) ( -40.70, z-score = -1.23, R) >droSec1.super_7 530411 120 + 3727775 GCGCAAGAUUUGCCAGCGCUCUGCCGGUUGGAUCGACUGUUGGGCAGUGCCCAUUCUGGCACUGUCAAUUCAGUAUGUUUAUCAUCCAGUUCCACCACUAGGACCAGAUCCUCAAGCGGA ((((...........))))(((((..(..(((((.((((...(((((((((......)))))))))....))))..........((((((......))).)))...))))).)..))))) ( -43.30, z-score = -2.26, R) >droWil1.scaffold_180703 2614758 120 - 3946847 GCGAAAAAUUUGCCAGCGUAGUGCCGGUUGGAUUGAUUGCUGGGCUGUGCCCAUAUUGGCAUUGUCCAUCCAAUAUGUCUAUCAUCCGGUGCCGCCGUUGGGUCCCGAUCCGCAAGCGGA ............((((((..(..((((.(((((..((((.(((((.(((((......))))).)))))..))))..)))))....))))..)..).))))).......((((....)))) ( -46.00, z-score = -1.56, R) >droAna3.scaffold_12916 12243185 120 - 16180835 GCGCAAGAUCUGCCAGCGCUCCGCCGGCUGGAUCGACUGCUGGGCCGUGCCUAUCCUGGCGCUGUCCAUUCAGUACGUGUAUCAUCCAGUUCCGCCGCUGGGUCCUGAUCCCCAGGCAGA .(....).((((((((((....((.((((((((((((((.(((((.(((((......))))).)))))..)))).))......)))))).)).))))))(((........))).)))))) ( -49.70, z-score = -1.36, R) >droVir3.scaffold_13246 1520000 120 - 2672878 GCGUAAGAUUUGCCAGCGCAGCGCCGGCUGGAUUGAUUGCUGGGCCGUGCCCAUUCUGGCGCUGUCCAUACAAUAUGUAUAUCAUCCGGUGCCGCCGCUGGGACCCGAUCCGCAGGCGGA ............(((((((.(((((((.(((..........((((.(((((......))))).))))((((.....)))).))).))))))).).)))))).......((((....)))) ( -49.40, z-score = -0.38, R) >droMoj3.scaffold_6500 4759130 120 + 32352404 GCGUAAGAUUUGCCAGCGCAGCGCCGGCUGGAUCGAUUGUUGGGCGGUGCCCAUAUUAGCACUGUCCAUCCAGUAUGUCUAUCAUCCGGUGCCGCCGCUGGGGCCUGAUCCCCAGGCGGA ............(((((((.(((((((.(((((..((((.((((((((((........))))))))))..))))..)))))....))))))).).)))))).(((((.....)))))... ( -56.90, z-score = -2.60, R) >droGri2.scaffold_15252 16073629 120 + 17193109 GCGCAAGAUUUGUCAGCGCAGCGCCGGUUGGAUUGACUGUUGGGCGGUGCCCAUUUUAGCUUUGUCCAUUCAAUAUGUAUAUCAUCCAGUGCCGCCGCUGGGACCAGAUCCUCAGGCGGA .(((..((((((....(.(((((.(((((((((.(((((.(((((...)))))...)))...(((.(((.....))).))))))))))..)))).))))).)..)))))).....))).. ( -39.00, z-score = 0.35, R) >dp4.chr4_group3 32146 120 - 11692001 GCGCAAGAUCUGCCAGCGCUCGGCUGGAUGGAUCGACUGCUGGGCCGUGCCCAUCCUGGCGCUGUCUAUCCAGUACGUUUACCAUCCGGUGCCCCCGCUGGGACCUGAUCCACAGGCGGA .(....).((((((((((...((((((((((..((((((.(((((.(((((......))))).)))))..)))).))....)))))))..)))..))))))..((((.....)))))))) ( -51.10, z-score = -1.00, R) >droPer1.super_8 1061317 120 - 3966273 GCGCAAGAUCUGCCAGCGCUCGGCUGGAUGGAUCGACUGCUGGGCCGUGCCCAUCCUGGCGCUGUCUAUCCAGUACGUUUACCAUCCGGUGCCCCCGCUGGGACCUGAUCCACAGGCGGA .(....).((((((((((...((((((((((..((((((.(((((.(((((......))))).)))))..)))).))....)))))))..)))..))))))..((((.....)))))))) ( -51.10, z-score = -1.00, R) >anoGam1.chr3R 49927674 120 - 53272125 GCGGCGGGUUUGUCAACGAGCGGUCGGAUGGAUUGACUGCUGGACCGUGCCGAUAGUGGCACUUUCGAUCAAGUACAUCUACCAUCCGAUGCCACCACUGGGACCGGAUCCGCAGGCCGA .(((((((((((....)))))((((((((((..(((((..(.((..((((((....))))))..)).)...))).))....)))))))..))).)).((((((.....))).))))))). ( -46.70, z-score = -1.03, R) >apiMel3.Group12 2256466 120 - 9182753 GAGAAAUGUUUGUCAGAAAGGAACAGGCUGGGUUGAUUGUUGGUCUGUACCAAUUGUGGCAUUAAGUAUUAAUUAUACAUAUCAUCCAUGUCCACCUUUAGGACCUACACCACAAGCAAA ......(((((((......((.((((((..(........)..)))))).))...(((((......((((.....))))...........((((.......)))))))))..))))))).. ( -28.70, z-score = -0.32, R) >triCas2.ChLG6 6676207 114 + 13544221 GCGGAACGAAUGUCAAAA------UGGUUGGGUGGACUUUUGGUGGGUGCCAAUCGGCGCGAUUAACAUUGUCUACACUUACCACCCGUGCCCACCCCUUGGUACCACCCCCCAGGCCGA .(((...(.....)....------(((..(((((((((...((((((((((....)))(((((....))))).................)))))))....))).)))))).)))..))). ( -37.90, z-score = 0.03, R) >consensus GCGCAAGAUUUGCCAGCGCUCUGCCGGUUGGAUCGACUGCUGGGCAGUGCCCAUUCUGGCACUGUCCAUUCAGUAUGUUUAUCAUCCGGUGCCACCGCUGGGACCUGAUCCUCAGGCGGA ((.........(((.((.....)).))).(((((.((((...(((.((((........)))).)))....))))...........(((((......))).))....)))))....))... (-20.19 = -19.47 + -0.71)

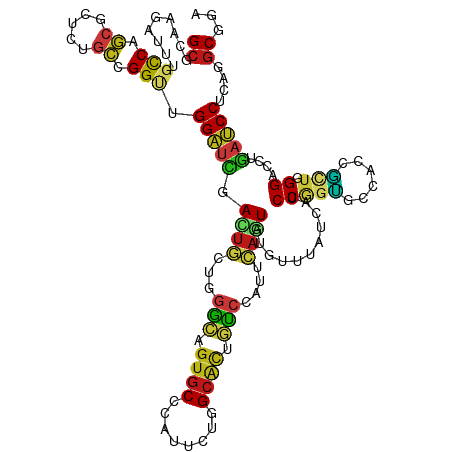
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:46:41 2011