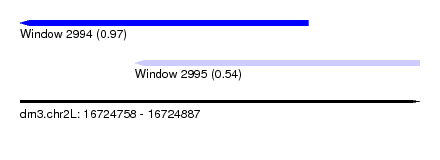
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 16,724,758 – 16,724,887 |
| Length | 129 |
| Max. P | 0.965588 |

| Location | 16,724,758 – 16,724,851 |
|---|---|
| Length | 93 |
| Sequences | 13 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 88.11 |
| Shannon entropy | 0.25873 |
| G+C content | 0.35814 |
| Mean single sequence MFE | -23.45 |
| Consensus MFE | -18.14 |
| Energy contribution | -18.59 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.39 |
| Structure conservation index | 0.77 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.965588 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
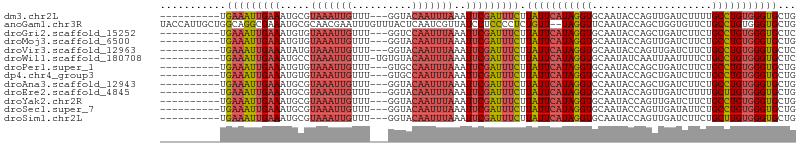
>dm3.chr2L 16724758 93 - 23011544 ----------UGAAAUUGAAAUGCGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUUUGCCUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((.((((....)))).......)))))))))))... ( -24.50, z-score = -2.88, R) >anoGam1.chr3R 18012116 104 + 53272125 UACCAUUGCUGGCAGGCGAAAUGCGCAACGAAUUUGUUUACUCAAUCGUUAUCCUCCCCUCUGUU--UAGGUUCAAUACCAGCUGGUGUUCUGCCUGUGGGUGCUG ...((..(((.(((((((.((((((((((((..(((......))))))))...............--..(((.....))).))..))))).))))))).)))..)) ( -27.30, z-score = -0.46, R) >droGri2.scaffold_15252 9753512 93 - 17193109 ----------UGAAAUUGAAAUGUGUAAAUUGUUU---GGUCCAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGCUGAUCUUCUGCCUGUGGGUGCUC ----------.(((((((((.....(((((((...---....)))))))..))))))))).(((((((((((((.......)).........)))))))))))... ( -21.50, z-score = -1.87, R) >droMoj3.scaffold_6500 25691015 93 - 32352404 ----------UGAAAUUGAAAUGUGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUCUGCCUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((.((((....)))).......)))))))))))... ( -24.50, z-score = -3.09, R) >droVir3.scaffold_12963 6536502 93 - 20206255 ----------UGAAAUUGAAAUAUGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUCUGCCUGUGGGUGCUC ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((.((((....)))).......)))))))))))... ( -24.50, z-score = -3.24, R) >droWil1.scaffold_180708 10779562 95 + 12563649 ----------UGAAAUUGAAAUGCCUAAAUUGUUU-UGUGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUAUCAAUUAAUUUUCUGCCUGUGGGUGCUC ----------.(((((((((.....((((((((..-.....))))))))..))))))))).(((((((((((....................)))))))))))... ( -22.15, z-score = -2.94, R) >droPer1.super_1 5738371 93 + 10282868 ----------UGAAAUUGAAAUGUGUAAAUUGUUU---GUGCCAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGCUGAUCUUCUGCCUGUGGGUGCUG ----------.(((((((((.....(((((((...---....)))))))..))))))))).(((((((((((((.......)).........)))))))))))... ( -21.50, z-score = -1.87, R) >dp4.chr4_group3 8634227 93 + 11692001 ----------UGAAAUUGAAAUGUGUAAAUUGUUU---GUGCCAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGCUGAUCUUCUGCCUGUGGGUGCUG ----------.(((((((((.....(((((((...---....)))))))..))))))))).(((((((((((((.......)).........)))))))))))... ( -21.50, z-score = -1.87, R) >droAna3.scaffold_12943 1101482 93 + 5039921 ----------UGAAAUUGAAAUGCGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUCCAAUACCAGCUGAUCUUCUGCCUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((....................)))))))))))... ( -22.85, z-score = -2.39, R) >droEre2.scaffold_4845 19796323 93 - 22589142 ----------UGAAAUUGAAAUGCGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUUUGCUUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((.((((....)))).......)))))))))))... ( -21.80, z-score = -2.03, R) >droYak2.chr2R 16760971 93 + 21139217 ----------UGAAAUUGAAAUGCGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUCUGCCUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((.((((....)))).......)))))))))))... ( -24.50, z-score = -2.89, R) >droSec1.super_7 371548 93 - 3727775 ----------UGAAAUUGAAAUGCGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUAUUCUGCCUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).((((((((((((.((((.(....).)))).))))))))))))... ( -26.50, z-score = -3.56, R) >droSim1.chr2L 16422857 93 - 22036055 ----------UGAAAUUGAAAUGCGUAAAUUGUUU---GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUCUGCUUGUGGGUGCUG ----------.(((((((((.....((((((((..---...))))))))..))))))))).(((((((((((.((((....)))).......)))))))))))... ( -21.80, z-score = -2.03, R) >consensus __________UGAAAUUGAAAUGCGUAAAUUGUUU___GGUACAAUUUAAAUUCGAUUUCUUAUUCAUAGGUGCAAUACCAGUUGAUCUUCUGCCUGUGGGUGCUG ...........(((((((((.....(((((((..........)))))))..))))))))).(((((((((((....................)))))))))))... (-18.14 = -18.59 + 0.45)

| Location | 16,724,795 – 16,724,887 |
|---|---|
| Length | 92 |
| Sequences | 14 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 78.99 |
| Shannon entropy | 0.46267 |
| G+C content | 0.34974 |
| Mean single sequence MFE | -21.92 |
| Consensus MFE | -7.45 |
| Energy contribution | -8.39 |
| Covariance contribution | 0.94 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.543278 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 16724795 92 - 23011544 GUAUCUUGUCGACCCGUGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGCGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))).--------- ( -22.20, z-score = -2.76, R) >apiMel3.GroupUn 377964998 99 + 399230636 GUGUGCUAUCAGGAAGAGUAAAUUUCCAAGUA---CAAUAUCAACUUAUAUUCUGUAUAUGGGU---AUUAACAUUUAA--UCCAUUGCUUGCAGAAAAAAUGAAUA .((((((....(((((......))))).))))---)).............(((((((.((((((---...........)--)))))....))))))).......... ( -19.60, z-score = -1.41, R) >anoGam1.chr3R 18012153 106 + 53272125 GCAUCCUUUCGUCGCGUGUCAACUUCCAGGUGCGCUAACUACCAUUGCUGGCAGGCGAAAUGCGC-AACGAAUUUGUUUACUCAAUCGUUAUCCUCCCCUCUGUUUA ((((...(((((((((..((........))..)))......(((....)))..))))))))))..-(((((..(((......))))))))................. ( -24.00, z-score = -0.88, R) >droGri2.scaffold_15252 9753549 92 - 17193109 GGGUCCUGUCCACGCGCGUCAAUUUCCAAGUG----AGUUUGAAAUUGAAAUGUGUAAAUUGUUU--GGUCCAAUUUAAAUUCGAUUUCUUAUUCAUA--------- (((...((.((....).).))...)))..(((----(((..(((((((((.....(((((((...--....)))))))..)))))))))..)))))).--------- ( -19.70, z-score = -1.65, R) >droMoj3.scaffold_6500 25691052 92 - 32352404 GCAUCCUGUCGACGCGUGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGUGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))).--------- ( -22.50, z-score = -2.36, R) >droVir3.scaffold_12963 6536539 92 - 20206255 GCAUCCUGUCGACGCGUGUCAACUUUCAGGUG----AGUUUGAAAUUGAAAUAUGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- ....((((..(((....)))......))))((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))..--------- ( -23.10, z-score = -2.81, R) >droWil1.scaffold_180708 10779599 94 + 12563649 GGAUUCUGUCCACUCGUGUUAAUUUCCAGGUG----AGUUUGAAAUUGAAAUGCCUAAAUUGUUUUGUGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- ((((...))))..................(((----(((..(((((((((.....((((((((.......))))))))..)))))))))..)))))).--------- ( -21.30, z-score = -2.80, R) >droPer1.super_1 5738408 92 + 10282868 GCAUUCUGUCGACUCGCGUCAACUUUCAGGUG----AGUUUGAAAUUGAAAUGUGUAAAUUGUUU--GUGCCAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....(((((((...--....)))))))..)))))))))..)))))).--------- ( -20.50, z-score = -1.92, R) >dp4.chr4_group3 8634264 92 + 11692001 GCAUUCUGUCGACUCGCGUCAACUUUCAGGUG----AGUUUGAAAUUGAAAUGUGUAAAUUGUUU--GUGCCAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....(((((((...--....)))))))..)))))))))..)))))).--------- ( -20.50, z-score = -1.92, R) >droAna3.scaffold_12943 1101519 92 + 5039921 GCAUCCUGUCGACCCGUGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGCGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))).--------- ( -22.20, z-score = -2.55, R) >droEre2.scaffold_4845 19796360 92 - 22589142 GUAUCCUGUCGACCCGUGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGCGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))).--------- ( -22.20, z-score = -2.79, R) >droYak2.chr2R 16761008 92 + 21139217 GUAUCCUGUCCACUCGUGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGCGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- ....((((..................))))((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))..--------- ( -20.47, z-score = -2.50, R) >droSec1.super_7 371585 92 - 3727775 GUAUCCUGUCGACCCGGGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGCGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))).--------- ( -24.30, z-score = -2.76, R) >droSim1.chr2L 16422894 92 - 22036055 GUAUCCUGUCGACCCGGGUCAACUUCCAGGUG----AGUUUGAAAUUGAAAUGCGUAAAUUGUUU--GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA--------- .......((.(((....))).))......(((----(((..(((((((((.....((((((((..--...))))))))..)))))))))..)))))).--------- ( -24.30, z-score = -2.76, R) >consensus GCAUCCUGUCGACCCGUGUCAACUUCCAGGUG____AGUUUGAAAUUGAAAUGCGUAAAUUGUUU__GGUACAAUUUAAAUUCGAUUUCUUAUUCAUA_________ ....((((..................))))...........(((((((((.....(((((((.........)))))))..))))))))).................. ( -7.45 = -8.39 + 0.94)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:46:12 2011