
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 1,606,519 – 1,606,637 |
| Length | 118 |
| Max. P | 0.984378 |

| Location | 1,606,519 – 1,606,637 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.69 |
| Shannon entropy | 0.52894 |
| G+C content | 0.52922 |
| Mean single sequence MFE | -40.15 |
| Consensus MFE | -24.06 |
| Energy contribution | -24.31 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.16 |
| SVM RNA-class probability | 0.984378 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 1606519 118 + 23011544 GGGCGACUUACGUUGUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGACGGCAUCUUCUGACAAUAUCUGAC-GCCGACCAUUGAG-GGGACCGAGGGCAGGGGUCGUGGUCGGCGUCAGC (((((((....))))))).((((((((((((((((...........)))))))....))).))))))((((-(((((((((((.(-....)))).(((....))).)))))))))))).. ( -46.40, z-score = -2.82, R) >droSim1.chr2L 1580346 118 + 22036055 GGGCGACUCACGUUGUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGACGGCAUCUUCUGACAAUAUCUGAC-GCCGACCAUUGAG-GGGCACGAGGGCAGGGGGCGUGGUCGGCGUCAGC (((((((....))))))).((((((((((((((((...........)))))))....))).))))))((((-(((((((((....-..((......)).......))))))))))))).. ( -44.52, z-score = -2.23, R) >droSec1.super_14 1549607 118 + 2068291 GGGCGACUCACGUUGUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGACGGCAUCUUCUGACAAUAUCUGAC-GCCGACCAUUGAG-GGGCACGAGGGCAGGGGGCGUGGUCGGCGUCAGC (((((((....))))))).((((((((((((((((...........)))))))....))).))))))((((-(((((((((....-..((......)).......))))))))))))).. ( -44.52, z-score = -2.23, R) >droYak2.chr2L 1579851 118 + 22324452 GGGCGACUCACGUUGUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGACGGCAUCUUCUGACAAUAUCUGAC-GCCGACCAUUGAG-GGGCACGGGGGCAGGGGGCAUGGUCGGCGUCAGC (((((((....))))))).((((((((((((((((...........)))))))....))).))))))((((-(((((((((....-..((......)).......))))))))))))).. ( -44.22, z-score = -2.42, R) >droEre2.scaffold_4929 1652834 118 + 26641161 GGGCGACUCACGUUGUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGAUGGCAUCUUCUGACAAUAUCUGAC-GCCGACCAUUGAG-GGGCGCGGGGGCAGGGGGCAUGGUCGGCGUCAGC (((((((....))))))).(((((((((((((..(...........)..))))....))).))))))((((-(((((((((....-..((......)).......))))))))))))).. ( -42.82, z-score = -1.67, R) >droAna3.scaffold_12916 15006208 106 + 16180835 GGGCGGCUCAGCUUAUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGAUGGCAUUUUCUGACAAUAUCUGAC-GCCGGCUAUUGAG-G------------CGGGGAAUGGCCGGUGUCAGC ..((.((((((.(((...((((((....))))))...))).))))).).))...............(((((-((((((((((...-.------------.....))))))))))))))). ( -35.20, z-score = -1.63, R) >dp4.chr4_group3 10207873 118 + 11692001 GGGCGGCUUACGUUAUCCUGAUAUUUCAAUGUUGUAUUAAAUCUGGAUGGCAUUUUCUGACAAUAUCUGAC-GCCGGCCAUCGAG-GGGAGUGUUGCCACAAGGGAUGGACGGCGUCAGC (((..((....))..)))(((....)))(((((((...((((((....)).))))....)))))))(((((-((((.(((((...-(((.......)).)....))))).))))))))). ( -35.90, z-score = -0.66, R) >droPer1.super_8 1414554 118 + 3966273 GGGCGGCUUACGUUAUCCUGAUAUUUCAAUGUUGUAUUAAAUCUGGAUGGCAUUUUCUGACAAUAUCUGAC-GCCGGCCAUCGAG-GGGAGUGUUGCCACAAGGGAUGGUCGGCGUCAGC (((..((....))..)))(((....)))(((((((...((((((....)).))))....)))))))(((((-((((((((((...-(((.......)).)....))))))))))))))). ( -40.30, z-score = -1.81, R) >droWil1.scaffold_180708 3411705 115 + 12563649 GGGCGGCUUAUGUUAUCCUGAUAUUUCAAUGUUGUAUUAAAUUUGGAUGGCAUUUUCUGACAAUAUCUGAC-GCCGGCCAUUGAU-GGGGUUAGGG---GGGAAGAUGGUCGGCGUCAGC ...(((...(((((((((((((((.........)))))).....)))))))))...))).......(((((-((((((((((...-..........---.....))))))))))))))). ( -39.07, z-score = -2.73, R) >anoGam1.chr3R 51405898 120 - 53272125 GACCGGCUGCUCGUUUCCUGAUACGGCGAUCCGGCGCACGAUUACGGCGAACUUUUCUGACAGCGACUACCCGCCGACGGCCGGACGGUUGCGGGGCGCUGCCCCACGAUCGACGACCGC ..(((((((.((((((((((((.(((((......))).)))))).)).))))..........(((......))).))))))))).((((((.(((((...))))).))))))........ ( -47.00, z-score = -0.78, R) >droMoj3.scaffold_6500 29159738 94 - 32352404 GGGCGACUCAUGUUAUCCUGAUAUUUCAAUGUUGUAUUAAAUUUGGAUGGCAUUUUCUGACAAUAUCUGAC-GCCGGCCA-------------------------AUGGUCGGUGUCAGC ....((...(((((((((((((((.........)))))).....)))))))))..)).........(((((-(((((((.-------------------------..)))))))))))). ( -30.70, z-score = -2.76, R) >droVir3.scaffold_12963 7671128 102 + 20206255 GGGCGGCUCAUGUUAUCCUGAUAUUUCAAUGUUGUAUUAAAUUUGGAUGGCAUUUUCUGACAAUAUCUGAC-GCCGGCCAUGG----------------CAGUGGGUGG-CGGUGCAGGC ...(((...(((((((((((((((.........)))))).....)))))))))...)))......((((.(-((((.((((..----------------..)))).)))-))...)))). ( -31.10, z-score = -0.47, R) >consensus GGGCGACUCACGUUAUCCUGAUAUUUCAAUGUUGUAUUAAACUGGGAUGGCAUUUUCUGACAAUAUCUGAC_GCCGACCAUUGAG_GGGCGCGGGGGCAGGGGGCAUGGUCGGCGUCAGC ...........(((((((((((((.........)))))).....)))))))...............(((((.(((((((((........................)))))))))))))). (-24.06 = -24.31 + 0.25)

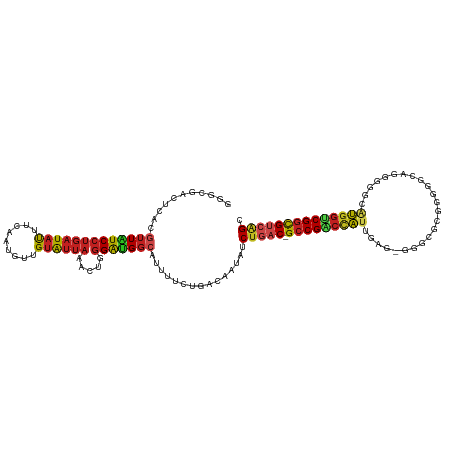
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:08:47 2011