
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 1,603,501 – 1,603,621 |
| Length | 120 |
| Max. P | 0.521987 |

| Location | 1,603,501 – 1,603,621 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.65 |
| Shannon entropy | 0.41508 |
| G+C content | 0.54056 |
| Mean single sequence MFE | -45.05 |
| Consensus MFE | -21.18 |
| Energy contribution | -20.98 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.54 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.47 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.521987 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 1603501 120 + 23011544 CAGCAGUGCCGCGAAGUGAGGACUGUCGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUAGUCUUCAUCGCU ..........((((..(((((((((((((...((((((((((.....)))(((........))).))))..)))((((((.((((....)).))..))))))))))))))))))))))). ( -50.20, z-score = -2.52, R) >droSim1.chr2L 1576909 120 + 22036055 CAGCAGUGCCGCGAAGUGAGGACUGUCGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUAGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUAGUCUUCAUCGCU ..........((((..(((((((((((((...((((((((((.....))).........(....)))))..)))((((((.((((....)).))..))))))))))))))))))))))). ( -48.50, z-score = -2.20, R) >droSec1.super_14 1546581 120 + 2068291 CAGCAGUGUCGCGAAGUGAGGACUGUCGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUAGUCUUCAUCGCU ..........((((..(((((((((((((...((((((((((.....)))(((........))).))))..)))((((((.((((....)).))..))))))))))))))))))))))). ( -50.20, z-score = -2.67, R) >droYak2.chr2L 1576677 120 + 22324452 CAGCAGUGUCGCGAAGUUCGCACUGUCGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUCUUCAUCGCU .(((..(((((((.....)))......(((.....)))..........)))).((..(((((..(((((((((..((((....))))..))).....))))))..)))))..))...))) ( -45.60, z-score = -1.59, R) >droEre2.scaffold_4929 1649698 120 + 26641161 CAGCAGUGCCGCGAGGUUCGCACGGUCGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUCUUCAUCGCU .(((.(.((((((((((((....))..))))))).))))(((.....)))...((..(((((..(((((((((..((((....))))..))).....))))))..)))))..))...))) ( -51.10, z-score = -2.27, R) >droAna3.scaffold_12916 15003254 120 + 16180835 CAGCAGUGCCGCGAAGUGAGGACAGUGGCCUCCCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCCGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUCUUCAUUGCU ..((......))..(((((((((.((((((.....))))(((.....))).......))(((..(((((((((((((.....)))))))((....))))))))..))))))))))))... ( -48.90, z-score = -2.45, R) >dp4.chr4_group3 10204565 120 + 11692001 CAGCAGUGCCGCGAGGUCCGCACUGUGGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUGUUCAUUGCU .(((((((((((((((.((((...)))))))))).))).(((.....)))...((..(((((..(((((((((..((((....))))..))).....))))))..)))))..)))))))) ( -56.90, z-score = -3.39, R) >droPer1.super_8 1411138 120 + 3966273 CAGCAGUGCCGCGAGGUCCGCACUGUGGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUGUUCAUUGCU .(((((((((((((((.((((...)))))))))).))).(((.....)))...((..(((((..(((((((((..((((....))))..))).....))))))..)))))..)))))))) ( -56.90, z-score = -3.39, R) >droWil1.scaffold_180708 3408596 120 + 12563649 CAGCAAUGUCGCGAGGUGCGAACAGUGGCCUCCCAAGCUUCAAAUAUGGAUAAAAUAACAAUAGCGGCAAGUGCAGCAAUUUGUGGUACAAUUAUUGUGGCAGUGAUUGUGUUUAUAGCA ..(((.(((((((((((((.....)).)))))(((...........)))..............))))))..))).((((((..((.((((.....)))).))..)))))).......... ( -29.70, z-score = 0.63, R) >droMoj3.scaffold_6500 29155424 120 - 32352404 CAGCAGUGCCGCGAGGUGCGCACUGUCGCCUCCCAGACCUCGAACAUGGAUAAGAUAACGAUUGCAGCAAGUGCCGCCAUUUGUGGCACCAUCAUUGUCGCUGUGAUUGUCUUCAUUGCA ..(((((.((((((((((((......))).......))))))....)))....((..(((((..((((..(((((((.....)))))))((....))..))))..)))))..))))))). ( -43.01, z-score = -1.68, R) >droVir3.scaffold_12963 7667997 120 + 20206255 CAACAAUGCCGCGAGGUGCGUACCGUUUCCUCGCAGACCUCCAACAUGGAUAAGAUAACCAUAGCGGCCAGUGCAGCCAUUUGUGGCACCAUCAUUGUGGCUGUGAUUGUGUUCAUUGCG ..((((((((((((((((((...........)))..)))))....((((.........)))).)))))....((((((((.((((....)).))..)))))))).))))).......... ( -38.10, z-score = -0.66, R) >droGri2.scaffold_15252 10754167 120 + 17193109 CAGCAGUGUCGCGAGGUGCGCACCGUCUCCUCGCAGACAUCCAACAUGGAUAAGAUAACGAUAGCGGCAAGUGCCGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUAUUCAUCGCA ..((...(((((((((.(((...)))..)))))).)))((((.....))))..((..(((((.(((((..(((((((.....)))))))(((....)))))))).)))))..))...)). ( -46.60, z-score = -2.60, R) >anoGam1.chr3R 51400380 120 - 53272125 ACCCAGUGCCGGGAGGUGCGCACCGUAGCCUCGCCAGCGUCCAACAUGGACAAGAUUACGAUCGCGGCCAGUGCGGCGAUCUGCGGCACGAUCAUUGUCGCGGUGAUCGUCUUCAUUGCC .....(((((((((((((((...)))).)))).)).((((((.....))))........((((((.((....)).)))))).)))))))(((....)))(((((((......))))))). ( -51.40, z-score = -1.60, R) >apiMel3.GroupUn 175214822 120 + 399230636 AAUCAAUGCAAAGAAGUAAGAACAGAAAAUUCGCCUACAUCUAAUAUGGAUAAGAUUACCAUUGCUGCGAGCGCAGCCAUAUGUGCCACUAUAGUGGUUGCCGUGAUAAUAUUUAUUGUU .......(((((((.(((.(((.......)))...))).)))....((((((..((((((((.(((((....)))))...))).(((((....)))))....)))))..)))))))))). ( -24.70, z-score = -0.17, R) >triCas2.ChLG5 9329444 120 + 18847211 UCACAAUGUCGUGAAGUGAGAACAGUUUCCACCGCUUCUUCCAACAUGGACAAAAUCACAAUUGCUGCAAGUGCAGCGAUUUGUGGAACCAUUGUAGUUGCCGUGGUCAUUUUCGUAGCG ((((......)))).((..(((.(((..((((.((..(((((.....))).....(((((((((((((....))))))).)))))).........))..)).))))..))))))...)). ( -34.00, z-score = -1.18, R) >consensus CAGCAGUGCCGCGAGGUGCGCACUGUCGCCUCGCAGGCCUCCAACAUGGACAAGAUAACGAUUGCGGCCAGUGCGGCCAUUUGUGGCACCAUCAUUGUGGCCGUGAUUGUCUUCAUUGCU ..((((((((((..(((....)))...............(((.....))).............)))))..((((.((.....)).))))....))))).((.((((......)))).)). (-21.18 = -20.98 + -0.20)

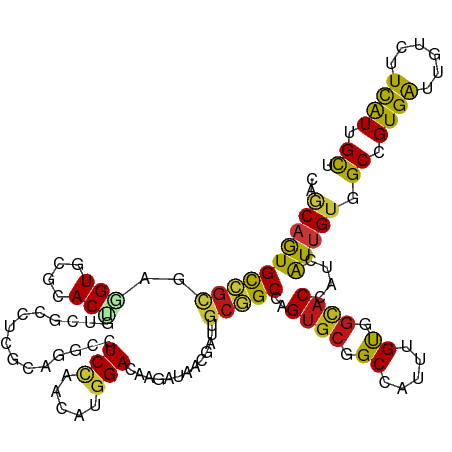
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:08:46 2011