| Sequence ID | dm3.chr2L |
|---|---|
| Location | 16,374,534 – 16,374,668 |
| Length | 134 |
| Max. P | 0.942578 |

| Location | 16,374,534 – 16,374,668 |
|---|---|
| Length | 134 |
| Sequences | 12 |
| Columns | 140 |
| Reading direction | forward |
| Mean pairwise identity | 90.10 |
| Shannon entropy | 0.22060 |
| G+C content | 0.32980 |
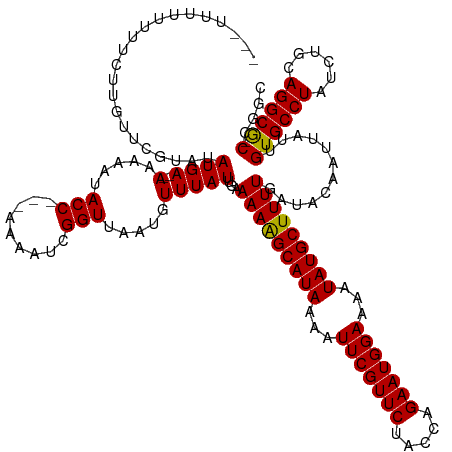
| Mean single sequence MFE | -31.07 |
| Consensus MFE | -24.07 |
| Energy contribution | -23.87 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.77 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.48 |
| SVM RNA-class probability | 0.942578 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
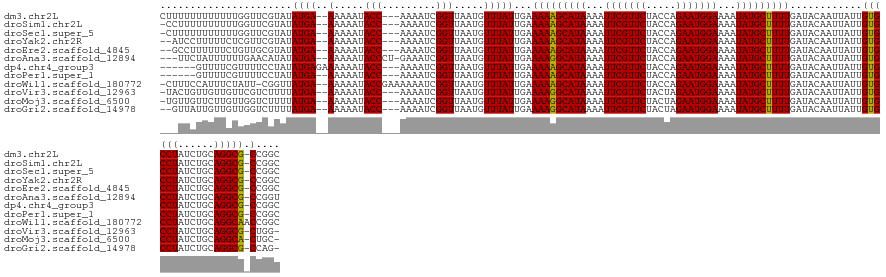
>dm3.chr2L 16374534 134 + 23011544 CUUUUUUUUUUUUGGUUCGUAUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC .........(((((((..........--......)))---))))((((((((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......)))))-)))). ( -31.08, z-score = -2.09, R) >droSim1.chr2L 16082931 133 + 22036055 -CCUUUUUUUUUUGGUUCGUAUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC -........(((((((..........--......)))---))))((((((((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......)))))-)))). ( -31.08, z-score = -2.09, R) >droSec1.super_5 5099872 133 + 5866729 -CUUUUUUUUUUUGGUUCGUAUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC -........(((((((..........--......)))---))))((((((((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......)))))-)))). ( -31.08, z-score = -2.15, R) >droYak2.chr2R 2828794 132 + 21139217 --AUCCUUUUUCUCGUUCGUAUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC --..((............((((((((--(.....(((---......))).....)))))...(((((((((...(((((((.....)))))))...))))))))).)))).......((((((......)))))-).)). ( -30.10, z-score = -2.25, R) >droEre2.scaffold_4845 19438704 132 + 22589142 --GCCUUUUUUCUGUUGCGUAUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC --(((.............((((((((--(.....(((---......))).....)))))...(((((((((...(((((((.....)))))))...))))))))).)))).......((((((......)))))-).))) ( -33.00, z-score = -2.51, R) >droAna3.scaffold_12894 1077 133 - 7079 ---UUCUAUUUUUUGAACAUAUAUGA--AAAAAUACCCU-GAAAUCGGUUAAUGUUUAUUGAAAAGGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGU ---...((((((((.(.......).)--)))))))....-...(((((((((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......)))))-))))) ( -29.09, z-score = -1.75, R) >dp4.chr4_group3 8083772 130 + 11692001 ------GUUUUCGUUUUCCUAUAUGAGAAAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC ------.(((((((........)))))))........---....((((((((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......)))))-)))). ( -30.39, z-score = -2.27, R) >droPer1.super_1 9534593 128 + 10282868 ------GUUUUCGUUUUCCUAUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCGGC ------.(((((((........))))--)))......---....((((((((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......)))))-)))). ( -30.29, z-score = -2.57, R) >droWil1.scaffold_180772 4495618 136 + 8906247 -CUUUCCAUUUCUAUU-CGGUUAUGA--AAAAAUACCGAAAAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCAACCGGC -.............((-((((.((..--....))))))))....((((((((((........(((((((((...(((((((.....)))))))...)))))))))........)))))(((((......)))))))))). ( -33.69, z-score = -3.32, R) >droVir3.scaffold_12963 7082552 132 - 20206255 -UACUGUUGUUGUUCGUCUUUUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAGGCAUAAAAUUCGUUCUACUAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CUGG- -....(((((...((.((....((((--(.....(((---......))).....))))).))(((((((((...(((((((.....)))))))...))))))))))).)))))....((((((......)))))-)...- ( -30.60, z-score = -2.45, R) >droMoj3.scaffold_6500 6983428 132 + 32352404 -UGUUGUUCUUGUUGGUCUUUUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAGGCAUAAAAUUCGUUCUACUAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCA-CUGC- -.(((((((...(((((.((((....--.)))).)))---))..(((((........)))))(((((((((...(((((((.....)))))))...))))))))))).)))))....((((((......)))))-)...- ( -32.20, z-score = -2.94, R) >droGri2.scaffold_14978 576383 131 + 1124632 --GUUAUUGUUGUUGGUCUUUUAUGA--AAAAAUACC---AAAAUCGGUUAAUGUUUAUUGAAAAGGCAUAAAAUUCGUUCUACUAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG-CCAG- --(((((((...(((((.((((....--.)))).)))---))...))).))))....((((.(((((((((...(((((((.....)))))))...)))))))))....))))....((((((......)))))-)...- ( -30.20, z-score = -2.12, R) >consensus ___UUUUUUUUCUUGUUCGUAUAUGA__AAAAAUACC___AAAAUCGGUUAAUGUUUAUUGAAAAAGCAUAAAAUUCGUUCUACCAGAAUGGAAAAUAUGCUUUUGAUACAAUUAUUGUGCCUAUCUGCAGGCG_CCGGC ............................................((((.(((((........(((((((((...(((((((.....)))))))...)))))))))........))))).((((......))))..)))). (-24.07 = -23.87 + -0.19)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:44:56 2011