
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 16,170,874 – 16,170,988 |
| Length | 114 |
| Max. P | 0.801735 |

| Location | 16,170,874 – 16,170,988 |
|---|---|
| Length | 114 |
| Sequences | 9 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.09 |
| Shannon entropy | 0.48069 |
| G+C content | 0.56430 |
| Mean single sequence MFE | -45.05 |
| Consensus MFE | -17.83 |
| Energy contribution | -17.01 |
| Covariance contribution | -0.82 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.40 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.801735 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
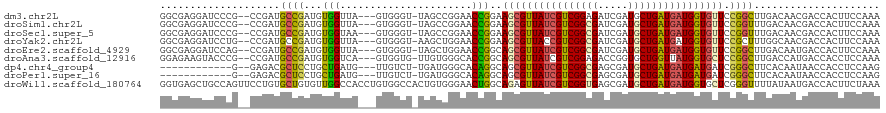
>dm3.chr2L 16170874 114 - 23011544 GGCGAGGAUCCCG--CCGAUGCCGAUGUGGUUA---GUGGGU-UAGCCGGAACCGGAAGCGUUAUCGUCGGAGAUCGAUGCUGAUGAUGGUGUUCCGGCUUGACAACGACCACUUCCAAA ((((.(....)))--))......((.((((((.---((..((-((((((((((((....))((((((((((.........)))))))))).)))))))).)))).)))))))).)).... ( -46.10, z-score = -2.25, R) >droSim1.chr2L 15879588 114 - 22036055 GGCGAGGAUCCCG--CCGAUGCCGAUGUGGUUA---GUGGGU-UAGCCGGAACCGGAAGCGUUAUCGUCGGCGAUCGAUGCUGAUGAUGGUGUUCCGGUUUGACAACGACCACUUCCAAA ((((.(....)))--))......((.((((((.---.((((.-...)).(((((((((.((..((((((((((.....))))))))))..)))))))))))..))..)))))).)).... ( -50.00, z-score = -3.12, R) >droSec1.super_5 4892403 114 - 5866729 GGCGAGGAUCCCG--CCGAUGCCGAUGUGGUAA---GUGGGU-UAGCCGGAACCGGAAGCGUUAUCGUCGGCGAUCGAUGCUGAUGAUGGUGUUCCGGUUUGACAACGACCACUUCCAAA ((((.(....)))--))......((.(((((..---.((((.-...)).(((((((((.((..((((((((((.....))))))))))..)))))))))))..))...))))).)).... ( -49.00, z-score = -2.89, R) >droYak2.chr2L 3159154 114 + 22324452 GGCGAGGAUCCUG--CCGAUGCCGAUGUGGUUA---GUGGGU-AAGCUGGAACCGGAAGCGUUACCGUCGGCGAUCGAUGCUGAUGAUGGUGUUCCGCUUUGGCAACGACCACUUCCAAA ((((.((......--))..))))....(((..(---((((..-..((..((..(((((.((..(.((((((((.....)))))))).)..))))))).))..)).....))))).))).. ( -40.80, z-score = -0.82, R) >droEre2.scaffold_4929 7867095 114 - 26641161 GGCGAGGAUCCAG--CCGAUGCCGAUGUGGUUA---GUGGGU-UAGCUGGAACCGGCAGCGUUAUCGUCGGCGAUCGAUGCUGAUGAUGGUGUUCCGGCUUGACAAUGACCACUUCCAAA ((((.((......--))..))))((.(((((((---....((-((((((((((((.(((((((((((....)))).)))))))....))))..)))))).))))..))))))).)).... ( -48.30, z-score = -2.82, R) >droAna3.scaffold_12916 15957324 114 + 16180835 GGAGAAGUACCCG--CCGAUGCCGAUGUGGUCA---GUGGUG-UUGUGGGCACCGGCAGCGUUAUCGUCGGAGACCGGUGCUGGUUAUGGUGCUCCGGCUUGACCAUGACCACCUCCAAA ((((.((((((..--((((((..(((((.(((.---..((((-(.....)))))))).)))))..)))))).....))))))(((((((((((....))...)))))))))..))))... ( -49.80, z-score = -2.05, R) >dp4.chr4_group4 5117308 102 - 6586962 ------------G--GAGACGCUCCUGCUGAUG---UUGUCU-UGAUGGGCACAGGCAGCGUUAUCGUCGGCGAGCGAUGCUGAUGAUGAUGAUCGGGCUUCACAAUAACCACCUCCAAG ------------(--(((..(((((((((..((---(.((((-....))))))))))))((((((((((((((.....))))))))))))))...))))..............))))... ( -35.09, z-score = -1.00, R) >droPer1.super_16 126438 102 + 1990440 ------------G--GAGACGCUCCUGCUGAUG---UUGUCU-UGAUGGGCACAGGCAGCGUUAUCGUCGGCGAGCGAUGCUGAUGAUGAUGAUCGGGCUUCACAAUAACCACCUCCAAG ------------(--(((..(((((((((..((---(.((((-....))))))))))))((((((((((((((.....))))))))))))))...))))..............))))... ( -35.09, z-score = -1.00, R) >droWil1.scaffold_180764 1631697 120 + 3949147 GGUGAGCUGCCAGUUCCUGUGCUGUGUUGGCCACCUGUGGCCACUGUGGGAACUGGCAGAGUUAUCGUCGGUGAGCGAUGCUGAUGAUGGUGCUCGGGUUUUAUAAUGACCACUUCUAAA ..((((((((((((((((..((...((.(((((....))))))).)))))))))))))...(((((((((((.......))))))))))).)))))((((.......))))......... ( -51.30, z-score = -3.74, R) >consensus GGCGAGGAUCCCG__CCGAUGCCGAUGUGGUUA___GUGGGU_UAGCGGGAACCGGCAGCGUUAUCGUCGGCGAUCGAUGCUGAUGAUGGUGUUCCGGCUUGACAAUGACCACUUCCAAA ....................((((...(((......................)))..((((((((((((((((.....)))))))))))))))).))))..................... (-17.83 = -17.01 + -0.82)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:44:24 2011