
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 15,467,997 – 15,468,150 |
| Length | 153 |
| Max. P | 0.636025 |

| Location | 15,467,997 – 15,468,150 |
|---|---|
| Length | 153 |
| Sequences | 9 |
| Columns | 165 |
| Reading direction | forward |
| Mean pairwise identity | 95.29 |
| Shannon entropy | 0.09783 |
| G+C content | 0.44197 |
| Mean single sequence MFE | -42.26 |
| Consensus MFE | -35.87 |
| Energy contribution | -36.81 |
| Covariance contribution | 0.94 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.636025 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
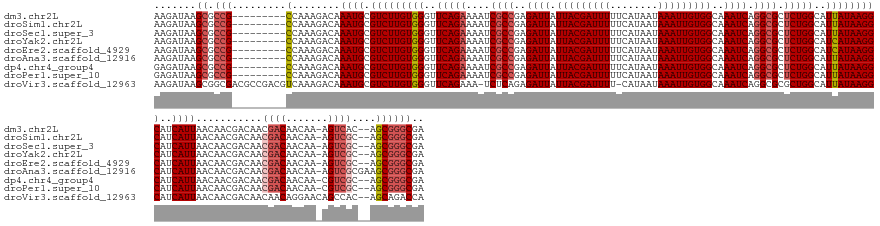
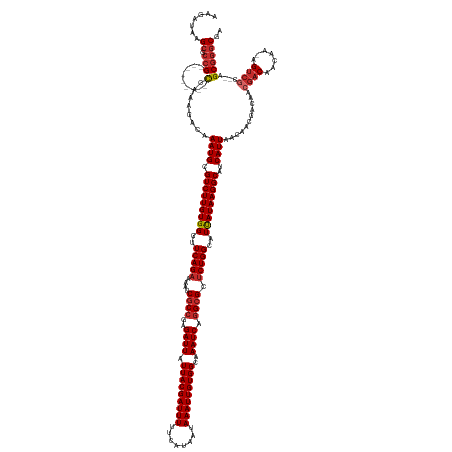
>dm3.chr2L 15467997 153 + 23011544 AAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA-AGUCAC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))............(((.....-.)))..--.)))))).. ( -41.60, z-score = -2.11, R) >droSim1.chr2L 15220770 153 + 22036055 AAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA-AGUCGC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.)))).--.)))))).. ( -42.90, z-score = -1.95, R) >droSec1.super_3 1720390 153 + 7220098 AAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA-AGUCGC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.)))).--.)))))).. ( -42.90, z-score = -1.95, R) >droYak2.chr2L 2459777 153 - 22324452 AAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUCAUAAGGCAUCAUUAACAACGACAACGACAACAA-AGUCGC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.)))).--.)))))).. ( -45.00, z-score = -2.50, R) >droEre2.scaffold_4929 7214047 153 + 26641161 AAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUCAUAAGGCAUCAUUAACAACGACAACGACAACAA-AGUCGC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.)))).--.)))))).. ( -45.00, z-score = -2.50, R) >droAna3.scaffold_12916 7772516 155 + 16180835 AAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA-AGUCGCGAAGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.))))....)))))).. ( -42.90, z-score = -1.71, R) >dp4.chr4_group4 4240510 153 - 6586962 GAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA-CGUCGC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.)))).--.)))))).. ( -42.90, z-score = -1.66, R) >droPer1.super_10 2078087 153 - 3432795 GAGAUAAGCGCCG---------CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA-CGUCGC--AGCGGGCGA .......((.(((---------(........((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..))))...........((((.....-.)))).--.)))))).. ( -42.90, z-score = -1.66, R) >droVir3.scaffold_12963 12143004 161 + 20206255 AAGAUAAGCGGCGACGCCGACGUCAAAGACAAAUGCGUCUUGUGGGUUCAGAAA-UCUCAGAGAUUAUUACGAUUUU-CAUAAUAAAUUGUGGCAAAUCAGGCGCGCUGGCAUUAUAAGGCAUCAUUAACAACGACAACAACAGGAACAGCCAC--AGCAGACCA .......(((....)))....(((..........(((((((.((((........-.)))).)((((.(((((((((.-......)))))))))..))))))))))((((((........))......................((.....)).)--))).))).. ( -34.20, z-score = 0.12, R) >consensus AAGAUAAGCGCCG_________CCAAAGACAAAUGCGUCUUGUGGGUUCAGAAAAUCGCCGAGAUUAUUACGAUUUUUCAUAAUAAAUUGUGGCAAAUCAGGCGCUCUGGCAUUAUAAGGCAUCAUUAACAACGACAACGACAACAA_AGUCGC__AGCGGGCGA ........((((...................((((.(((((((((..(((((....((((..((((.(((((((((........)))))))))..)))).)))).)))))..)))))))))..)))).....((....((((.......)))).....)))))). (-35.87 = -36.81 + 0.94)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:42:53 2011