| Sequence ID | dm3.chr2L |
|---|---|
| Location | 14,894,426 – 14,894,546 |
| Length | 120 |
| Max. P | 0.913630 |

| Location | 14,894,426 – 14,894,546 |
|---|---|
| Length | 120 |
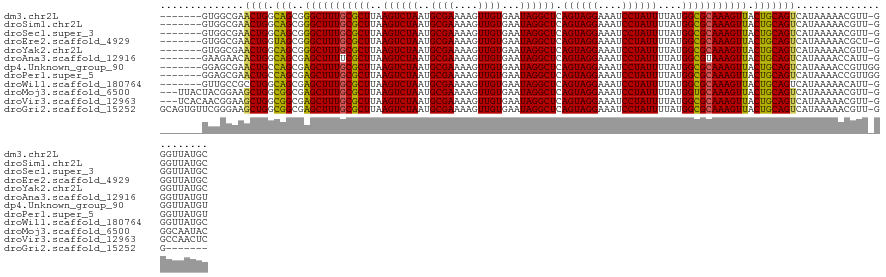
| Sequences | 12 |
| Columns | 128 |
| Reading direction | reverse |
| Mean pairwise identity | 90.34 |
| Shannon entropy | 0.21505 |
| G+C content | 0.44347 |
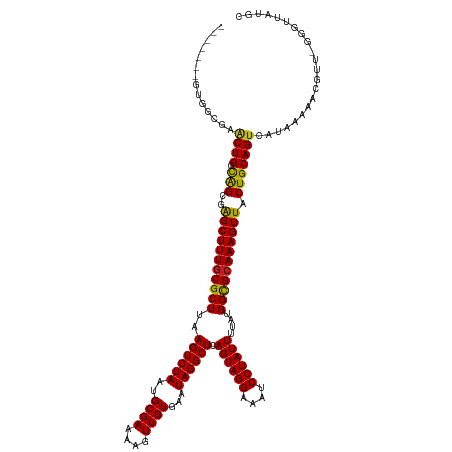
| Mean single sequence MFE | -36.73 |
| Consensus MFE | -33.44 |
| Energy contribution | -32.86 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.91 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.23 |
| SVM RNA-class probability | 0.913630 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 14894426 120 - 23011544 -------GUGGCGAACUGGCAGCGGGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU-GGGUUAUGC -------(((((......((((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))).))))).........-......... ( -36.60, z-score = -1.56, R) >droSim1.chr2L 14641575 120 - 22036055 -------GUGGCGAACUGGCAGCGGGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU-GGGUUAUGC -------(((((......((((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))).))))).........-......... ( -36.60, z-score = -1.56, R) >droSec1.super_3 1164183 120 - 7220098 -------GUGGCGAACUGGCAGCGGGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU-GGGUUAUGC -------(((((......((((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))).))))).........-......... ( -36.60, z-score = -1.56, R) >droEre2.scaffold_4929 6637772 120 - 26641161 -------GUGGCGAACUGGUAGCGGGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGCU-GGGUUAUGC -------..((((...(((..((((((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))..))))..)))......))))-......... ( -36.00, z-score = -1.35, R) >droYak2.chr2L 14997157 120 - 22324452 -------GUGGCGAACUGGCAGCGGGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU-GGGUUAUGC -------(((((......((((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))).))))).........-......... ( -36.60, z-score = -1.56, R) >droAna3.scaffold_12916 15225860 120 + 16180835 -------GAAGAACACUGGCAGCGAGCUUUUCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGUAAAGUUACUGCAGUCAUAAAACCAUU-GGGUUAUGU -------........(((...((.((((((((((...........))))))))))))...)))((((((((((....))).((((((((((((.......))).)))))))))..)))-))))..... ( -32.60, z-score = -1.65, R) >dp4.Unknown_group_90 3460 121 - 23183 -------GGAGCGAACUGCCAGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAACCGUUGGGGUUAUGU -------.......(((((.((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))))))(((((..((...))..))))). ( -38.80, z-score = -2.06, R) >droPer1.super_5 2425132 121 - 6813705 -------GGAGCGAACUGCCAGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAACCGUUGGGGUUAUGU -------.......(((((.((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))))))(((((..((...))..))))). ( -38.80, z-score = -2.06, R) >droWil1.scaffold_180764 556761 120 + 3949147 -------GUUGCCGCCUGGCAGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACAUU-GGGUUAUGC -------((((((....)))))).((((((.(((...........))).)))))).......(((((((((((....))).((((((((((((.......))).)))))))))..)))-))))).... ( -36.70, z-score = -1.37, R) >droMoj3.scaffold_6500 23686663 124 + 32352404 ---UUACUACGGAAGCUGGCGGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGUGCAAAGUUACUGCAGUCAUAAAAACGUU-GGGCAAUAC ---...(((((.....((((.((.(((((((((((..((((((..((((....))))...)))))).((((((....))))))....)))))))))))...)).))))......)).)-))....... ( -34.20, z-score = -1.02, R) >droVir3.scaffold_12963 15458948 124 + 20206255 ---UCACAACGGAAGCUGGCGGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU-GGCCAACUC ---...(((((.....((((.((.(((((((((((..((((((..((((....))))...)))))).((((((....))))))....)))))))))))...)).))))......))))-)........ ( -39.80, z-score = -2.32, R) >droGri2.scaffold_15252 685283 120 - 17193109 GCAGUGUUCGGGAAGCUGGCGGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU-GG------- .((((((((((....)))((((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))).........))))))-).------- ( -37.40, z-score = -1.53, R) >consensus _______GUGGCGAACUGGCAGCGAGCUUUGCGCUUAAGUCUAAUGCGAAAAGUUGUGAAUAGGCUCAGUAGGAAAUCCUAUUUUAUGGCGCAAAGUUACUGCAGUCAUAAAAACGUU_GGGUUAUGC ..............((((.(((..(((((((((((..((((((..((((....))))...)))))).((((((....))))))....))))))))))).)))))))...................... (-33.44 = -32.86 + -0.58)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:41:44 2011