| Sequence ID | dm3.chr2L |
|---|---|
| Location | 1,286,362 – 1,286,480 |
| Length | 118 |
| Max. P | 0.744803 |

| Location | 1,286,362 – 1,286,480 |
|---|---|
| Length | 118 |
| Sequences | 14 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 69.16 |
| Shannon entropy | 0.71660 |
| G+C content | 0.62893 |
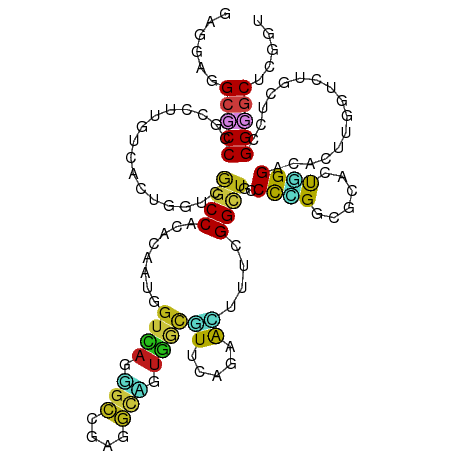
| Mean single sequence MFE | -52.41 |
| Consensus MFE | -20.01 |
| Energy contribution | -19.41 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 2.14 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.744803 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 1286362 118 - 23011544 GACGAUGCUCCCGCCUUGUCACUGGUUGGCCAAACAAUGGUCAGUGCCGAGGCAGUGGCGUUCAGAAUUUUCGGUUGCCCUGGAGCACUGGGACACUUAGUCUGCUCCGGGGCUCUGU ...(((((..(.((((((.(((((....((((.....))))))))).)))))).)..))))).........(((..((((((((((((((((...))))))..)))))))))).))). ( -53.30, z-score = -2.84, R) >droSim1.chr2L 1258869 118 - 22036055 GACGAUGCACCCGCCUUGUCACUGGUGGGCCAAACAAUGGUCAGUGCCGAGGCAGUGGCGUUCAGAAUUUUCGGUUGGCCCGGGGCACUGGGACACUUAGUCUGCUCCGGGGCUCUGU ...(((((..(.((((((.(((((....((((.....))))))))).)))))).)..))))).........(((..(.((((((((((((((...))))))..)))))))).).))). ( -48.20, z-score = -0.65, R) >droSec1.super_14 1236708 118 - 2068291 GACGAUGCUCCCGCCUUGUCACUGGUGGGCCAAACAAUGGUCAGUGCCGAGGCAGUGGCGUUAAGAAUUUUCGGUUGUCCCGGGGCACUGGGACACUUAGUCUGCUCCGGGGCUCUGU ...(((((..(.((((((.(((((....((((.....))))))))).)))))).)..))))).........(((..((((((((((((((((...))))))..)))))))))).))). ( -52.50, z-score = -2.37, R) >droYak2.chr2L 1262230 118 - 22324452 GAGGAGGCGCCCGCCUUGUCACUGGUGGGCCACACAAUGGUCAGUGCCGAGGCAGUGGCGUUCAGCACCUUGGGCUGUCCCGGUGCACUGGGACACUUGGUCUGCUCCGGGGCUCUGU ((((..(((((((((........))))))).........((((.(((....))).)))).....)).))))((((((((((((....))))))))(((((......)))))))))... ( -55.40, z-score = -0.51, R) >droEre2.scaffold_4929 1326247 118 - 26641161 GACGAUGCACCCGCCUUGUCGCUGGUGGGCCAGACGAUGGUCAGUGCCGAGGCAGUGGCGUUCAGAACUUUCGGCUGACCCGGGGCACUGGGACACUUGGUCUGCUCCGGCGCCCUGU (((((.((....)).)))))...(((.((((....((..((((.(((....))).))))..)).((....)))))).)))((((((.((((..((.......))..)))).)))))). ( -50.60, z-score = -0.02, R) >droAna3.scaffold_12916 4748651 118 - 16180835 GACGAAGCUCCUGCCUUGUCACUCGUCGGCCAGACAAUUGUCAGGGCCGAGGAUGUGGCAUUCAGGACCUUGGGCUGCCCGGGCGCCGUGGGGCAUUUACUCUGUUCCGGGGCUCUGU ..(.(((.(((((...((((((((.((((((.(((....)))..)))))).)).))))))..))))).))).)......((((.(((.(((((((.......))))))).))))))). ( -56.00, z-score = -2.27, R) >dp4.chr4_group3 9765545 118 - 11692001 GAGGAGGCCCCCGCCUUGUCGCUGGUCGGCCACACGAUCGUCAGGGCCGAGGCCGUGGCGUUCAGGACCUUCGGCUGGCCGGGCGCCGUCGGGCACUUGCUCUGCUCCGGGGCCCGGU ((.(((((....))))).))(((((((((((....((.(((((.(((....))).))))).))..(.....)))))))))((((.(((..(((((.......)))))))).))))))) ( -61.90, z-score = -0.12, R) >droPer1.super_8 965743 118 - 3966273 GAGGAGGCCCCCGCCUUGUCGCUGGUCGGCCACACGAUCGUCAGGGCCGAGGCCGUGGCGUUCAGGACCUUCGGCUGGCCGGGCGCCGUCGGGCACUUGCUCUGCUCCGGGGCCCGGU ((.(((((....))))).))(((((((((((....((.(((((.(((....))).))))).))..(.....)))))))))((((.(((..(((((.......)))))))).))))))) ( -61.90, z-score = -0.12, R) >droWil1.scaffold_180708 2912540 118 - 12563649 GAUGAGGCUCCAGCCUUAUCACUUGUAGGCCACACUAUGGUCAGGGCAGAGGCUGUGGCAUUCAGUAUCUUGGGUUGUCCUGGAGCUGUAGGACAUUUACUUUGUUCGGGAGCUCGAU ((((((((....))))))))..(..(((......)))..).((((((((..((((.......))))........))))))))(((((.(.(((((.......))))).).)))))... ( -42.00, z-score = -1.04, R) >droVir3.scaffold_12963 7221168 118 - 20206255 GGGGAGGCGCCCAGCUUGUCGCUGGUGGGCCACACAAUUGUCAUGGCCGUGGCGGUGGCAUUCAGCACUGUCGGCUGCCCCGGUGCCGUGGGGCAUUUGCUUUGCUCGGGCGCACGAU ((((.(((.((((((.....))))).).))).......((((((.((....)).))))))..((((.......)))))))).((((((..((((....))))..)...)))))..... ( -54.80, z-score = -0.09, R) >droMoj3.scaffold_6500 3519349 118 - 32352404 CUGGAGGCGCCCACCUUGUUGCUGCGCGUCCAGCUGAGGGUGACCGAAUCGUCGGUCUUCUCCACCAGCUGCGGCUUGCCGGGCGGACCCGGAUAGGUGGAUAGCUCGGGGGCGCGAU ......((((((.((..(((((..(..(((((((((..((.((((((....))))))....))..)))))).)))...(((((....)))))....)..).))))..))))))))... ( -59.80, z-score = -1.73, R) >droGri2.scaffold_15252 10360927 118 - 17193109 GGCGAUGAGCCCAACUUGUCACUGGUAGGCCACACAAUUGUCAUUGCCGCUGCAGUGGCAUUCAGCACUGUCGGCUGCCCCGGCGCCGUGGGGCAUUUACUUUGCUCGGGUGCACGAU ((((((((...(((.((((...(((....))).)))))))))))))))(((((((((........))))).)))).....(((((((...(((((.......))))).))))).)).. ( -44.20, z-score = 0.25, R) >anoGam1.chr3R 1121988 118 + 53272125 UCGGAAGCACCGGACUUGAUGCUGGGCAGCCAAGAGAUGGUAAUGGAGCUGUUGCUAAUGUUAACAAUUUGGGGCUUUCCCGGGGCACUUGGGAAUUCGGAUGGUUCAGGUGCCCGAU ((((..((((((((((.....(((((.((((..(((.((.((((..(((....)))...)))).)).)))..))))((((((((...)))))))))))))..))))).))))))))). ( -42.00, z-score = -1.15, R) >triCas2.ChLG8 14868486 118 - 15773733 CUGGACGACCCGAUUUUGUUAUUCUGACCCCACGUGAUCGUGACACUGCCCUCCGAUUGGUUGAGGGCGUGUGGACGACUUGGAGGCCCCGGAAGCAUGAUAGCUUCCGGGGCUUUGA .........((((..(((((..........((((....)))).((((((((((.........))))))).)))))))).))))(((((((((((((......)))))))))))))... ( -51.20, z-score = -3.99, R) >consensus GAGGAGGCGCCCGCCUUGUCACUGGUCGGCCACACAAUGGUCAGGGCCGAGGCAGUGGCGUUCAGAACUUUCGGCUGCCCCGGCGCACUGGGACACUUGGUCUGCUCCGGGGCUCGGU ......(((((................((((........((((.(((....))).))))((.....))....))))..((((......))))................)))))..... (-20.01 = -19.41 + -0.61)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:08:12 2011