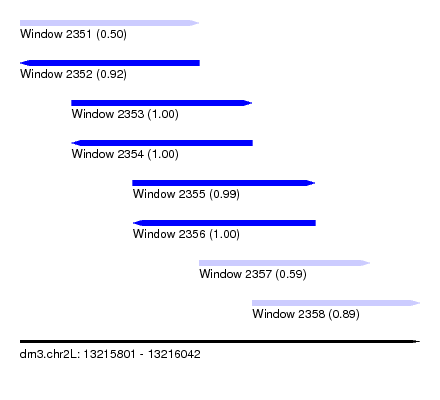
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 13,215,801 – 13,216,042 |
| Length | 241 |
| Max. P | 0.999512 |

| Location | 13,215,801 – 13,215,909 |
|---|---|
| Length | 108 |
| Sequences | 14 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 78.28 |
| Shannon entropy | 0.53132 |
| G+C content | 0.39541 |
| Mean single sequence MFE | -23.92 |
| Consensus MFE | -13.12 |
| Energy contribution | -13.36 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.24 |
| Mean z-score | -0.90 |
| Structure conservation index | 0.55 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215801 108 + 23011544 UGGUCAUGGAGAGCCUGUUCGUUUUCCGUUUCAA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC (((..(((((((((......))))))))).)))(---(.((((((((((..(((....)))..)))....))))..))).))....(((((((........)))))))... ( -25.50, z-score = -1.39, R) >droSim1.chr2L_random 496014 108 + 909653 UGCUCAUGGAGACCCUGUUCGUUUUCCGUUUCAA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC ((((((((((((.(......).))))).......---.........(((..(((....)))..))).....))).)))).......(((((((........)))))))... ( -21.10, z-score = -0.26, R) >droSec1.super_16 1370086 108 + 1878335 UGCUCACCGAGACCCUGUUCGUUUUUCGUUUCAA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC ((((((((((((..(.....)..)))))......---.........(((..(((....)))..))).....))).)))).......(((((((........)))))))... ( -21.50, z-score = -0.75, R) >droYak2.chr2L 9643563 107 + 22324452 UGCUCAUGGAGACCCUGCUUGUUUU-CGUUUAAA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC (((((((((((((.......)))))-).......---.........(((..(((....)))..))).....))).)))).......(((((((........)))))))... ( -20.90, z-score = 0.03, R) >droEre2.scaffold_4929 14417772 108 - 26641161 UGCUCAUGGAGACCCUGUUCGUUUUCCCUUACAA---UUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC ((((((((((((.(......).))))).......---.........(((..(((....)))..))).....))).)))).......(((((((........)))))))... ( -20.20, z-score = -0.30, R) >droAna3.scaffold_13340 22987596 107 + 23697760 AGAGUAUUGAGUGGUUACU-UUCUGCCAGUCUCA---GGUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC ((((....((.((((....-....)))).)).((---((((((((((((..(((....)))..)))....))))..)))))))))))((((((........)))))).... ( -29.20, z-score = -1.90, R) >dp4.chr4_group4 4123363 108 + 6586962 UGAUUAUGGUGUGGUGGCUGUUUUGCUGUUUCCA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUU ..((((.(((((...(((((....((((....))---))...(((((((..(((....)))..)))....))))................)))))..)))))))))..... ( -26.10, z-score = -1.03, R) >droPer1.super_10 264002 108 - 3432795 UGCCUAUGGUGUGGCGGCUGUUUUGCUGUUUCCA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUU .(((........)))((.((((..((((....((---(.((((((((((..(((....)))..)))....))))..))).))).......))))..)))).))........ ( -28.10, z-score = -1.18, R) >droWil1.scaffold_180708 407133 108 - 12563649 CUAUCAUGGAGAGGCAAGCUGUUGGCUGUUUCAA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUU ((((((.((.((((((.((.....))))))))..---((((..((((((..(((....)))..)))...(((..(((...)))..)))...))).)))).))))))))... ( -27.30, z-score = -0.79, R) >droVir3.scaffold_12723 2221729 108 - 5802038 CGGUGGUGUACUAUUACGAGAUUACCAUUGUAAA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUU .(((((((.(((.((((((........)))))))---)))))))))(((..(((....)))..)))....................(((((((........)))))))... ( -25.00, z-score = -1.06, R) >droMoj3.scaffold_6500 15233883 108 + 32352404 AAAUGGUGUAGUGCUGUGAGUUUAGCAAUGUAGA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUU ....(((((...((((((((..((....))((((---.....(((((((..(((....)))..)))....)))).....))))..)))).))))...)))))......... ( -25.70, z-score = -0.76, R) >droGri2.scaffold_15252 4051861 107 + 17193109 UGCUGUUAUAGUGUUA-GAGUUUUCGUGAGAUUA---GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUU .(((((((..(((((.-(((((...((((((.((---(.((((((((((..(((....)))..)))....))))..))).))).)))))).)))))))))).))))))).. ( -29.40, z-score = -2.53, R) >apiMel3.Group1 9104400 109 + 25854376 UGUUUACGAGGAAAGUUUCAGUUUAACGACACUACACGUAACUAUAUAUAUAUCUUACAGCUAAGAUCAACGUGUAAUAUUCGCUAUUACGAAGUUAAUAUCUGAUAGG-- .................((((.(((((.....(((((((............((((((....))))))..)))))))...((((......)))))))))...))))....-- ( -17.44, z-score = 0.34, R) >anoGam1.chr3R 11138237 90 + 53272125 ----------------GUUUGUUGUUC--UUCAA---GUUAUCGCUUCUCGGCCUAAAGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUC ----------------...........--....(---((((((...(((..(((....)))..))).....((((.....(((.......)))....))))..))))))). ( -17.50, z-score = -1.03, R) >consensus UGCUCAUGGAGAGCCUGUUGGUUUUCCGUUUCAA___GUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUC ..........................................(((((((..(((....)))..)))....))))............(((((((........)))))))... (-13.12 = -13.36 + 0.24)

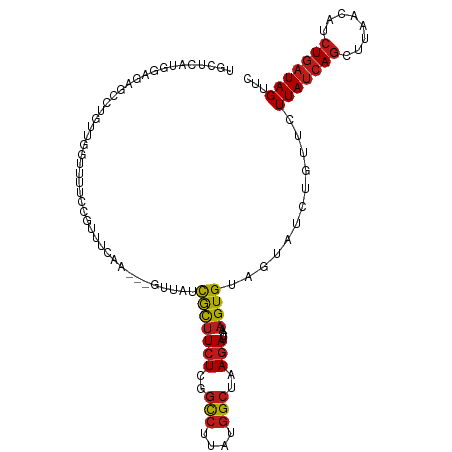
| Location | 13,215,801 – 13,215,909 |
|---|---|
| Length | 108 |
| Sequences | 14 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 78.28 |
| Shannon entropy | 0.53132 |
| G+C content | 0.39541 |
| Mean single sequence MFE | -20.44 |
| Consensus MFE | -12.56 |
| Energy contribution | -12.59 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.61 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.25 |
| SVM RNA-class probability | 0.915965 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215801 108 - 23011544 GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UUGAAACGGAAAACGAACAGGCUCUCCAUGACCA ......((((..((((.(((........((((........)))).(((..(((....)))..))))))..))))---))))..((.....))....((.((.....)))). ( -18.50, z-score = -0.57, R) >droSim1.chr2L_random 496014 108 - 909653 GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UUGAAACGGAAAACGAACAGGGUCUCCAUGAGCA ......((((((((....(((.......))).....))).)))))(((..(((....)))..))).((.....(---(((...((.....))..)))).((.....)))). ( -20.00, z-score = -0.43, R) >droSec1.super_16 1370086 108 - 1878335 GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UUGAAACGAAAAACGAACAGGGUCUCGGUGAGCA ....((((((........)))))).............(((.....(((..(((....)))..)))..(((...(---(((...((.....))..))))...)))))).... ( -21.30, z-score = -0.60, R) >droYak2.chr2L 9643563 107 - 22324452 GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UUUAAACG-AAAACAAGCAGGGUCUCCAUGAGCA ......((((........))))(((((.((((........)))).)))))(((((.(((((..((((......)---)))....(-........)..)))))..))).)). ( -17.10, z-score = 0.39, R) >droEre2.scaffold_4929 14417772 108 + 26641161 GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAA---UUGUAAGGGAAAACGAACAGGGUCUCCAUGAGCA ....((((((........))))))........(((......(((.(((..(((....)))..)))..)))....---..))).((..(..(.....)..)..))....... ( -19.50, z-score = 0.13, R) >droAna3.scaffold_13340 22987596 107 - 23697760 GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUACC---UGAGACUGGCAGAA-AGUAACCACUCAAUACUCU ......((((.((((..(((........((((........)))).(((..(((....)))..)))))))))).)---)))((.(((.....-.....))).))........ ( -19.40, z-score = -0.51, R) >dp4.chr4_group4 4123363 108 - 6586962 AAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UGGAAACAGCAAAACAGCCACCACACCAUAAUCA ....((((((........))))))......(((............(((..(((....)))..))).((.....(---((....))).......))............))). ( -20.80, z-score = -2.56, R) >droPer1.super_10 264002 108 + 3432795 AAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UGGAAACAGCAAAACAGCCGCCACACCAUAGGCA ......((((((((....(((.......))).....))).)))))(((..(((....)))..))).((.....(---((....))).......)).(((........))). ( -25.00, z-score = -2.82, R) >droWil1.scaffold_180708 407133 108 + 12563649 AAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UUGAAACAGCCAACAGCUUGCCUCUCCAUGAUAG ...((((((...(.(((((((.(((((.((((........)))).)))))(((....)))((((.........)---)))..........))))))).)......)))))) ( -23.20, z-score = -2.02, R) >droVir3.scaffold_12723 2221729 108 + 5802038 AAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UUUACAAUGGUAAUCUCGUAAUAGUACACCACCG ....((((((........))))))........(((((........(((..(((....)))..))).((((....---.((((....))))..))))..)))))........ ( -20.20, z-score = -1.29, R) >droMoj3.scaffold_6500 15233883 108 - 32352404 AAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UCUACAUUGCUAAACUCACAGCACUACACCAUUU .........(.(((...((((.(((((.((((........)))).)))))(((....))).(((.((((((...---.....))))))...))).))))...))).).... ( -22.90, z-score = -3.09, R) >droGri2.scaffold_15252 4051861 107 - 17193109 AAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC---UAAUCUCACGAAAACUC-UAACACUAUAACAGCA .................((((.(((((.((((........)))).)))))(((....))).((((((......)---)..))))..........-...........)))). ( -17.50, z-score = -1.38, R) >apiMel3.Group1 9104400 109 - 25854376 --CCUAUCAGAUAUUAACUUCGUAAUAGCGAAUAUUACACGUUGAUCUUAGCUGUAAGAUAUAUAUAUAGUUACGUGUAGUGUCGUUAAACUGAAACUUUCCUCGUAAACA --....((((...(((((.((((....))))(((((((((((.......(((((((.........)))))))))))))))))).))))).))))................. ( -22.61, z-score = -1.46, R) >anoGam1.chr3R 11138237 90 - 53272125 GAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCUUUAGGCCGAGAAGCGAUAAC---UUGAA--GAACAACAAAC---------------- ..(((.((((((((....(((.......))).....))).)))))(((..(((....)))..))))))......---.....--...........---------------- ( -18.10, z-score = -2.04, R) >consensus GAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAC___UUGAAACGGAAAACCAACAGGCUCUCCAUGAGCA ......((((((((....(((.......))).....))).)))))(((..(((....)))..))).............................................. (-12.56 = -12.59 + 0.03)

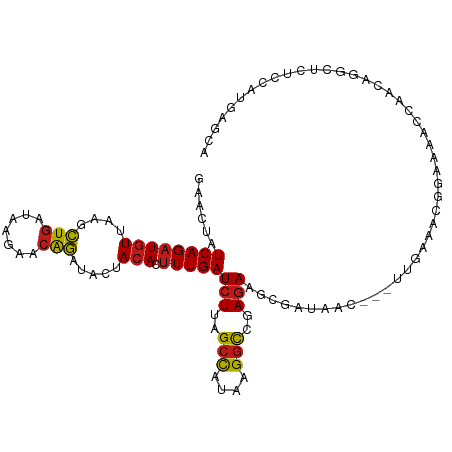
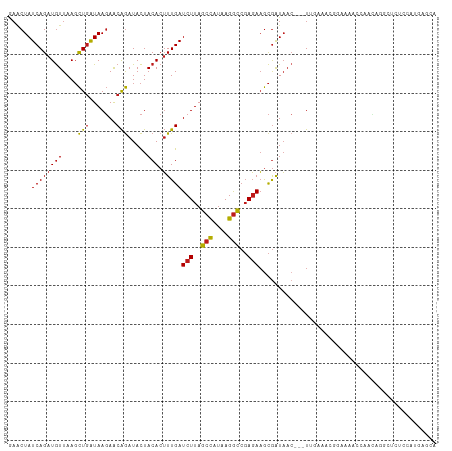
| Location | 13,215,832 – 13,215,941 |
|---|---|
| Length | 109 |
| Sequences | 14 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 90.31 |
| Shannon entropy | 0.23991 |
| G+C content | 0.37272 |
| Mean single sequence MFE | -29.30 |
| Consensus MFE | -25.51 |
| Energy contribution | -25.11 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.96 |
| Structure conservation index | 0.87 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.85 |
| SVM RNA-class probability | 0.995857 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215832 109 + 23011544 ---CAAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ---..((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))) ( -30.20, z-score = -3.05, R) >droSim1.chr2L_random 496045 109 + 909653 ---CAAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ---..((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))) ( -30.20, z-score = -3.05, R) >droSec1.super_16 1370117 109 + 1878335 ---CAAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ---..((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))) ( -30.20, z-score = -3.05, R) >droYak2.chr2L 9643594 108 + 22324452 ----AAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ----.((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))) ( -30.20, z-score = -3.12, R) >droEre2.scaffold_4929 14417803 109 - 26641161 ---CAAUUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ---........(((((((..(((....)))..)))....))))..............(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))) ( -29.90, z-score = -3.29, R) >droAna3.scaffold_13340 22987627 108 + 23697760 ----CAGGUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ----((((((((((((((..(((....)))..)))....))))..))))))).....(((((.(((((((.((...((.(((((...)))))))..)).))))))).))))) ( -36.30, z-score = -4.58, R) >dp4.chr4_group4 4123394 109 + 6586962 ---CCAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAU ---.(((.((((((((((..(((....)))..)))....))))..))).))).....(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))) ( -30.30, z-score = -3.47, R) >droPer1.super_10 264033 109 - 3432795 ---CCAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAU ---.(((.((((((((((..(((....)))..)))....))))..))).))).....(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))) ( -30.30, z-score = -3.47, R) >droWil1.scaffold_180708 407164 109 - 12563649 ---CAAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAU ---..((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))) ( -27.80, z-score = -2.70, R) >droVir3.scaffold_12723 2221760 109 - 5802038 ---AAAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAU ---..((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))) ( -27.80, z-score = -2.76, R) >droMoj3.scaffold_6500 15233914 109 + 32352404 ---AGAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAU ---(((.....(((((((..(((....)))..)))....)))).....)))......(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))) ( -30.10, z-score = -2.94, R) >droGri2.scaffold_15252 4051891 109 + 17193109 ---UUAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAU ---.(((.((((((((((..(((....)))..)))....))))..))).))).....(((((.(((((((.((...(((.((((...)))))))..)).))))))).))))) ( -28.60, z-score = -2.93, R) >apiMel3.Group1 9104431 109 + 25854376 CUACACGUAACUAUAUAUAUAUCUUACAGCUAAGAUCAACGUGUAAUAUUCGCUAUUACGAAGUUAAUAUCUGAUAGG--CUUUAUUGAAAUGUU-UAAGCGGGCUAUUAAC .(((((((............((((((....))))))..)))))))...((((......))))(((((((((((.((((--(...........)))-))..)))).))))))) ( -18.34, z-score = -0.10, R) >anoGam1.chr3R 11138250 109 + 53272125 ---CAAGUUAUCGCUUCUCGGCCUAAAGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUCUCCCAUAGGGAGACAACAAAUGUUAAACUGAU ---..((.((((((((((..(((....)))..)))....))))..))).))......(((((.(((((((.((...(.((((((...)))))))..)).))))))).))))) ( -29.90, z-score = -2.87, R) >consensus ___CAAGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAU ...........(((((((..(((....)))..)))....))))..............(((((.(((((((.((.....((((((...))))))...)).))))))).))))) (-25.51 = -25.11 + -0.40)

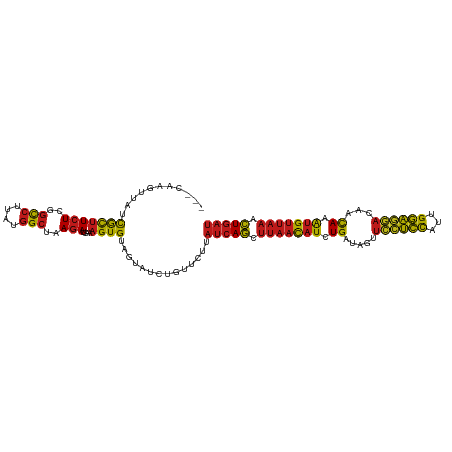
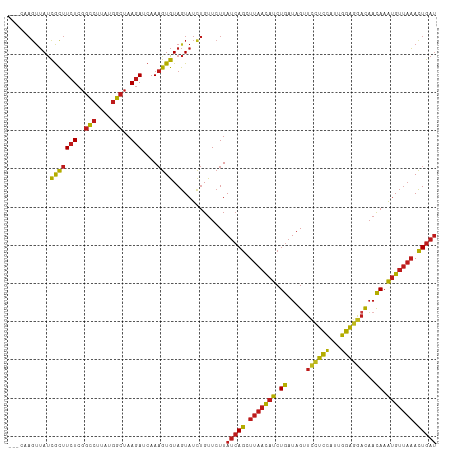
| Location | 13,215,832 – 13,215,941 |
|---|---|
| Length | 109 |
| Sequences | 14 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 90.31 |
| Shannon entropy | 0.23991 |
| G+C content | 0.37272 |
| Mean single sequence MFE | -35.80 |
| Consensus MFE | -33.29 |
| Energy contribution | -32.89 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.41 |
| Mean z-score | -5.18 |
| Structure conservation index | 0.93 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.59 |
| SVM RNA-class probability | 0.999005 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215832 109 - 23011544 AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUUG--- ((((((((((((((((...((((((...)))))).....))))))))))))))))(((..((((........)))).(((..(((....)))..)))........))).--- ( -38.40, z-score = -5.84, R) >droSim1.chr2L_random 496045 109 - 909653 AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUUG--- ((((((((((((((((...((((((...)))))).....))))))))))))))))(((..((((........)))).(((..(((....)))..)))........))).--- ( -38.40, z-score = -5.84, R) >droSec1.super_16 1370117 109 - 1878335 AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUUG--- ((((((((((((((((...((((((...)))))).....))))))))))))))))(((..((((........)))).(((..(((....)))..)))........))).--- ( -38.40, z-score = -5.84, R) >droYak2.chr2L 9643594 108 - 22324452 AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUU---- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..)))...........---- ( -38.00, z-score = -5.90, R) >droEre2.scaffold_4929 14417803 109 + 26641161 AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAAAUUG--- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..)))............--- ( -38.00, z-score = -5.90, R) >droAna3.scaffold_13340 22987627 108 - 23697760 AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUACCUG---- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..)))...........---- ( -38.00, z-score = -5.73, R) >dp4.chr4_group4 4123394 109 - 6586962 AUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUGG--- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....(((...(........).(((..(((....)))..)))........))).--- ( -36.30, z-score = -5.24, R) >droPer1.super_10 264033 109 + 3432795 AUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUGG--- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....(((...(........).(((..(((....)))..)))........))).--- ( -36.30, z-score = -5.24, R) >droWil1.scaffold_180708 407164 109 + 12563649 AUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUUG--- ((((((((((((((((...((((((...)))))).....))))))))))))))))(((..((((........)))).(((..(((....)))..)))........))).--- ( -36.00, z-score = -5.47, R) >droVir3.scaffold_12723 2221760 109 + 5802038 AUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUUU--- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..)))............--- ( -35.60, z-score = -5.42, R) >droMoj3.scaffold_6500 15233914 109 - 32352404 AUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUCU--- ((((((((((((((((...((((((...)))))).....)))))))))))))))).(((.((((........)))).(((..(((....)))..))).........)))--- ( -35.70, z-score = -5.15, R) >droGri2.scaffold_15252 4051891 109 - 17193109 AUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUAA--- ((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..)))............--- ( -35.60, z-score = -5.44, R) >apiMel3.Group1 9104431 109 - 25854376 GUUAAUAGCCCGCUUA-AACAUUUCAAUAAAG--CCUAUCAGAUAUUAACUUCGUAAUAGCGAAUAUUACACGUUGAUCUUAGCUGUAAGAUAUAUAUAUAGUUACGUGUAG (((((((....((((.-............)))--)........)))))))(((((....)))))...(((((((.......(((((((.........)))))))))))))). ( -18.13, z-score = -0.51, R) >anoGam1.chr3R 11138250 109 - 53272125 AUCAGUUUAACAUUUGUUGUCUCCCUAUGGGAGAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCUUUAGGCCGAGAAGCGAUAACUUG--- ((((((((((((((((..(((((((...)))))).)...))))))))))))))))(((..((((........)))).(((..(((....)))..)))........))).--- ( -38.30, z-score = -5.07, R) >consensus AUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACUUG___ ((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..)))............... (-33.29 = -32.89 + -0.40)
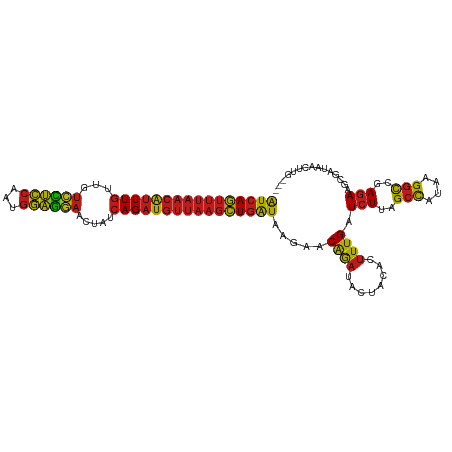
| Location | 13,215,869 – 13,215,979 |
|---|---|
| Length | 110 |
| Sequences | 14 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 88.57 |
| Shannon entropy | 0.27981 |
| G+C content | 0.40932 |
| Mean single sequence MFE | -32.52 |
| Consensus MFE | -28.10 |
| Energy contribution | -27.53 |
| Covariance contribution | -0.57 |
| Combinations/Pair | 1.49 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.26 |
| SVM RNA-class probability | 0.987065 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215869 110 + 23011544 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGAGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))--).....)))))((((....))))... ( -35.40, z-score = -3.03, R) >droSim1.chr2L_random 496082 110 + 909653 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGAGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))--).....)))))((((....))))... ( -35.40, z-score = -3.03, R) >droSec1.super_16 1370154 110 + 1878335 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGAGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))--).....)))))((((....))))... ( -35.40, z-score = -3.03, R) >droYak2.chr2L 9643630 110 + 22324452 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGAGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))--).....)))))((((....))))... ( -35.40, z-score = -3.03, R) >droEre2.scaffold_4929 14417840 110 - 26641161 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGAGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))--).....)))))((((....))))... ( -35.40, z-score = -3.03, R) >droAna3.scaffold_13340 22987663 110 + 23697760 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCUG--ACGGAGUGCUAGGAGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....))).....--))))).)))))((((....))))... ( -34.90, z-score = -2.97, R) >dp4.chr4_group4 4123431 110 + 6586962 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGGGCUUGCUCCACC ........((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))--).(((((((.....))..)))))... ( -33.00, z-score = -2.49, R) >droPer1.super_10 264070 110 - 3432795 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGGGCUUGCUCCACC ........((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))--).(((((((.....))..)))))... ( -33.00, z-score = -2.49, R) >droWil1.scaffold_180708 407201 110 - 12563649 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAACAG--ACGGAGUGCUAGGGGCUUGCUCCACC ...(((((((((((...(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))...((....)))--))))).)))))((((....))))... ( -34.20, z-score = -2.93, R) >droVir3.scaffold_12723 2221797 110 - 5802038 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAAUUG--ACGGAGUGCUAGGGGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....))).....--))))).)))))((((....))))... ( -31.60, z-score = -2.34, R) >droMoj3.scaffold_6500 15233951 110 + 32352404 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAAUAG--ACGGAGUGCUAGGGGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....))).....--))))).)))))((((....))))... ( -31.60, z-score = -2.28, R) >droGri2.scaffold_15252 4051928 110 + 17193109 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAG--ACGGAGUGCUAGGGGCUUGCUCCACC ........((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))--).(((((((.....))..)))))... ( -33.00, z-score = -2.49, R) >apiMel3.Group1 9104471 109 + 25854376 GUGUAAUAUUCGCUAUUACGAAGUUAAUAUCUGAUAG--GCUUUAUUGAAAUGUU-UAAGCGGGCUAUUAACCCUCCAAAUUAUCCACGCGAUAAUAAUCGUUCGUUGAGAA .(((((((.....)))))))..(((((((((((.(((--((...........)))-))..)))).)))))))...........((.(((((((....))))..))).))... ( -17.10, z-score = 0.84, R) >anoGam1.chr3R 11138287 110 + 53272125 GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUCUCCCAUAGGGAGACAACAAAUGUUAAACUGAUUUUUGGCAAGGG--GAGGAAAGUUCGGGGCUUGCUCCACU ..........((((...(((((.(((((((.((...(.((((((...)))))))..)).))))))).)))))....))))...(--(((..((((.....)))).))))... ( -29.90, z-score = -0.64, R) >consensus GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAG__ACGGAGUGCUAGGGGCUUGCUCCACC ...(((((((((((((.(((((.(((((((.((.....((((((...))))))...)).))))))).)))))....))).......))))).)))))((((....))))... (-28.10 = -27.53 + -0.57)


| Location | 13,215,869 – 13,215,979 |
|---|---|
| Length | 110 |
| Sequences | 14 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 88.57 |
| Shannon entropy | 0.27981 |
| G+C content | 0.40932 |
| Mean single sequence MFE | -37.45 |
| Consensus MFE | -30.33 |
| Energy contribution | -29.81 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.46 |
| Mean z-score | -5.00 |
| Structure conservation index | 0.81 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.96 |
| SVM RNA-class probability | 0.999512 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215869 110 - 23011544 GGUGGAGCAAGCUCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -41.30, z-score = -5.67, R) >droSim1.chr2L_random 496082 110 - 909653 GGUGGAGCAAGCUCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -41.30, z-score = -5.67, R) >droSec1.super_16 1370154 110 - 1878335 GGUGGAGCAAGCUCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -41.30, z-score = -5.67, R) >droYak2.chr2L 9643630 110 - 22324452 GGUGGAGCAAGCUCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -41.30, z-score = -5.67, R) >droEre2.scaffold_4929 14417840 110 + 26641161 GGUGGAGCAAGCUCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -41.30, z-score = -5.67, R) >droAna3.scaffold_13340 22987663 110 - 23697760 GGUGGAGCAAGCUCCUAGCACUCCGU--CAGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (..((((...((.....)).))))..--)...........((((((((((((((((...((((((...)))))).....))))))))))))))))................. ( -38.10, z-score = -4.86, R) >dp4.chr4_group4 4123431 110 - 6586962 GGUGGAGCAAGCCCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -38.20, z-score = -5.39, R) >droPer1.super_10 264070 110 + 3432795 GGUGGAGCAAGCCCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -38.20, z-score = -5.39, R) >droWil1.scaffold_180708 407201 110 + 12563649 GGUGGAGCAAGCCCCUAGCACUCCGU--CUGUUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--((((((......((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -39.60, z-score = -5.76, R) >droVir3.scaffold_12723 2221797 110 + 5802038 GGUGGAGCAAGCCCCUAGCACUCCGU--CAAUUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (..((((...((.....)).))))..--)...........((((((((((((((((...((((((...)))))).....))))))))))))))))................. ( -35.00, z-score = -4.85, R) >droMoj3.scaffold_6500 15233951 110 - 32352404 GGUGGAGCAAGCCCCUAGCACUCCGU--CUAUUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (..((((...((.....)).))))..--)...........((((((((((((((((...((((((...)))))).....))))))))))))))))................. ( -34.50, z-score = -4.57, R) >droGri2.scaffold_15252 4051928 110 - 17193109 GGUGGAGCAAGCCCCUAGCACUCCGU--CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC (((((((...((.....)).))))((--(((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))))).... ( -38.20, z-score = -5.39, R) >apiMel3.Group1 9104471 109 - 25854376 UUCUCAACGAACGAUUAUUAUCGCGUGGAUAAUUUGGAGGGUUAAUAGCCCGCUUA-AACAUUUCAAUAAAGC--CUAUCAGAUAUUAACUUCGUAAUAGCGAAUAUUACAC ...((.(((..((((....))))))).))..((((((.((((.....))))((((.-............))))--...)))))).........((((((.....)))))).. ( -19.02, z-score = -0.53, R) >anoGam1.chr3R 11138287 110 - 53272125 AGUGGAGCAAGCCCCGAACUUUCCUC--CCCUUGCCAAAAAUCAGUUUAACAUUUGUUGUCUCCCUAUGGGAGAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC .((((.(((((....((....))...--..))))).....((((((((((((((((..(((((((...)))))).)...))))))))))))))))...........)))).. ( -37.00, z-score = -4.94, R) >consensus GGUGGAGCAAGCCCCUAGCACUCCGU__CUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC ...((((...((.....)).))))................((((((((((((((((...((((((...)))))).....))))))))))))))))................. (-30.33 = -29.81 + -0.52)


| Location | 13,215,909 – 13,216,012 |
|---|---|
| Length | 103 |
| Sequences | 13 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 95.47 |
| Shannon entropy | 0.10421 |
| G+C content | 0.51382 |
| Mean single sequence MFE | -31.94 |
| Consensus MFE | -31.49 |
| Energy contribution | -30.53 |
| Covariance contribution | -0.96 |
| Combinations/Pair | 1.19 |
| Mean z-score | -0.79 |
| Structure conservation index | 0.99 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.589429 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215909 103 + 23011544 CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -32.60, z-score = -1.04, R) >droSim1.chr2L_random 496122 103 + 909653 CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -32.60, z-score = -1.04, R) >droSec1.super_16 1370194 103 + 1878335 CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -32.60, z-score = -1.04, R) >droYak2.chr2L 9643670 103 + 22324452 CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -32.60, z-score = -1.04, R) >droEre2.scaffold_4929 14417880 103 - 26641161 CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -32.60, z-score = -1.04, R) >droAna3.scaffold_13340 22987703 103 + 23697760 CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCUGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG .(((((..(..((((.....))))...)..)....))))....(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -31.60, z-score = -0.80, R) >dp4.chr4_group4 4123471 103 + 6586962 CUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -31.10, z-score = -0.53, R) >droPer1.super_10 264110 103 - 3432795 CUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -31.10, z-score = -0.53, R) >droWil1.scaffold_180708 407241 103 - 12563649 CUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAACAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))...........((....))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -31.50, z-score = -0.67, R) >droVir3.scaffold_12723 2221837 103 - 5802038 CUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAAUUGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG .(((((..(..((((.....))))...)..)....))))....(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -30.30, z-score = -0.49, R) >droMoj3.scaffold_6500 15233991 103 + 32352404 CUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAAUAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG .(((((..(..((((.....))))...)..)....))))....(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -30.50, z-score = -0.55, R) >droGri2.scaffold_15252 4051968 103 + 17193109 CUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...))))((((.....))))....((((((...))))))(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). ( -31.10, z-score = -0.53, R) >anoGam1.chr3R 11138327 103 + 53272125 UCCCAUAGGGAGACAACAAAUGUUAAACUGAUUUUUGGCAAGGGGAGGAAAGUUCGGGGCUUGCUCCACUUCUUCCGCGGGUUGGCCCGGUAUUGCAGUACCG .......(((...((((...(((((((......))))))).(.(((((((.((..((((....))))))))))))).)..)))).)))(((((....))))). ( -35.00, z-score = -0.94, R) >consensus CUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCG ((((...)))).....((((.(((.....))).))))......(((((((.(...((((....)))).))))))))..(((....)))(((((....))))). (-31.49 = -30.53 + -0.96)

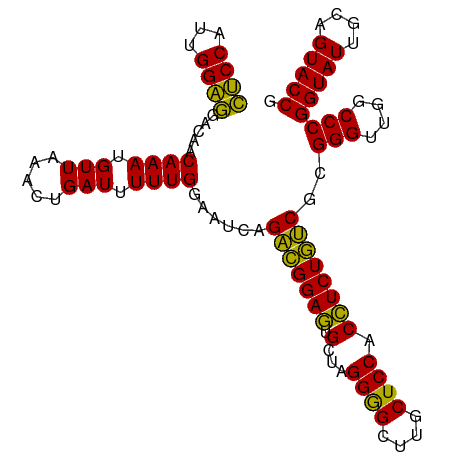
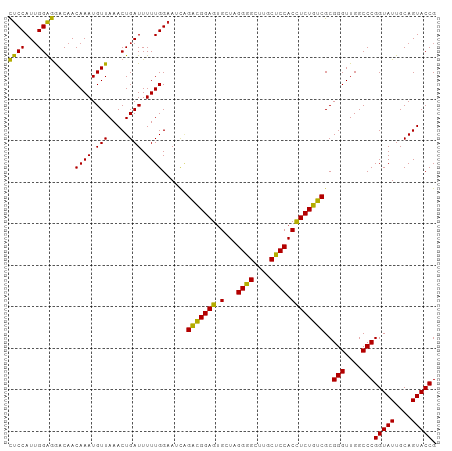
| Location | 13,215,941 – 13,216,042 |
|---|---|
| Length | 101 |
| Sequences | 14 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 84.94 |
| Shannon entropy | 0.35350 |
| G+C content | 0.52769 |
| Mean single sequence MFE | -35.65 |
| Consensus MFE | -31.10 |
| Energy contribution | -30.76 |
| Covariance contribution | -0.34 |
| Combinations/Pair | 1.42 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.87 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.894717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 13215941 101 + 23011544 UUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUUAAUAAUAAAU--- ...........(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----.............--- ( -35.20, z-score = -1.30, R) >droSim1.chr2L_random 496154 101 + 909653 UUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUGAAUAAUAAAU--- .((..(.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----)..))........--- ( -36.90, z-score = -1.62, R) >droSec1.super_16 1370226 101 + 1878335 UUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUGAAUAAUAAAU--- .((..(.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----)..))........--- ( -36.90, z-score = -1.62, R) >droYak2.chr2L 9643702 101 + 22324452 UUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUGAAUAAUAAAU--- .((..(.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----)..))........--- ( -36.90, z-score = -1.62, R) >droEre2.scaffold_4929 14417912 101 - 26641161 UUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUGAAAAAUAAAU--- (((..(.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----)..))).......--- ( -37.80, z-score = -1.89, R) >droAna3.scaffold_13340 22987735 101 + 23697760 UUUUGGAAUCUGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAC-----CUAAUACAUAAAU--- ....((.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...))))))).)-----)............--- ( -36.50, z-score = -1.42, R) >dp4.chr4_group4 4123503 101 + 6586962 UUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUAAAUAAUCAAU--- .(((((.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----)))))........--- ( -36.50, z-score = -1.12, R) >droPer1.super_10 264142 101 - 3432795 UUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUUAAUAAUAAAU--- ...........(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----.............--- ( -34.30, z-score = -0.56, R) >droWil1.scaffold_180708 407273 101 - 12563649 UUUUGGAAACAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUGAAUAAUAAAU--- ...((....))(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----.............--- ( -36.70, z-score = -1.15, R) >droVir3.scaffold_12723 2221869 101 - 5802038 UUUUGGAAAUUGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----UUAACCGAUAAAU--- ..((((.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----....)))).....--- ( -37.40, z-score = -1.15, R) >droMoj3.scaffold_6500 15234023 100 + 32352404 UUUUGGAAAUAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUU-CGGCCCAA-----CUAAGAAAUAAAU--- ((((((.....(((((((.(...((((....)))).))))))))..((((((.((((((...........))))))...-))))))..-----)))))).......--- ( -38.10, z-score = -1.64, R) >droGri2.scaffold_15252 4052000 101 + 17193109 UUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA-----CUAAAUAAUUAAU--- .(((((.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..-----)))))........--- ( -36.50, z-score = -1.21, R) >apiMel3.Group1 9104540 99 + 25854376 -CCUCCAAAUUAUCCACGCGAUAAUAA-UCGUUCGUUGAGAAUCGUUCGUGUGCUUUAUCUUGAGCCA--GUAAAGUUUUCGGGCCAA---CGAUGAAUUGUUAAU--- -.............(((((((......-(((.....))).......))))))).(((((((((.(((.--(.........).))))))---.))))))........--- ( -19.22, z-score = -0.18, R) >anoGam1.chr3R 11138359 107 + 53272125 UUUUGGCAAGGGGAGGAAAGUUCGGGGCUUGCUCCACUUCUUCCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAU--CGGCCCACAUACUCUUAUUAACAAAAAUU (((((....(.(((((((.((..((((....))))))))))))).)(((((((((((((...........)))))).)--))))))...............)))))... ( -40.20, z-score = -1.78, R) >consensus UUUUGGAAUCAGACGGAGUGCUAGGAGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAA_____CUGAAUAAUAAAU___ ...........(((((((.(...((((....)))).))))))))..((((((.((((((...........))))))....))))))....................... (-31.10 = -30.76 + -0.34)

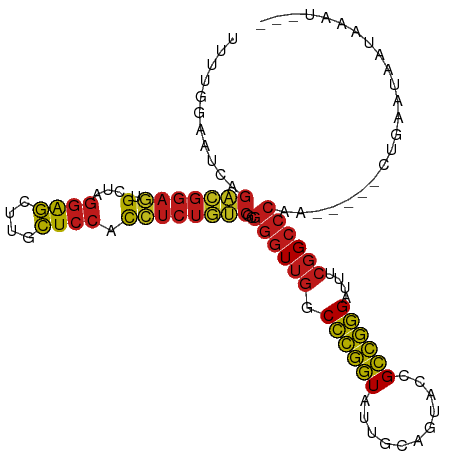
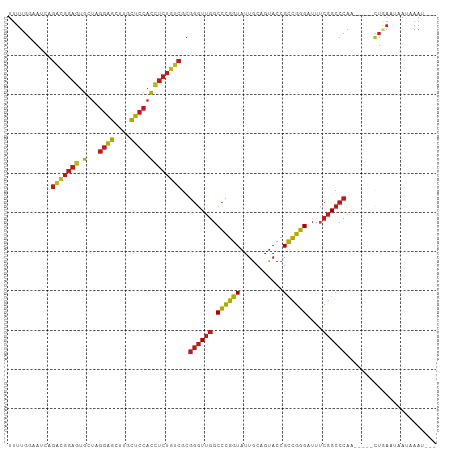
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:37:26 2011