| Sequence ID | dm3.chrX |
|---|---|
| Location | 20,107,432 – 20,107,578 |
| Length | 146 |
| Max. P | 0.930699 |

| Location | 20,107,432 – 20,107,578 |
|---|---|
| Length | 146 |
| Sequences | 6 |
| Columns | 158 |
| Reading direction | forward |
| Mean pairwise identity | 69.55 |
| Shannon entropy | 0.57212 |
| G+C content | 0.45568 |
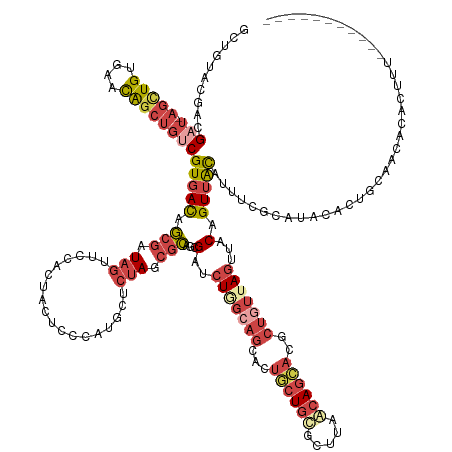
| Mean single sequence MFE | -46.51 |
| Consensus MFE | -20.50 |
| Energy contribution | -22.27 |
| Covariance contribution | 1.77 |
| Combinations/Pair | 1.42 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.44 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.36 |
| SVM RNA-class probability | 0.930699 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
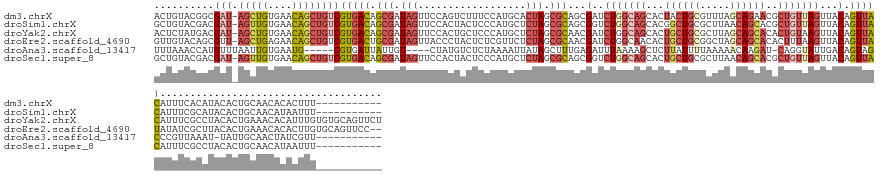
>dm3.chrX 20107432 146 + 22422827 ACUGUACGGCGAU-AGCUGUGAACAGCUGUCGUGACAGCGAUAGUUCCAGUCUUUCCAUGCACUAGCGCAGCGAUCUGGCAGCACUACUGCGUUUAGCAGAACGCUGUUAGUUACAGUUACAUUUCACAUACACUGCAACACACUUU----------- (((((((.(((((-(((((....))))))))))(((((((...(((((((....((..(((......)))..)).)))).)))....((((.....))))..))))))).).)))))).............................----------- ( -50.20, z-score = -2.46, R) >droSim1.chrX 15436754 146 + 17042790 GCUGUACGACGAU-AGUUGUGAACAGCUGUCGUGACAGCGAUAGUUCCACUACUCCCAUGCUCUAGCGCAGCGGUCUGGCAGCACGGCUGCGCUUAACAGCACGCUGUUAGUUACAGUUACAUUUCGCAUACACUGCAACAUAAUUU----------- (((((((.(((((-(((((....))))))))))(((((((.(((.....)))......((((..((((((((.(((.....).)).))))))))....))))))))))).).))))))........(((.....)))..........----------- ( -55.10, z-score = -3.24, R) >droYak2.chrX 19346500 157 + 21770863 ACUCUAUGACGAU-AGCUGUGAACAGCUGUCGUGACAGCGAUAGUUCCACUGCUCCCAUGCUCUAGCGCAACGAUCUGGCAGCACUGCUGCGCUUAGCAGCACACUGUAAGUUACAGUUACAUUUCGCCUACACUGAAACACAUUUGUGUGCAGUUCU ......(.(((((-(((((....)))))))))).)..((((.........((((.((((((......)))......))).)))).((((((.....)))))).((((((...))))))......))))....((((..((((....)))).))))... ( -50.10, z-score = -0.95, R) >droEre2.scaffold_4690 10201850 155 + 18748788 GUUGUACAGCGUU-AGCUGAGAACAGCUGUCGUGACUGCGAUAGUUACCCUACUCUCGUUCUCUAGCGCAACGAUCUGGCAACACUGCUGCGGCUAGCAGCACACUUUAAGUUACAGUUAUAUAUCGCUUACACUGAAACACACUUGUGCAGUUCC-- (((((.(((((((-.((((((((((((((((((....)))))))))...........)))))).)))..))))..))))))))..((((((.....))))))(((.....(((.((((..............)))).)))......))).......-- ( -45.14, z-score = -0.57, R) >droAna3.scaffold_13417 5281839 136 - 6960332 UUUAAACCAUGUUUAAUUGUGAAUG-----CGUGAUUAUUGU----CUAUGUCUCUAAAAUUAUAGCUUUGAGAUUUAAAAGCUCUUAUUUUAAAAACAAGAU-CAGGUAUUGACAGUAGCCCGUUAAAU-UAUUGCAACUAUCGUU----------- ..(((((...)))))(((((.((((-----(.(((((.((((----.........((((((...((((((........))))))...))))))...)))))))-)).))))).)))))............-................----------- ( -23.20, z-score = -0.50, R) >droSec1.super_8 2302660 146 + 3762037 GCUGUACGACGAU-AGUUGUGAACAGCUGUCGUGACAGCGAUAGUUCCACUACUCCCAUGCUCUAGCGCAGCGGUCUGGCAGCACUGCUGCGCUUAACAGCACGCUGUUAGUUACAGUUACAUUUCGCCUACACUGCAACAUAAUUU----------- (((((((.(((((-(((((....))))))))))(((((((.(((.....)))......((((..((((((((((((.....).)))))))))))....))))))))))).).))))))........((.......))..........----------- ( -55.30, z-score = -3.43, R) >consensus GCUGUACGACGAU_AGCUGUGAACAGCUGUCGUGACAGCGAUAGUUCCACUACUCCCAUGCUCUAGCGCAGCGAUCUGGCAGCACUGCUGCGCUUAACAGCACGCUGUUAGUUACAGUUACAUUUCGCAUACACUGCAACACACUUU___________ ((((((((((.....)))))....(((((((((....))))))))).....................)))))...(((((((...((((((.....))))))..)))))))...((((..............))))...................... (-20.50 = -22.27 + 1.77)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 02:03:01 2011