
| Sequence ID | dm3.chrX |
|---|---|
| Location | 19,495,260 – 19,495,377 |
| Length | 117 |
| Max. P | 0.603820 |

| Location | 19,495,260 – 19,495,377 |
|---|---|
| Length | 117 |
| Sequences | 14 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 74.57 |
| Shannon entropy | 0.58798 |
| G+C content | 0.49611 |
| Mean single sequence MFE | -36.83 |
| Consensus MFE | -13.20 |
| Energy contribution | -11.84 |
| Covariance contribution | -1.36 |
| Combinations/Pair | 1.92 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.603820 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
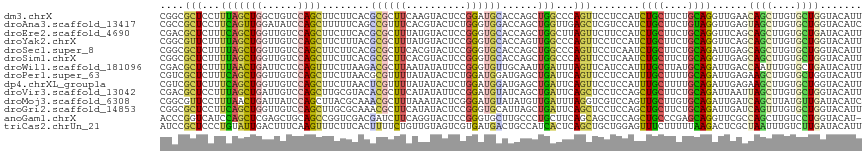
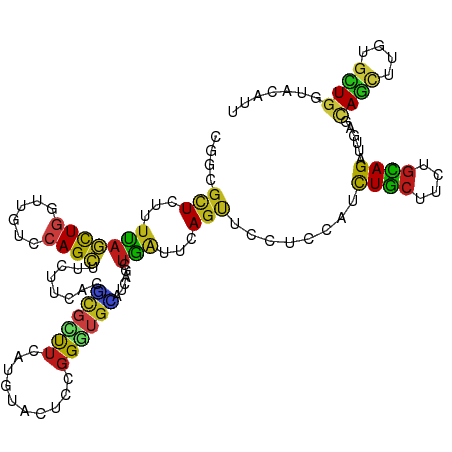
>dm3.chrX 19495260 117 + 22422827 CGGCGCUCCUUUAGCUGGCUGUCCAGCUUCUUCACGCGCUUCAAGUACUCCGGAUGCACCAGCUGGCCCAGUUCCUCCAUCUGCUUCUGCAGGUUGAACAGCUUGUGCUGGUACAUU .(((((......((((((....)))))).......)))))............((((.((((((.(((...((((....((((((....)))))).)))).)))...)))))).)))) ( -41.42, z-score = -1.64, R) >droAna3.scaffold_13417 1131162 117 + 6960332 CGCCGCUCCUUCAGUUGGAUAUCCAGCUUUUUCAGCCGUUUCACGUACUCUGGGUGGACCAGCUGGUUGAGCUCGUCCAUCUGCUUCUGUAGGUUGAGUAGCUUGUGCUGGUACAUC (((((((.....((((((....)))))).....)))((.....)).......)))).((((((....((((((..((.((((((....)))))).))..)))))).))))))..... ( -38.50, z-score = -1.15, R) >droEre2.scaffold_4690 9722805 117 + 18748788 CGACGCUCUUUCAGCUGGUUGUCCAGCUUCUUCACGCGCUUUAUGUACUCCGGGUGCACCAGCUGGCUUAGUUCUUCCAUCUGCUUCUGCAGGUUCAGCAGCUUGUGCUGAUACAUU .((.(((...((((((((((..((.((........))((.....)).....))..).)))))))))...)))))....((((((....))))))((((((.....))))))...... ( -35.40, z-score = -0.82, R) >droYak2.chrX 9925876 117 - 21770863 CGGCGUUCUUUUAGCUGGUUGUCCAGCUUCUUUACGCGCUUUAUGUACUCCGGGUGCACCAGUUGGCCCAGUUCCUCCAUCUGCUUCUGCAGGUUCAGCAGCUUGUGCUGGUACAUU ..((((......((((((....)))))).....)))).....((((((...((((((....))..)))).........((((((....)))))).(((((.....))))))))))). ( -38.60, z-score = -1.71, R) >droSec1.super_8 1775074 117 + 3762037 CGGCGCUCUUUUAGCUGGUUGUCCAGCUUCUUCACGCGCUUCACGUACUCCGGGUGCACCAGCUGGCCCAGUUCCUCAAUCUGCUUCUGCAGAUUGAGCAGCUUGUGCUGGUACAUU .(((((......((((((....)))))).......)))))....((((.....))))((((((....(.((((.((((((((((....)))))))))).)))).).))))))..... ( -49.72, z-score = -4.08, R) >droSim1.chrX 15053093 117 + 17042790 CGGCGCUCUUUUAGCUGGUUGUCCAGCUUCUUCACGCGCUUCACGUACUCCGGGUGCACCAGCUGGCCCAGUUCCUCAAUCUGCUUCUGCAGGUUGAGCAGCUUGUGCUGGUACAUU .(((((......((((((....)))))).......)))))....((((.....))))((((((....(.((((.((((((((((....)))))))))).)))).).))))))..... ( -49.12, z-score = -3.63, R) >droWil1.scaffold_181096 7271746 117 + 12416693 CGACGCUCUUUUAACUGAUUCUCCAGUUUCUUAAGACGCUUAAUAUAUUCCGGGUGUUGCAAUUGAUUUAGUUCAUCCAUUUGCUUAUGCAGAUUGACCAAUUUGUGCUGAUACAUU ....((((((..(((((......)))))....)))).))..............((((((((..(((......)))((.((((((....)))))).))........))).)))))... ( -22.60, z-score = -0.72, R) >droPer1.super_63 65733 117 - 372969 CGUCGCUCUUUCAGCUGGUUGUCCAGCUUCUUAACGCGUUUUAUAUACUCUGGAUGGAUGAGCUGAUUCAGUUCCUCCAUUUGCUUUUGCAGAUUGAGAAGCUUGUGCUGGUACAUU ..........((((((((....)))((((((((((.................((((((.((((((...)))))).))))))(((....)))).)))))))))....)))))...... ( -34.40, z-score = -1.57, R) >dp4.chrXL_group1a 5640407 117 + 9151740 CGUCGCUCUUUCAGCUGGUUGUCCAGCUUCUUAACUCGUUUUAUAUACUCUGGAUGGAUGAGCUGAUUCAGUUCCUCCAUUUGCUUUUGCAGAUUGAGAAGCUUGUGCUGGUACAUU ..........((((((((....)))(((((((((.((...............((((((.((((((...)))))).))))))(((....))))))))))))))....)))))...... ( -35.70, z-score = -2.29, R) >droVir3.scaffold_13042 3598438 117 + 5191987 CGACGCUCCUUUAGCUGAUUGUCCAGCUUGCGUACACGCUUCAUAUAUUCCGGAUGUAUCAGCUGAUUCAGCUCCUCCAGCUGCUUCUGCAGAUUAAUUAGCUUGUGCUGGUACAUU ..((((......(((((......))))).))))...................(((((((((((......((((.......((((....)))).......))))...))))))))))) ( -34.04, z-score = -1.70, R) >droMoj3.scaffold_6308 823499 117 + 3356042 CGGCGUUCCUUUAACUGAUUAUCCAGCUUACGCAAACGCUUUAAAUACUCGGGAUGUAUAUGUUGAUUUAGGUCGUCCAGUUGCUUGUGCAGAUUGAUCAGCUUAUGUUGAUACAUC .((((..((((((((....(((((.((....))...((...........))))))).....)))))...))).))))(((((((....))).))))((((((....))))))..... ( -25.00, z-score = 0.24, R) >droGri2.scaffold_14853 10125404 117 + 10151454 CGGCGCUCCUUCAGCUGGUUGUCCAGCUUGCGCAAACGCUUCAUAUACUCCGGGUGCAUUAGCUGAUUCAGCUCCUCCAGCUGCUUCUGCAGAUUGAUCAGUUUGUGCUGGUACAUU ..((((((....((((((....)))))).(((....)))............))))))((((((.((..(((((.....)))))..)).(((((((....)))))))))))))..... ( -37.40, z-score = -0.67, R) >anoGam1.chrX 13458268 116 + 22145176 ACCCGGUCAUCCAGCUCGAGCUGCAGCCGGUCGACGAUCUUCAGGUACUCCGGGUGCUUGCCCUGCUUCAGCAGCUCCAGCUGCCCGAGCAGGUUCGCCAGCUUGUCCUGGUACAU- .....((...((((..(((((((((.((((...((.........))...)))).)))..(((((((((..(((((....)))))..)))))))...)).))))))..)))).))..- ( -48.40, z-score = -1.94, R) >triCas2.chrUn_21 448492 117 - 494127 AUCCGCUCCCUGUAUUGACUUUCAAGUUUCUUCACUUUUCUGUUGUAGUCGUGAUGACUGCCAUCACUCAGCUGCUGGAGUUUCUUUUUAAGACUCGCUAAUUUGUCUUGAUACAUU ....(((((.......(((....((((......))))....)))(((((.((((((.....))))))...))))).)))))......(((((((..........)))))))...... ( -25.30, z-score = -1.62, R) >consensus CGGCGCUCUUUUAGCUGGUUGUCCAGCUUCUUCACGCGCUUCAUGUACUCCGGGUGCAUCAGCUGAUUCAGUUCCUCCAUCUGCUUCUGCAGAUUGAGCAGCUUGUGCUGGUACAUU ....(((...(((((((......))))........((((((..........))))))......)))...)))........((((....))))......((((....))))....... (-13.20 = -11.84 + -1.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 02:01:36 2011