
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 11,987,557 – 11,987,668 |
| Length | 111 |
| Max. P | 0.990919 |

| Location | 11,987,557 – 11,987,661 |
|---|---|
| Length | 104 |
| Sequences | 11 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 67.00 |
| Shannon entropy | 0.68798 |
| G+C content | 0.55832 |
| Mean single sequence MFE | -42.23 |
| Consensus MFE | -16.43 |
| Energy contribution | -15.24 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.83 |
| Mean z-score | -2.20 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.45 |
| SVM RNA-class probability | 0.990919 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
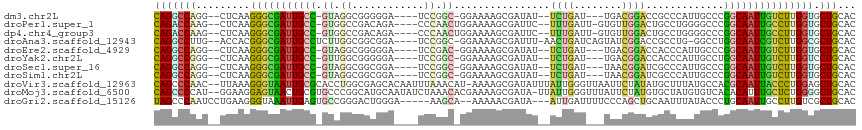
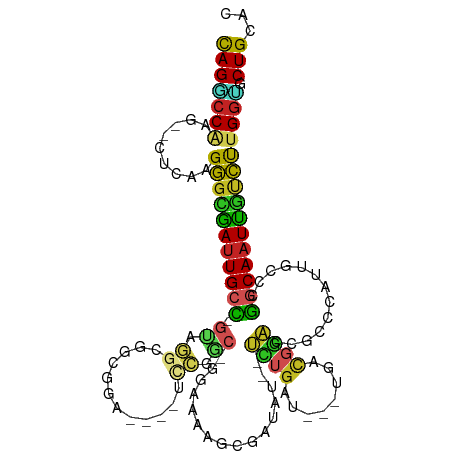
>dm3.chr2L 11987557 104 - 23011544 CAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGGGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UGACGGACCGCCCAUUGCCCGGCAAUUGUCUUGGUGCUGCAC ...((..((--(.(.((((((((((((-(..((((((((.----((((.(-((((........)--))))..---...)))))).)))...)))))))))))))))).).))))).. ( -45.40, z-score = -1.82, R) >droPer1.super_1 1552241 107 + 10282868 CAGACCAAG--CUCAAGGGCGAUUGCC-GUGGCCGACAGA----CCCAACUGGAAAAGCGAUUC--UUUGAUU-GUGUUGGACUGCCUGGGGCCCGGCAAUUGCCUUGGUGCUGCAC .......((--(.(.((((((((((((-(.((((..(((.----((((((.......((((((.--...))))-)))))))...).))).))))))))))))))))).).))).... ( -45.21, z-score = -2.72, R) >dp4.chr4_group3 4406959 107 + 11692001 CAGACCAAG--CUCAAGGGCGAUUGCC-GUGGCCGACAGA----CCCAACUGGAAAAGCGAUUC--UUUGAUU-GUGUUGGACUGCCUGGGGCCCGGCAAUUGCCUUGGUGCUGCAC .......((--(.(.((((((((((((-(.((((..(((.----((((((.......((((((.--...))))-)))))))...).))).))))))))))))))))).).))).... ( -45.21, z-score = -2.72, R) >droAna3.scaffold_12943 2784560 108 + 5039921 CAGGCCUUG--ACCACGGGCGAUUGCCUCUUGGCGGCGGA----UCCGGC-GGAAAAGCGAUUU-AACUGAUCAGUAUCGGACCGCCUG-GGCCUGGCAAUCGUCUUGGCGCUGCAC ...((..((--.(((.(((((((((((....((((((((.----((((((-......))((((.-....)))).....)))))))))..-.))).))))))))))))))))..)).. ( -49.40, z-score = -2.87, R) >droEre2.scaffold_4929 13218453 104 + 26641161 CAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGGGGA----UCCGAC-GGAAAAGCGAUAU--UCUGAU---UGACGGACCACCCAUUGCCCGGCAAUUGUCUUGGUGCUGCAC ...((..((--(.(.((((((((((((-(..((((((((.----((((.(-((((........)--))))..---...)))))).)))...)))))))))))))))).).))))).. ( -43.20, z-score = -2.11, R) >droYak2.chr2L 8410270 104 - 22324452 CAGGCCGGG--CUCAAGGGCGAUUGCC-GUUGGCGGGGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UGACGGACCACCCAUUGCCUGGCAAUUGUCUUGGUCCUGCAC ...((.(((--((..((((((((((((-(..((((((((.----((((.(-((((........)--))))..---...)))))).)))...))))))))))))))))))))).)).. ( -47.70, z-score = -2.75, R) >droSec1.super_16 186984 104 - 1878335 CAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGCGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UAACGGAUCGCCCAUUGCCCGGCAAUUGUCUUGGUGCUGCAC ...((..((--(.(.((((((((((((-(..(((((((.(----((((.(-((((........)--))))..---...)))))))))....)))))))))))))))).).))))).. ( -46.00, z-score = -2.36, R) >droSim1.chr2L 11795530 104 - 22036055 CAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGCGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UAACGGAUCGCCCAUUGCCCGGCAAUUGUCUUGGUGCUGCAC ...((..((--(.(.((((((((((((-(..(((((((.(----((((.(-((((........)--))))..---...)))))))))....)))))))))))))))).).))))).. ( -46.00, z-score = -2.36, R) >droVir3.scaffold_12963 7003903 114 + 20206255 CAGCCCAAC--UUAAAGGGUAAUUGCGCACCUGGCGAGCACAAUUUAAACAU-AAAAGCGAUAUUUAUUGGGUUAAUUCUAUAUGCUUUAUGCCACGCAAUUACCCUGGAGCUGCAC (((((((..--.....(((((((((((....(((((((((............-.....((((....))))((......))...)))))...)))))))))))))))))).))))... ( -33.31, z-score = -2.51, R) >droMoj3.scaffold_6500 15605234 114 + 32352404 CAGCCCCAU--GGAAGGAGUAACUGCGUGCCCGGCAUGCAAUAUCUAAACACGAAAAGCGAUA-UUAUUGGGUUUAUUCUAUGUGCUAUGUGUCACACAUUUGCUCUGGGGCUGCAC ((((((((.--....(((((((((((((((...))))))((((((..............))))-))....)).)))))))..(.((.(((((...)))))..)).)))))))))... ( -35.54, z-score = -1.31, R) >droGri2.scaffold_15126 6657904 107 - 8399593 UAGCCCAAUCCUGAAGGGUAAAUUGAGUGCCGGGACUGGGA-----AAGCA--AAAAACGAUA---AUUGAUUUUCCCAGCUGCAAUUUAUACCCUGCAAUUGCCUUGUCGCUGCAC ((((.(((......((((((...(((((((..((.((((((-----(((((--(.........---.))).))))))))))))).)))))))))))((....)).)))..))))... ( -27.60, z-score = -0.68, R) >consensus CAGGCCAAG__CUCAAGGGCGAUUGCC_GUAGGCGGCGGA____UCCGGC_GGAAAAGCGAUAU__UCUGAU___UGACGGACCGCCCAUUGCCCGGCAAUUGUCUUGGUGCUGCAC (((((((.........(((((((((((..................((....)).............((((........)))).............))))))))))))))).)))... (-16.43 = -15.24 + -1.19)
| Location | 11,987,564 – 11,987,668 |
|---|---|
| Length | 104 |
| Sequences | 11 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 64.63 |
| Shannon entropy | 0.72981 |
| G+C content | 0.54383 |
| Mean single sequence MFE | -37.88 |
| Consensus MFE | -14.23 |
| Energy contribution | -12.97 |
| Covariance contribution | -1.25 |
| Combinations/Pair | 1.89 |
| Mean z-score | -1.15 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.749203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
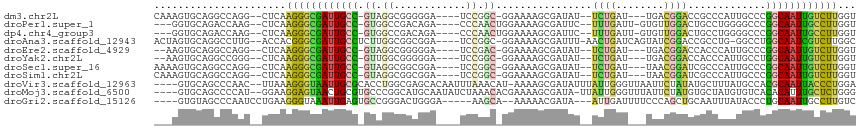
>dm3.chr2L 11987564 104 - 23011544 CAAAGUGCAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGGGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UGACGGACCGCCCAUUGCCCGGCAAUUGUCUUGGU ..........((((..--.....(((((((((((-(..((((((((.----((((.(-((((........)--))))..---...)))))).)))...))))))))))))))))))) ( -40.51, z-score = -0.68, R) >droPer1.super_1 1552248 104 + 10282868 ---GGUGCAGACCAAG--CUCAAGGGCGAUUGCC-GUGGCCGACAGA----CCCAACUGGAAAAGCGAUUC--UUUGAUU-GUGUUGGACUGCCUGGGGCCCGGCAAUUGCCUUGGU ---(((....)))..(--((..((((((((((((-(.((((..(((.----((((((.......((((((.--...))))-)))))))...).))).)))))))))))))))))))) ( -42.91, z-score = -2.02, R) >dp4.chr4_group3 4406966 104 + 11692001 ---GGUGCAGACCAAG--CUCAAGGGCGAUUGCC-GUGGCCGACAGA----CCCAACUGGAAAAGCGAUUC--UUUGAUU-GUGUUGGACUGCCUGGGGCCCGGCAAUUGCCUUGGU ---(((....)))..(--((..((((((((((((-(.((((..(((.----((((((.......((((((.--...))))-)))))))...).))).)))))))))))))))))))) ( -42.91, z-score = -2.02, R) >droAna3.scaffold_12943 2784567 108 + 5039921 ACUAGUGCAGGCCUUG--ACCACGGGCGAUUGCCUCUUGGCGGCGGA----UCCGGC-GGAAAAGCGAUUU-AACUGAUCAGUAUCGGACCGCCUG-GGCCUGGCAAUCGUCUUGGC ................--.(((.(((((((((((....((((((((.----((((((-......))((((.-....)))).....)))))))))..-.))).)))))))))))))). ( -46.00, z-score = -2.07, R) >droEre2.scaffold_4929 13218460 102 + 26641161 --AAGUGCAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGGGGA----UCCGAC-GGAAAAGCGAUAU--UCUGAU---UGACGGACCACCCAUUGCCCGGCAAUUGUCUUGGU --........((((..--.....(((((((((((-(..((((((((.----((((.(-((((........)--))))..---...)))))).)))...))))))))))))))))))) ( -38.31, z-score = -0.90, R) >droYak2.chr2L 8410277 102 - 22324452 --AAGUGCAGGCCGGG--CUCAAGGGCGAUUGCC-GUUGGCGGGGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UGACGGACCACCCAUUGCCUGGCAAUUGUCUUGGU --(((.(((.((((((--(((((.(((....)))-.)))).(((((.----((((.(-((((........)--))))..---...)))))).)))...)))))))...))))))... ( -42.10, z-score = -1.41, R) >droSec1.super_16 186991 104 - 1878335 AAAAGUGCAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGCGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UAACGGAUCGCCCAUUGCCCGGCAAUUGUCUUGGU ..(((.(((.(((.((--(....((((...((((-(....)))))((----((((.(-((((........)--))))..---...))))))))))...))).)))...))))))... ( -42.50, z-score = -1.67, R) >droSim1.chr2L 11795537 104 - 22036055 CAAAGUGCAGGCCAGG--CUCAAGGGCGAUUGCC-GUAGGCGGCGGA----UCCGGC-GGAAAAGCGAUAU--UCUGAU---UAACGGAUCGCCCAUUGCCCGGCAAUUGUCUUGGU ..(((.(((.(((.((--(....((((...((((-(....)))))((----((((.(-((((........)--))))..---...))))))))))...))).)))...))))))... ( -42.50, z-score = -1.68, R) >droVir3.scaffold_12963 7003910 110 + 20206255 ----GUGCAGCCCAAC--UUAAAGGGUAAUUGCGCACCUGGCGAGCACAAUUUAAACAU-AAAAGCGAUAUUUAUUGGGUUAAUUCUAUAUGCUUUAUGCCACGCAAUUACCCUGGA ----............--....((((((((((((....(((((((((............-.....((((....))))((......))...)))))...))))))))))))))))... ( -28.10, z-score = -0.96, R) >droMoj3.scaffold_6500 15605241 110 + 32352404 ----GUGCAGCCCCAU--GGAAGGAGUAACUGCGUGCCCGGCAUGCAAUAUCUAAACACGAAAAGCGAUA-UUAUUGGGUUUAUUCUAUGUGCUAUGUGUCACACAUUUGCUCUGGG ----......((((((--((((.....((((((((((...))))))((((((..............))))-))....))))..))))))).((.(((((...)))))..))...))) ( -25.34, z-score = 1.27, R) >droGri2.scaffold_15126 6657911 103 - 8399593 ----GUGUAGCCCAAUCCUGAAGGGUAAAUUGAGUGCCGGGACUGGGA-----AAGCA--AAAAACGAUA---AUUGAUUUUCCCAGCUGCAAUUUAUACCCUGCAAUUGCCUUGUC ----........((((..((.((((((...(((((((..((.((((((-----(((((--(.........---.))).))))))))))))).))))))))))).))))))....... ( -25.50, z-score = -0.56, R) >consensus ___AGUGCAGGCCAAG__CUCAAGGGCGAUUGCC_GUAGGCGGCGGA____UCCGGC_GGAAAAGCGAUAU__UCUGAU___UGACGGACCGCCCAUUGCCCGGCAAUUGUCUUGGU ......................((((((((((((..................((....)).............((((........)))).............))))))))))))... (-14.23 = -12.97 + -1.25)
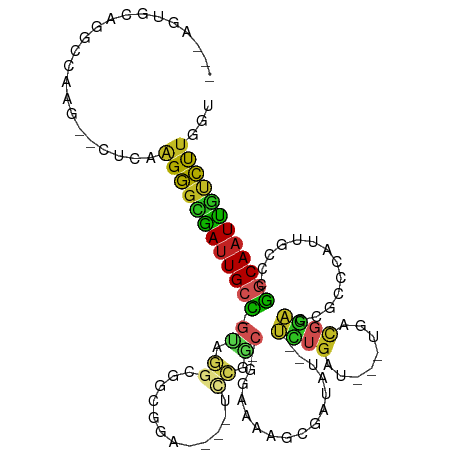
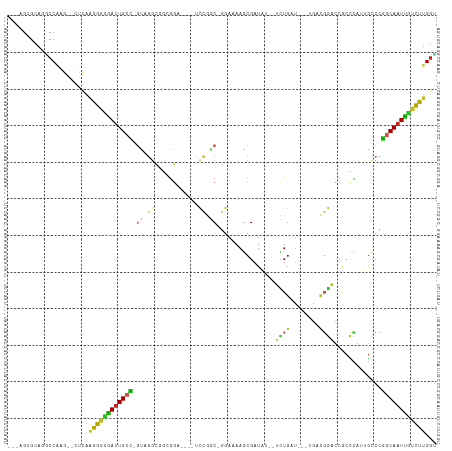
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:34:07 2011