
| Sequence ID | dm3.chrX |
|---|---|
| Location | 19,137,769 – 19,137,924 |
| Length | 155 |
| Max. P | 0.506778 |

| Location | 19,137,769 – 19,137,924 |
|---|---|
| Length | 155 |
| Sequences | 11 |
| Columns | 180 |
| Reading direction | forward |
| Mean pairwise identity | 62.73 |
| Shannon entropy | 0.76509 |
| G+C content | 0.48408 |
| Mean single sequence MFE | -48.85 |
| Consensus MFE | -15.00 |
| Energy contribution | -14.97 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.31 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.506778 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 19137769 155 + 22422827 AAAAUGUCCAGAAAGUACGGCAAGCUGCAGCAGCAGUUCCUCUGUUGCUAUCCGGUGAACAAGAUGGCCUGGUCGGUGAGUAAUCCAUGUGAUACAUUAUGACCCGGCCACCUGUUG----------------AAUCGAUUUGGUG----UCCAU-----UGCAGUCGGUCACGCUGCCC ........(((...((..(((..(((((((((((((.....))))))))...((((.((((.(.(((((.(((((.((((((.((.....))))).)))))))).)))))).)))).----------------.))))........----.....-----.)))))..))))).)))... ( -49.70, z-score = -0.84, R) >droGri2.scaffold_14853 5141261 152 + 10151454 ----AGAGCAACAUGUUUGGCAAACUGCAGCAGCAAUUCCUCUGCUGCUAUCCCGUCAACAAAAUGGCCUGGUCGGUAAGU-----GUGUG-UGUGUUCAAAUAAUGUUGACAAUAA----------------AAUUAGUCACUGCCU--CUCGUUUAUUUGUAGACGCUGCAAUUGCCG ----.((((.....))))(((((..((((((((((.......)))))))....((((.(((((((((...((.((((.(.(-----(....-(((..((((......))))..))).----------------...)).).)))))).--.))))...))))).))))..))).))))). ( -38.90, z-score = -0.31, R) >droMoj3.scaffold_6473 7403166 144 - 16943266 ----AGAGCAACAAGUUCGCCAAACUGCGGCAGCAAUUUCUGUGUUGCUAUCCCGUCAAUAAGAUGGCCUGGUCGGUAAGU-----GGAUAAUGAGCACUUGUCA-AUUGGCAAUUG----------------UAUUGGAU----------UUAUUGUCAUGUAGACGCUGCAACUGCCG ----.((((.....))))((((......(((((((.......)))))))...(((((.....)))))..))))(((((.((-----.(((((((((((.(((((.-...))))).))----------------).......----------)))))))).(((((...)))))))))))) ( -39.31, z-score = 0.34, R) >droVir3.scaffold_12726 706722 143 + 2840439 ----ACAGCAACAAGUUUGGCAAGCUGCAGCAGCAAUUUCUUUGCUGUUAUCCCGUCAAUAAGAUGGCCUGGUCGGUAAGU-----AGUCGUGGGUUAUUUGUAAUAUCA--AAUUG----------------UUGCCACU----------UGCUUGCCGUACAGACGCUGGAGCUGCCG ----..............((((.(((.(((((((((.....)))))......(((((.....))))).(((..((((((((-----((..((((...(((((......))--)))..----------------...)))))----------)))))))))..)))..)))).))))))). ( -53.20, z-score = -3.07, R) >droPer1.super_13 132865 176 + 2293547 AAAAUGACUGACAGGUUCAGCAAAAUGCAGCAGCAAUUCCUGUGCUGUUAUCCAGUCAAUAAAAUGUCGUGGUCGGUAUGU--AUUUGGCUGUGACU--GCACCUGUCCACCUGUUGCAUCUUUAACUUUUGUAAACAACUUUGUAACAAUUUGUAAACUCGCAGUCGGUGAAACUACCA .....(((((.(..(((..((((((((((((((........((((.((((..(((((((....((..((....))..))..--..))))))))))).--))))........))))))))).........((((..(((....))).))))))))).)))..))))))((((....)))). ( -49.39, z-score = -1.99, R) >dp4.chrXL_group1e 4080005 164 - 12523060 AAAAUGACUGACAGGUUCAGCAAAAUGCAGCAGCAAUUCCUGUGCUGUUAUCCAGUCAAUAAAAUGUCCUGGUCGGUAUGU--AUUUGGCUGUGACU--GCACCUGUCCACCUGUUGCAUCUUUAA----------CAACUU--UAAAAAUUUGUAUAAGCGCAGUCGGUGAAACUACCA .........(((((((.(((((......(((((((.......)))))))...(((((((....(((.((.....)))))..--..))))))))).))--).)))))))((((..((((..(((..(----------(((.((--....)).))))..))).))))..))))......... ( -45.00, z-score = -1.63, R) >droAna3.scaffold_12903 494959 164 + 802071 CAAAUGGCACAAAAGUACGACAAACUACAGCAGCAAUUCCUGUGCUGCUAUCCGGUUAAAAAGAUGGCCUGGUCGGUAAGU-UACAUGGCCGGGAAU---CCCCCACUCACCUGUUGCUCCUCCUA-------AAUCGUGUCGGUUUUUUUCCGU-----UGUAGUCGGUGAAGUUGCCU .....(((((........(((.......(((((((.......)))))))....(((((......)))))..)))((..(((-.(((.((..(((...---..))).....))))).)))...))..-------....)))))(((..(((((((.-----......))).))))..))). ( -43.30, z-score = 0.12, R) >droEre2.scaffold_4690 9396985 156 + 18748788 AAAAUGUCCGGGAAGUACGGCAAGCUGCAGCAGCAGUUCCUCUGCUGCUAUCCGGUGUACAAGAUGGCCUGGUCGGUGAGUAUAUAC-AUAU-GUUU---GCGCCGUCCACAUGUUUCAUACC----------AAUCGGUUUGGUG----UCCAU-----UGCAGUCGGUCAAGUUGCCA .........((.((.(..(((..(((((((((((((.....)))))))).............(((((..(((.(((((...((((..-..))-))..---.))))).)))........(((((----------((.....))))))----)))))-----))))))..))).).)).)). ( -52.70, z-score = -1.30, R) >droYak2.chrX 9582994 160 - 21770863 AAAAUGUCCGGGAAGUACGACAAGCUGCAGCAGCAGUUCCUCUGCUGCUAUCCGGUGAACAAGAUGGCCUGGUCGGUGAGUAGACCCCGUGUUGCAU---GCCGCGGCCACCUUUUGCAUCCC----------AAUCUGUUUGUUGCA--UCCAU-----UGCAGUCGGUCAAGUUGCCA ....((.((((((.(((.((...(((((((((((((.....))))))))....((((..(((.((((...((((........)))))))).)))..)---))))))))....)).))).))))----------.........((((((--.....-----))))).))).))........ ( -55.60, z-score = -1.00, R) >droSec1.super_8 1431130 152 + 3762037 AAAAUGUCCACAAAGUACGGCAAGCUGCAGCAGCAGUUCCUCUGCUGCUAUCCGGUGAACAAGAUGGCCUGGUCGGUGAGUAAUCCAUGUGAUACAU---GACCCGGCCACCUGUUG----------------AAUCGGCUUGGUG----UCCAU-----UGCAGUCGGUCAAGCUGCCC ..((((..(((.((((...((.....))((((((((.....))))))))...((((.((((.(.(((((.((((((........))(((.....)))---)))).)))))).)))).----------------.)))))))).)))----..)))-----)(((((.......))))).. ( -55.10, z-score = -1.63, R) >droSim1.chrX 14803146 152 + 17042790 AAAAUGUCCACAAAGUACGGCAAACUGCAGCAGCAGUUCCUCUGCUGCUAUCCGGUGAACAAGAUGGCCUGGUCGGUGAGUAAUCCAUGUGAUACAU---GACCCGGCCACCUGUUG----------------AAUCGGCUUGGUG----UCCAU-----UGCAGUCGGUCAAGCUGCCC ..((((..(((.((((...((.....))((((((((.....))))))))...((((.((((.(.(((((.((((((........))(((.....)))---)))).)))))).)))).----------------.)))))))).)))----..)))-----)(((((.......))))).. ( -55.10, z-score = -1.93, R) >consensus AAAAUGACCAACAAGUACGGCAAACUGCAGCAGCAAUUCCUCUGCUGCUAUCCGGUCAACAAGAUGGCCUGGUCGGUAAGU__ACCAGGUGAUGAAU___GACCCGGCCACCUGUUG________________AAUCGGCUUGGUG____UCCAU_____UGCAGUCGGUGAAGCUGCCA .......................((((((((((((.......))))))).................(((.....)))....................................................................................))))).............. (-15.00 = -14.97 + -0.02)
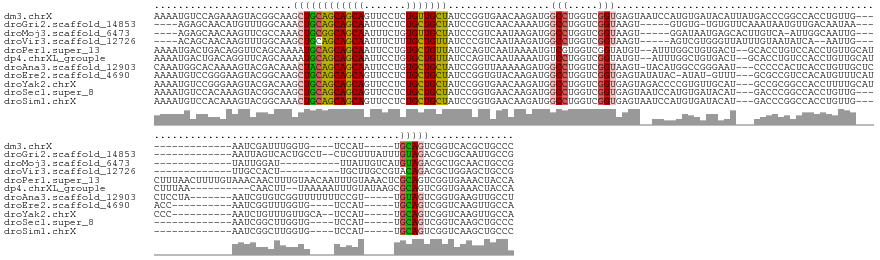


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:59:39 2011