
| Sequence ID | dm3.chrX |
|---|---|
| Location | 18,022,248 – 18,022,349 |
| Length | 101 |
| Max. P | 0.998677 |

| Location | 18,022,248 – 18,022,349 |
|---|---|
| Length | 101 |
| Sequences | 15 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 79.95 |
| Shannon entropy | 0.45507 |
| G+C content | 0.47670 |
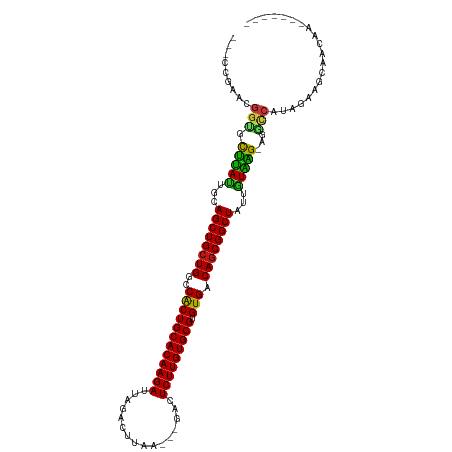
| Mean single sequence MFE | -36.85 |
| Consensus MFE | -34.64 |
| Energy contribution | -34.30 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.39 |
| Mean z-score | -3.25 |
| Structure conservation index | 0.94 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.44 |
| SVM RNA-class probability | 0.998677 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
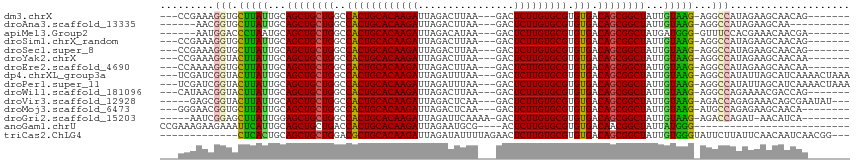
>dm3.chrX 18022248 101 + 22422827 ---CCGAAAGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAGAAGCAACAG------- ---((....)).((((.((((((((((((..((((((((((((..........---...))))))))).))).)))))))...))))).-))))..............------- ( -38.12, z-score = -2.92, R) >droAna3.scaffold_13335 1731751 95 - 3335858 ------AACGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAGAAGCAA---------- ------...(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..)))..........---------- ( -37.92, z-score = -3.17, R) >apiMel3.Group2 1929959 98 + 14465785 ------AAUGGACCCUAAUGCAGCUGCUGGCCACUGCACAAGAUUAGACAUAA---GACUCUUGUGCGUGUGACAGCGGCUAUGAUGGG-GUUUCCACGAAACAACGA------- ------..(((((((((.(..((((((((..((((((((((((..........---...))))))))).))).))))))))..).))))-)..))))...........------- ( -40.32, z-score = -4.89, R) >droSim1.chrX_random 4635957 101 + 5698898 ---CCGAAAGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAGAAGCAACAG------- ---((....)).((((.((((((((((((..((((((((((((..........---...))))))))).))).)))))))...))))).-))))..............------- ( -38.12, z-score = -2.92, R) >droSec1.super_8 332204 101 + 3762037 ---CCGAAAGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAGAAGCAACAG------- ---((....)).((((.((((((((((((..((((((((((((..........---...))))))))).))).)))))))...))))).-))))..............------- ( -38.12, z-score = -2.92, R) >droYak2.chrX 16646323 101 + 21770863 ---CCGAAAGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAGAAGCAACAA------- ---......(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..))).............------- ( -37.02, z-score = -3.16, R) >droEre2.scaffold_4690 8350136 101 + 18748788 ---CCAAAAGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAGAAGCAACAA------- ---......(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..))).............------- ( -37.82, z-score = -2.87, R) >dp4.chrXL_group3a 720227 108 - 2690836 ---UCGAUCGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGAUUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAUUAGCAUCAAAACUAAA ---......(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..)))...((((........)))). ( -37.52, z-score = -3.30, R) >droPer1.super_11 2708523 108 + 2846995 ---UCGAUCGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGAUUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAUAUUAGCAUCAAAACUAAA ---......(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..)))...((((........)))). ( -37.52, z-score = -3.30, R) >droWil1.scaffold_181096 12033233 101 + 12416693 ---CAUAACGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGGCCAGAAAACGACCAG------- ---......(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..))).............------- ( -37.12, z-score = -3.69, R) >droVir3.scaffold_12928 6460039 103 - 7717345 -----GAGCGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUCAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGACCAGAGAAACAGCGAAUAU--- -----....(((.(((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-..))).................--- ( -36.82, z-score = -3.03, R) >droMoj3.scaffold_6473 13732376 100 + 16943266 ---GGGAACGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUCAA---GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AUGCCAGAGAAGCAACA-------- ---......(((((((((...((((((((..((((((((((((..........---...))))))))).))).))))))))...)))))-.))))............-------- ( -37.92, z-score = -2.38, R) >droGri2.scaffold_15203 8453539 99 + 11997470 -----AAUCGGAGCUUAUUGGAGCUGCUGGCCACUGCACAAGAUUAGAUUCAAAA-GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG-AGACCAGAU-AACAUCA-------- -----.(((((..(((((...((((((((..((((((((((((............-...))))))))).))).))))))))...)))))-...)).)))-.......-------- ( -37.36, z-score = -3.99, R) >anoGam1.chrU 21248182 85 - 59568033 CCGAAAGAAGAAAUUCAUUGCAGCUGCUGACCACUGCACAAGAUUAGAAUGCG----ACUCUUGUGCGUGUGACAACGGCUAUUAUGGG-------------------------- (((...(((....))).....(((((.((..((((((((((((...(....).----..))))))))).))).)).)))))....))).-------------------------- ( -26.30, z-score = -2.71, R) >triCas2.ChLG4 5712258 99 - 13894384 -------------CUCACUGCAGCUGCUGGACGCUGCACAAGAUUAGAUAUUUUAGAACUCUUGUGCGUGUGACAGCGGCUAUUGUGGGUAUUCUUAUUCAACAAUCAACGG--- -------------(((((...((((((((.((((.(((((((((((((...)))))...))))))))))))..))))))))...))))).......................--- ( -34.80, z-score = -3.58, R) >consensus ___CCGAACGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUAA___GACUCUUGUGCGUGUGACAGCGGCUAUUGUAAG_AGGCCAUAGAAGCAACAA_______ .........(((.(((((...((((((((..((((((((((((................))))))))).))).))))))))...)))))...))).................... (-34.64 = -34.30 + -0.33)
| Location | 18,022,248 – 18,022,349 |
|---|---|
| Length | 101 |
| Sequences | 15 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 79.95 |
| Shannon entropy | 0.45507 |
| G+C content | 0.47670 |
| Mean single sequence MFE | -30.82 |
| Consensus MFE | -25.47 |
| Energy contribution | -25.56 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.83 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.75 |
| SVM RNA-class probability | 0.994956 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 18022248 101 - 22422827 -------CUGUUGCUUCUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCUUUCGG--- -------.((((((..((..(((..-.........)))((((((((..((((((((...---..........)))))))).)))).))))))..))))))............--- ( -31.12, z-score = -1.67, R) >droAna3.scaffold_13335 1731751 95 + 3335858 ----------UUGCUUCUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCGUU------ ----------........((((...-((((...((((.((((((((..((((((((...---..........)))))))).)))).)))).))))...))))..)))).------ ( -30.02, z-score = -1.99, R) >apiMel3.Group2 1929959 98 - 14465785 -------UCGUUGUUUCGUGGAAAC-CCCAUCAUAGCCGCUGUCACACGCACAAGAGUC---UUAUGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAUUAGGGUCCAUU------ -------..........(((((..(-((.....((((.((((((((..((((((((...---..........)))))))).)))).)))).)))).....)))))))).------ ( -34.72, z-score = -3.34, R) >droSim1.chrX_random 4635957 101 - 5698898 -------CUGUUGCUUCUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCUUUCGG--- -------.((((((..((..(((..-.........)))((((((((..((((((((...---..........)))))))).)))).))))))..))))))............--- ( -31.12, z-score = -1.67, R) >droSec1.super_8 332204 101 - 3762037 -------CUGUUGCUUCUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCUUUCGG--- -------.((((((..((..(((..-.........)))((((((((..((((((((...---..........)))))))).)))).))))))..))))))............--- ( -31.12, z-score = -1.67, R) >droYak2.chrX 16646323 101 - 21770863 -------UUGUUGCUUCUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCUUUCGG--- -------(((((((..((..(((..-.........)))((((((((..((((((((...---..........)))))))).)))).))))))..)))))))...........--- ( -31.92, z-score = -2.29, R) >droEre2.scaffold_4690 8350136 101 - 18748788 -------UUGUUGCUUCUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCUUUUGG--- -------(((((((..((..(((..-.........)))((((((((..((((((((...---..........)))))))).)))).))))))..)))))))...((....))--- ( -32.12, z-score = -1.86, R) >dp4.chrXL_group3a 720227 108 + 2690836 UUUAGUUUUGAUGCUAAUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAAUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCGAUCGA--- .(((((......)))))..(((...-((((...((((.((((((((..((((((((...---..........)))))))).)))).)))).))))...))))..))).....--- ( -32.62, z-score = -2.45, R) >droPer1.super_11 2708523 108 - 2846995 UUUAGUUUUGAUGCUAAUAUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAAUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCGAUCGA--- .(((((......)))))..(((...-((((...((((.((((((((..((((((((...---..........)))))))).)))).)))).))))...))))..))).....--- ( -32.62, z-score = -2.45, R) >droWil1.scaffold_181096 12033233 101 - 12416693 -------CUGGUCGUUUUCUGGCCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCGUUAUG--- -------..(((((.....))))).-((((...((((.((((((((..((((((((...---..........)))))))).)))).)))).))))...))))..........--- ( -31.72, z-score = -2.24, R) >droVir3.scaffold_12928 6460039 103 + 7717345 ---AUAUUCGCUGUUUCUCUGGUCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUGAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCGCUC----- ---.................(((..-((((...((((.((((((((..((((((((...---..........)))))))).)))).)))).))))...)))).)))....----- ( -31.52, z-score = -2.00, R) >droMoj3.scaffold_6473 13732376 100 - 16943266 --------UGUUGCUUCUCUGGCAU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC---UUGAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCGUUCCC--- --------((((((..((..(((..-.........)))((((((((..((((((((...---..........)))))))).)))).))))))..))))))............--- ( -30.92, z-score = -1.47, R) >droGri2.scaffold_15203 8453539 99 - 11997470 --------UGAUGUU-AUCUGGUCU-CUUACAAUAGCCGCUGUCACACGCACAAGAGUC-UUUUGAAUCUAAUCUUGUGCAGUGGCCAGCAGCUCCAAUAAGCUCCGAUU----- --------.......-(((.((...-((((....(((.((((((((..((((((((.((-....))......)))))))).)))).)))).)))....))))..))))).----- ( -29.30, z-score = -2.32, R) >anoGam1.chrU 21248182 85 + 59568033 --------------------------CCCAUAAUAGCCGUUGUCACACGCACAAGAGU----CGCAUUCUAAUCUUGUGCAGUGGUCAGCAGCUGCAAUGAAUUUCUUCUUUCGG --------------------------..(((..((((.((((.(.(((((((((((..----..........)))))))).)))).)))).))))..)))............... ( -26.10, z-score = -3.23, R) >triCas2.ChLG4 5712258 99 + 13894384 ---CCGUUGAUUGUUGAAUAAGAAUACCCACAAUAGCCGCUGUCACACGCACAAGAGUUCUAAAAUAUCUAAUCUUGUGCAGCGUCCAGCAGCUGCAGUGAG------------- ---.........................(((..((((.((((..((.(((((((((................)))))))).).)).)))).))))..)))..------------- ( -25.39, z-score = -1.78, R) >consensus _______UUGUUGCUUCUAUGGCCU_CUUACAAUAGCCGCUGUCACACGCACAAGAGUC___UUAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCGUUCGG___ ..........................((((...((((.((((((((..((((((((................)))))))).)))).)))).))))...))))............. (-25.47 = -25.56 + 0.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:55:13 2011