
| Sequence ID | dm3.chrX |
|---|---|
| Location | 17,835,071 – 17,835,194 |
| Length | 123 |
| Max. P | 0.966544 |

| Location | 17,835,071 – 17,835,184 |
|---|---|
| Length | 113 |
| Sequences | 13 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 82.96 |
| Shannon entropy | 0.38056 |
| G+C content | 0.49562 |
| Mean single sequence MFE | -38.99 |
| Consensus MFE | -31.84 |
| Energy contribution | -31.80 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.82 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.966544 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
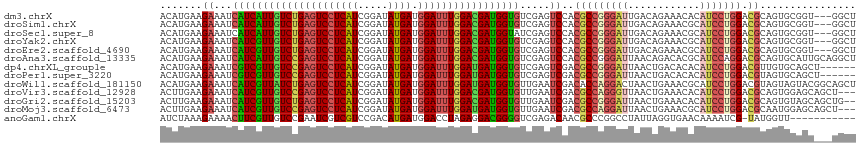
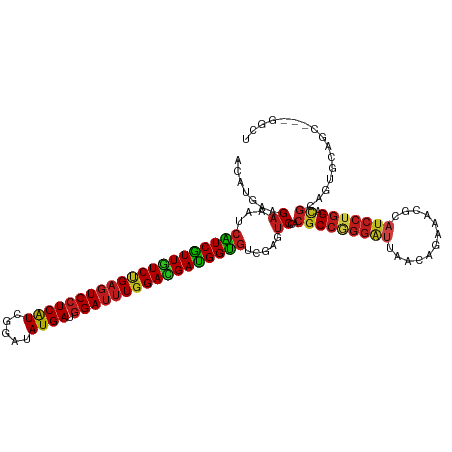
>dm3.chrX 17835071 113 - 22422827 ACAUGAAGAAAUCAUCAUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACACAUCCUGGACGCAGUGCGGU---GGCU ............((((((((((..(((((((((.....)))).)))))..))))))))))...((.((((.(((((((((.......)).))))))).(((...)))))---)))) ( -43.20, z-score = -2.41, R) >droSim1.chrX 13886919 113 - 17042790 ACAUGAAGAAAUCAUCAUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCGGU---GGCU ............((((((((((..(((((((((.....)))).)))))..))))))))))...((.((((.(((((((((.......)).))))))).(((...)))))---)))) ( -42.90, z-score = -2.17, R) >droSec1.super_8 136753 113 - 3762037 ACAUGAAGAAAUCAUCAUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUAUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCGGU---GGCU .............(((((((((..(((((((((.....)))).)))))..)))))))))....((.((((.(((((((((.......)).))))))).(((...)))))---)))) ( -40.60, z-score = -1.82, R) >droYak2.chrX 16453683 113 - 21770863 ACAUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCGGU---GGCU ............((((((((((..(((((((((.....)))).)))))..))))))))))...((.((((.(((((((((.......)).))))))).(((...)))))---)))) ( -42.50, z-score = -1.87, R) >droEre2.scaffold_4690 8160160 113 - 18748788 ACAUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCGGU---GGCU ............((((((((((..(((((((((.....)))).)))))..))))))))))...((.((((.(((((((((.......)).))))))).(((...)))))---)))) ( -42.50, z-score = -1.87, R) >droAna3.scaffold_13335 1536856 116 + 3335858 ACAUGAAGAAAUCAUCAUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUAACAGACACGCAUCCAGGACGCAGUGCAUUGCAGGCU ..((((.....))))((((((((((((((((((.....)))).))))))))))))))(((((.(((((......)))))....)))))...........((..(((...))).)). ( -39.70, z-score = -1.57, R) >dp4.chrXL_group1e 5178215 110 - 12523060 ACAUGAAGAAAUCGUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUCGAGUCGACGCCGGGAUUAACUGACACACAUCCUGGACGUUGUGCAGCU------ ...........(((((((..(((((((((((((.....)))).)))))))))..))))...)))((((((((((((((............))))))).))))).))....------ ( -38.90, z-score = -1.46, R) >droPer1.super_3220 4062 110 - 4900 ACAUGAAGAAAUCGUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUCGAGUCGACGCCGGGAUUAACUGACACACAUCCUGGACGUAGUGCAGCU------ ...(((......(..(((..(((((((((((((.....)))).)))))))))..)))..).....)))((((((((((............))))))).))).........------ ( -36.30, z-score = -0.83, R) >droWil1.scaffold_181150 1804179 116 + 4952429 ACAUGAAGAAAUCAUCGUUAUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUUGAAUCGACACCAGGACUAACUGAAACGCAUCCUGGACGUAGUAGUACGGCAGCU ...((..((...((((((((((..(((((((((.....)))).)))))..)))))))))).....))..))((((((..............)))))).((((....))))...... ( -35.14, z-score = -1.65, R) >droVir3.scaffold_12928 4839933 113 + 7717345 ACUUGAAGAAAUCAUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUUGAAUCGACGCCAGGGUUAACUGAAACACAUCCUGGACGCAGUGGAGCAGCU--- .............((((((((((((((((((((.....)))).))))))))))))))))((((..(((.(((((((((............))))))).))...)))..)))).--- ( -40.90, z-score = -2.70, R) >droGri2.scaffold_15203 1593454 114 - 11997470 ACUUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUUGAAUCGACGCCGGGAUUAACUGAAACACAUCCUGGACGCAGUGUAGCAGCUG-- ..((((...((.((((((((((..(((((((((.....)))).)))))..))))))))))))...))))(((((((((............))))))).))((((......))))-- ( -39.00, z-score = -2.54, R) >droMoj3.scaffold_6473 2256181 113 + 16943266 ACUUGAAGAAAUCAUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUUGAAUCGACGCCAGGAUUAACUGAAACGCAUCCUGGACGCAAUGGAGCAGCU--- .............(((((..(((((((((((((.....)))).)))))))))..)))))((((..(((.(((((((((............))))))).))...)))..)))).--- ( -38.00, z-score = -2.27, R) >anoGam1.chrX 17353068 104 + 22145176 AUCUAAAGAAAACUUCGUUGUCCGAAUCGUCGUCCGACAUGAUGGACCUAGAGGACGGGGUCGAGACAACGCCCGGCCUAUUAGGUGAACAAAAUCG-UAUGGUU----------- ..........((((..((((..(((....)))..))))(((((.((((((((((.(.(((.((......))))).)))).))))))...)...))))-)..))))----------- ( -27.20, z-score = 0.36, R) >consensus ACAUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUAACAGAAACGCAUCCUGGACGCAGUGCAGC___GGCU .......((...(((((((((((((((((((((.....)))).))))))))))))))))).....))..(((((((((............))))))).))................ (-31.84 = -31.80 + -0.04)
| Location | 17,835,077 – 17,835,194 |
|---|---|
| Length | 117 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.03 |
| Shannon entropy | 0.29361 |
| G+C content | 0.48998 |
| Mean single sequence MFE | -39.54 |
| Consensus MFE | -32.44 |
| Energy contribution | -32.92 |
| Covariance contribution | 0.49 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.82 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.08 |
| SVM RNA-class probability | 0.888137 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
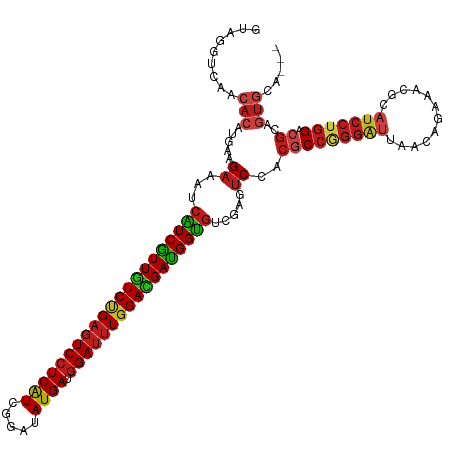
>dm3.chrX 17835077 117 - 22422827 GUAGGUCAACACAUGAAGAAAUCAUCAUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACACAUCCUGGACGCAGUGCG--- (((((((.((((((((.....))))(((((((..(((((((((.....)))).)))))..))))))))))).))..)).(((((((((((.......)).))))))).))...))).--- ( -40.40, z-score = -1.69, R) >droSim1.chrX 13886925 117 - 17042790 GUAGGUCAACACAUGAAGAAAUCAUCAUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCG--- (((((((.((((((((.....))))(((((((..(((((((((.....)))).)))))..))))))))))).))..)).(((((((((((.......)).))))))).))...))).--- ( -40.10, z-score = -1.46, R) >droSec1.super_8 136759 117 - 3762037 GUAGGUCAACACAUGAAGAAAUCAUCAUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUAUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCG--- (((((.(.......((.....))(((((((((..(((((((((.....)))).)))))..))))))))).....).)).(((((((((((.......)).))))))).))...))).--- ( -37.10, z-score = -0.98, R) >droYak2.chrX 16453689 117 - 21770863 GUAGGUCAACACAUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCG--- (((((((.((((((((.....))))(((((((..(((((((((.....)))).)))))..))))))))))).))..)).(((((((((((.......)).))))))).))...))).--- ( -39.70, z-score = -1.11, R) >droEre2.scaffold_4690 8160166 117 - 18748788 GUAGGUCAACACAUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUGACAGAAACGCAUCCUGGACGCAGUGCG--- (((((((.((((((((.....))))(((((((..(((((((((.....)))).)))))..))))))))))).))..)).(((((((((((.......)).))))))).))...))).--- ( -39.70, z-score = -1.11, R) >droAna3.scaffold_13335 1536865 117 + 3335858 GUAGGUCAACACAUGAAGAAAUCAUCAUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUAACAGACACGCAUCCAGGACGCAGUGCA--- ....(((.....(((..........((((((((((((((((((.....)))).))))))))))))))(((((.(((((......)))))....))))).)))....)))........--- ( -40.10, z-score = -1.81, R) >dp4.chrXL_group1e 5178218 117 - 12523060 GUAGGUCAACACAUGAAGAAAUCGUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUCGAGUCGACGCCGGGAUUAACUGACACACAUCCUGGACGUUGUGCA--- .....((.((((((((.....))))(((..(((((((((((((.....)))).)))))))))..))))))).))((((((((((((((............))))))).))))).)).--- ( -42.30, z-score = -1.59, R) >droPer1.super_3220 4065 117 - 4900 GUAGGUCAACACAUGAAGAAAUCGUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUCGAGUCGACGCCGGGAUUAACUGACACACAUCCUGGACGUAGUGCA--- (((((((.((((((((.....))))(((..(((((((((((((.....)))).)))))))))..))))))).)).)).((((((((((............))))))).)))..))).--- ( -39.70, z-score = -1.00, R) >droWil1.scaffold_181150 1804185 120 + 4952429 GUAGGUCAACACAUGAAGAAAUCAUCGUUAUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUUGAAUCGACACCAGGACUAACUGAAACGCAUCCUGGACGUAGUAGUACG ((...(((((((((((.....))))(((((((..(((((((((.....)))).)))))..))))))))))))))....)).((((((..............)))))).((((....)))) ( -39.74, z-score = -2.56, R) >droVir3.scaffold_12928 4839939 117 + 7717345 GUAGGUCAACACUUGAAGAAAUCAUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUUGAAUCGACGCCAGGGUUAACUGAAACACAUCCUGGACGCAGUGGA--- ...(((((((((.(((.....))).((((((((((((((((((.....)))).)))))))))))))))))))).)))(.(((((((((............))))))).)))......--- ( -43.20, z-score = -2.80, R) >droGri2.scaffold_15203 1593461 117 - 11997470 GUAGGUCAACACUUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUUGAAUCGACGCCGGGAUUAACUGAAACACAUCCUGGACGCAGUGUA--- ........(((((.((...((.((((((((((..(((((((((.....)))).)))))..))))))))))))...))(.(((((((((............))))))).)))))))).--- ( -40.70, z-score = -2.50, R) >droMoj3.scaffold_6473 2256187 117 + 16943266 GUAGGUCAACACUUGAAGAAAUCAUCGUUGUCCGAGUCCUCAUCGGAUAUGAUGGAUUUGGAUGAUGGUGUUGAAUCGACGCCAGGAUUAACUGAAACGCAUCCUGGACGCAAUGGA--- ...(((((((((.(((.....))).(((..(((((((((((((.....)))).)))))))))..))))))))).)))(.(((((((((............))))))).)))......--- ( -41.40, z-score = -2.79, R) >anoGam1.chrX 17353074 108 + 22145176 GCAGGUCAACAUCUAAAGAAAACUUCGUUGUCCGAAUCGUCGUCCGACAUGAUGGACCUAGAGGACGGGGUCGAGACAACGCCCGGCCUAUUAGGUGAACAAAAUCGU------------ ..(((((....((....))......((((((((((.((((((((((......)))))(....))))))..))).)))))))...)))))....(((.......)))..------------ ( -29.90, z-score = -0.19, R) >consensus GUAGGUCAACACAUGAAGAAAUCAUCGUUGUCUGAGUCCUCAUCGGAUAUGAUGGAUUUGGACGAUGGUGUCGAGUCCACGCCGGGAUUAACAGAAACGCAUCCUGGACGCAGUGCA___ .........(((.....((...(((((((((((((((((((((.....)))).))))))))))))))))).....))..(((((((((............))))))).))..)))..... (-32.44 = -32.92 + 0.49)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:54:34 2011