
| Sequence ID | dm3.chrX |
|---|---|
| Location | 16,281,044 – 16,281,161 |
| Length | 117 |
| Max. P | 0.898040 |

| Location | 16,281,044 – 16,281,161 |
|---|---|
| Length | 117 |
| Sequences | 15 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 85.86 |
| Shannon entropy | 0.31733 |
| G+C content | 0.52909 |
| Mean single sequence MFE | -44.07 |
| Consensus MFE | -27.41 |
| Energy contribution | -26.24 |
| Covariance contribution | -1.17 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.62 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.898040 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
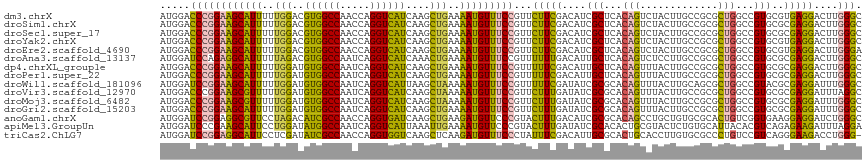
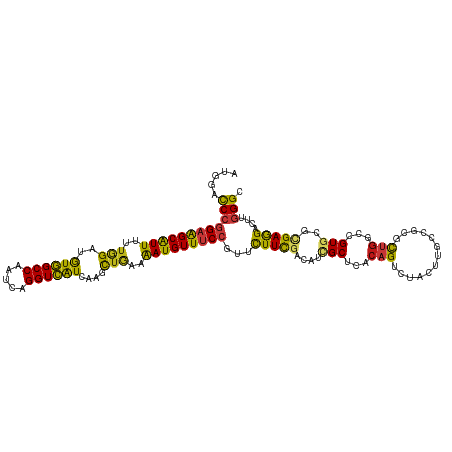
>dm3.chrX 16281044 117 + 22422827 AUGGACCCGGAAGCAUUUUUGGACGUGGCCAACCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUCGACAUCGCUCACAGUCUACUUGCCGCGCUGGCCGUGCGUGAGGACUUGGGC .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...)))).....(((((.((((..((..(.((((.....)))))..))))))))))) ( -45.50, z-score = -2.22, R) >droSim1.chrX 12544024 117 + 17042790 AUGGACCCGGAAGCAUUUUUGGACGUGGCCAACCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUCGACAUCGCUCACAGUCUACUUGCCGCGCUGGCCGUGCGCGAGGACUUGGGC .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...)))).....(((((.((((..(((((.((((.....)))))))))))))))))) ( -47.70, z-score = -2.72, R) >droSec1.super_17 100943 117 + 1527944 AUGGACCCGGAAGCAUUUUUGGACGUGGCCAACCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUCGACAUCGCUCACAGUCUACUUGCCGCGCUGGCCGUGCGCGAGGACUUGGGC .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...)))).....(((((.((((..(((((.((((.....)))))))))))))))))) ( -47.70, z-score = -2.72, R) >droYak2.chrX 10401018 117 + 21770863 AUGGACCCGGAAGCAUUUUUGGACGUGGCCAACCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUCGACAUCGCUCACAGUCUACUUGCCGCGCUGGCCGUGCGUGAGGACUUGGGC .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...)))).....(((((.((((..((..(.((((.....)))))..))))))))))) ( -45.50, z-score = -2.22, R) >droEre2.scaffold_4690 7932240 117 - 18748788 AUGGACCCGGAAGCAUUUUUGGACGUGGCCAACCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUCGACAUCGCUCACAGUCUACUUGCCGCGCUGGCCGUGCGUGAGGACUUGGGA .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...))))......((((.((((..((..(.((((.....)))))..)))))))))). ( -43.10, z-score = -1.54, R) >droAna3.scaffold_13137 759190 117 - 1373759 AUGGAUCCAGAGGCAUUUUUAGACGUGGCCAAUCAGGUCAUCAAACUGAAAAUGUUUCCGUUUUUUGACAUUGCUCACAGUCUCCUUGCCGCGCUGGCCGUGCGCGAGGACUUGGGC .........(((((((((((((..((((((.....))))))....))))))))))))).(((....)))...(((((.((..(((((((.((((.....)))))))))))))))))) ( -41.60, z-score = -2.04, R) >dp4.chrXL_group1e 9908664 117 + 12523060 AUGGACCCGGAAGCAUUUUUGGAUGUGGCCAAUCAGGUCAUCAAGCUGAAAAUGUUUCCGUUUUUCGACAUUGCUCACAGUUUACUUGCCGCGCUGGCCGUGCGCGAGGACUUGGGC .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...)))).....(((((.((((..(((((.((((.....)))))))))))))))))) ( -45.80, z-score = -2.73, R) >droPer1.super_22 1361744 117 - 1688296 AUGGACCCGGAAGCAUUUUUGGAUGUGGCCAAUCAGGUCAUCAAGCUGAAAAUGUUUCCGUUUUUCGACAUUGCUCACAGUUUACUUGCCGCGCUGGCCGUGCGCGAGGACUUGGGC .((((..((((((((((((..(..((((((.....))))))....)..))))))))))))...)))).....(((((.((((..(((((.((((.....)))))))))))))))))) ( -45.80, z-score = -2.73, R) >droWil1.scaffold_181096 5946394 117 - 12416693 AUGGAUCCGGAAGCAUUUUUGGAUGUGGCCAAUCAGGUCAUUAAGCUAAAAAUGUUUCCGUUUUUCGAUAUCGCGCACAGUUUACUUGCAGCGCUGGCCGUACGCGAGGAUUUGGGC ..((((((((((((((((((((..((((((.....))))))....))))))))))))))...........(((((.((.((......)).((....)).)).))))))))))).... ( -42.60, z-score = -3.00, R) >droVir3.scaffold_12970 4304473 117 - 11907090 AUGGACCCGGAAGCGUUUUUGGAUGUGGCCAAUCAGGUCAUCAAGCUAAAAAUGUUUCCGUUCUUUGAUAUCGCGCACAGUUUACUUGCCGCGCUGGCCGUGCGCGAGGAUUUAGGC .((((.((((((((((((((((..((((((.....))))))....))))))))))))))...........((((((((((....)).(((.....))).)))))))))).))))... ( -45.80, z-score = -2.94, R) >droMoj3.scaffold_6482 2096782 117 + 2735782 AUGGACCCGGAAGCGUUUUUGGAUGUGGCCAAUCAGGUCAUCAAGCUAAAAAUGUUUCCGUUCUUUGAUAUCGCGCACAGUUUACUUGCCGCGCUGGCCGUGCGCGAGGAUUUGGGC .....(((((((((((((((((..((((((.....))))))....))))))))))))))...........((((((((((....)).(((.....))).))))))))......))). ( -47.50, z-score = -3.06, R) >droGri2.scaffold_15203 7016324 117 + 11997470 AUGGACCCGGAAGCAUUUUUGGAUGUGGCCAAUCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUUGAUAUCGCGCACAGUUUACUUGCCGCGCUGGCCGUGCGCGAGGAUUUGGGC .....((((((((((((((..(..((((((.....))))))....)..)))))))))))...........((((((((((....)).(((.....))).))))))))......))). ( -47.40, z-score = -2.98, R) >anoGam1.chrX 7194614 117 + 22145176 AUGGAUCCGGAGGCGUUCCUAGACAUCGCCAACCAGGUGAUCAAGCUGAAGAUGUUCCCGUACUUUGACAUCGCGCACAGCCUGCUGUGCGCACUGUCGGUGAAGGAGGAUCUGGGC ..((((((((..(((((..(((..((((((.....))))))....)))..)))))..)).((((..((((..((((((((....))))))))..)))))))).....)))))).... ( -48.40, z-score = -2.35, R) >apiMel3.GroupUn 375571130 117 - 399230636 AUGGAUCCCGAAGCAUUCCUGGAUAUGGCCAAUCAGGUCAUUAAAUUGAAAAUGUUCCCGUACUUUGAUAUCGCACACUGCGUACUCUGUGCAUUACACGUCAGAGAAGAUUUAGGA ................((((((((((((((.....)))))).....................(((((((..(((.....)))......(((.....))))))))))...)))))))) ( -25.20, z-score = 0.46, R) >triCas2.ChLG7 11323043 116 - 17478683 AUGGAUCCGGAGGCAUUCCUCGAUAUCGCCAACCAGGUGGUCAAGCUCAAGAUGUUUCCCUAUUUCGACAUUGCGCACUGCACCUUGUGCGCCCUGUCCGUCAGGGAAGACCUGGG- .(((.....((((....)))).......))).((((((.(((........))).(((((((.....((((..((((((........))))))..))))....))))))))))))).- ( -41.50, z-score = -1.52, R) >consensus AUGGACCCGGAAGCAUUUUUGGAUGUGGCCAAUCAGGUCAUCAAGCUGAAAAUGUUUCCGUUCUUCGACAUCGCUCACAGUCUACUUGCCGCGCUGGCCGUGCGCGAGGACUUGGGC .....((((((((((((..(((..((((((.....))))))....)))..)))))))))...(((((....(((...(((.............)))...)))..)))))....))). (-27.41 = -26.24 + -1.17)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:49:29 2011