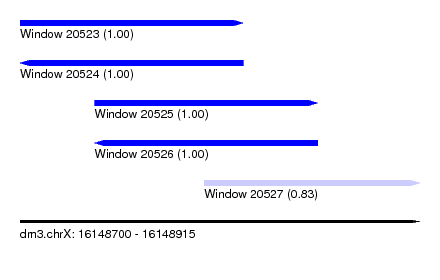
| Sequence ID | dm3.chrX |
|---|---|
| Location | 16,148,700 – 16,148,915 |
| Length | 215 |
| Max. P | 0.998956 |

| Location | 16,148,700 – 16,148,820 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.88 |
| Shannon entropy | 0.07211 |
| G+C content | 0.37012 |
| Mean single sequence MFE | -35.68 |
| Consensus MFE | -36.00 |
| Energy contribution | -35.12 |
| Covariance contribution | -0.87 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.39 |
| Structure conservation index | 1.01 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.34 |
| SVM RNA-class probability | 0.998384 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
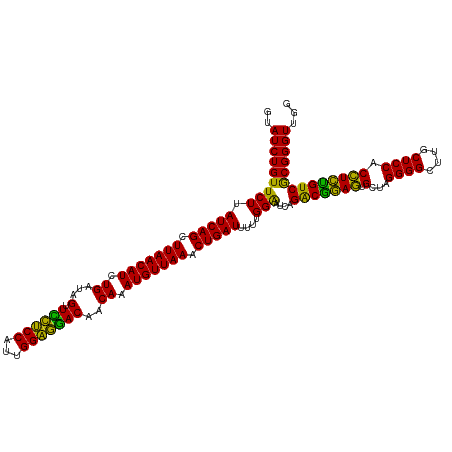
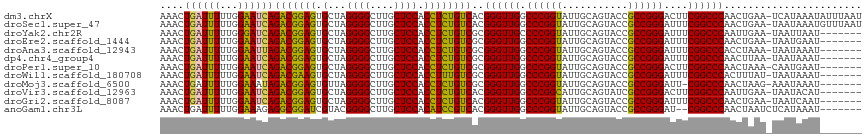
>dm3.chrX 16148700 120 + 22422827 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))) ( -37.10, z-score = -3.77, R) >droSim1.chrX 12469064 120 + 17042790 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))) ( -37.10, z-score = -3.77, R) >droSec1.super_47 197066 120 + 216421 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))) ( -37.10, z-score = -3.77, R) >droYak2.chr2R 6278635 119 + 21139217 AGUUAUCGCUUCUCGGCCUUUUGGCUAAGAUCAA-GUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGGAUCAGA ......(((((((..(((....)))..)))...)-))).....((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))) ( -37.00, z-score = -3.42, R) >droEre2.scaffold_1444 4776 120 - 5501 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))) ( -37.10, z-score = -3.77, R) >droAna3.scaffold_12943 3151857 120 - 5039921 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUUAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))).)))) ( -35.20, z-score = -3.29, R) >dp4.chr4_group4 2460381 120 - 6586962 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))) ( -34.70, z-score = -3.37, R) >droPer1.super_10 406285 120 - 3432795 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))) ( -34.70, z-score = -3.37, R) >droWil1.scaffold_180708 11895397 120 + 12563649 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))) ( -34.70, z-score = -3.37, R) >droMoj3.scaffold_6500 17112638 120 - 32352404 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAAUAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)).)))))) ( -32.80, z-score = -2.90, R) >droVir3.scaffold_12963 18754581 120 - 20206255 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))) ( -34.70, z-score = -3.37, R) >droGri2.scaffold_8087 1359 120 - 4350 AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))).)))) ( -34.20, z-score = -2.98, R) >anoGam1.chr3L 4334920 120 + 41284009 AGGUAUCGCUUCUCGGCCUAAAGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUCUCCCAUUGGGAGACGACAAAUGUUAAACUGAUUUUUGGAAAGAGG (((((((((((((..(((....)))..)))....))))..)))))).((((.(((((.(((((((.((...(.((((((...)))))))..)).))))))).)))))....))))..... ( -37.50, z-score = -2.96, R) >consensus AGUUAUCGCUUCUCGGCCUUAUGGCUAAGAUCAAAGUGUAGUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGA ......(((((((..(((....)))..)))....)))).....((((((((.(((((.(((((((.((...(.((((((...)))))))..)).))))))).)))))....)))).)))) (-36.00 = -35.12 + -0.87)

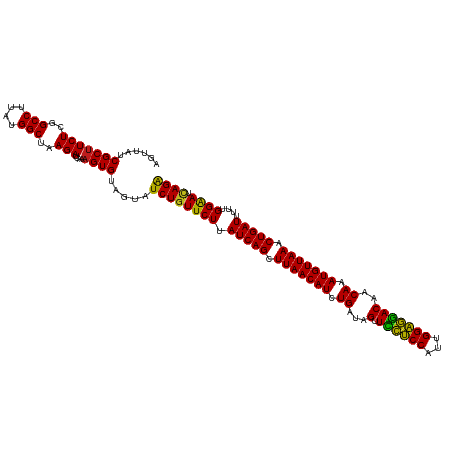
| Location | 16,148,700 – 16,148,820 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.88 |
| Shannon entropy | 0.07211 |
| G+C content | 0.37012 |
| Mean single sequence MFE | -39.35 |
| Consensus MFE | -37.29 |
| Energy contribution | -36.75 |
| Covariance contribution | -0.54 |
| Combinations/Pair | 1.09 |
| Mean z-score | -5.66 |
| Structure conservation index | 0.95 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.35 |
| SVM RNA-class probability | 0.998418 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 16148700 120 - 22422827 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -41.50, z-score = -6.13, R) >droSim1.chrX 12469064 120 - 17042790 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -41.50, z-score = -6.13, R) >droSec1.super_47 197066 120 - 216421 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -41.50, z-score = -6.13, R) >droYak2.chr2R 6278635 119 - 21139217 UCUGAUCCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACAC-UUGAUCUUAGCCAAAAGGCCGAGAAGCGAUAACU ((((.((......((((((((((((((((...((((((...)))))).....))))))))))))))))..)).))))........-....(((..(((....)))..))).......... ( -40.50, z-score = -6.05, R) >droEre2.scaffold_1444 4776 120 + 5501 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -41.50, z-score = -6.13, R) >droAna3.scaffold_12943 3151857 120 + 5039921 UCUAAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU .............((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..))).......... ( -38.30, z-score = -5.50, R) >dp4.chr4_group4 2460381 120 + 6586962 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -39.10, z-score = -5.75, R) >droPer1.super_10 406285 120 + 3432795 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -39.10, z-score = -5.75, R) >droWil1.scaffold_180708 11895397 120 - 12563649 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -39.10, z-score = -5.75, R) >droMoj3.scaffold_6500 17112638 120 + 32352404 UCUAUUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU .............((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..))).......... ( -35.90, z-score = -5.02, R) >droVir3.scaffold_12963 18754581 120 + 20206255 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -39.10, z-score = -5.75, R) >droGri2.scaffold_8087 1359 120 + 4350 UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU ((((.(((.....((((((((((((((((..((.((((...))))..))...))))))))))))))))..))))))).............(((..(((....)))..))).......... ( -36.20, z-score = -4.96, R) >anoGam1.chr3L 4334920 120 - 41284009 CCUCUUUCCAAAAAUCAGUUUAACAUUUGUCGUCUCCCAAUGGGAGAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCUUUAGGCCGAGAAGCGAUACCU .(((..........(((((((((((((((..(((((((...)))))).)...)))))))))))))))(((((.((((........)))).)))))(((....))).)))........... ( -38.30, z-score = -4.54, R) >consensus UCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUACUACACUUUGAUCUUAGCCAUAAGGCCGAGAAGCGAUAACU .............((((((((((((((((...((((((...)))))).....)))))))))))))))).....((((........)))).(((..(((....)))..))).......... (-37.29 = -36.75 + -0.54)

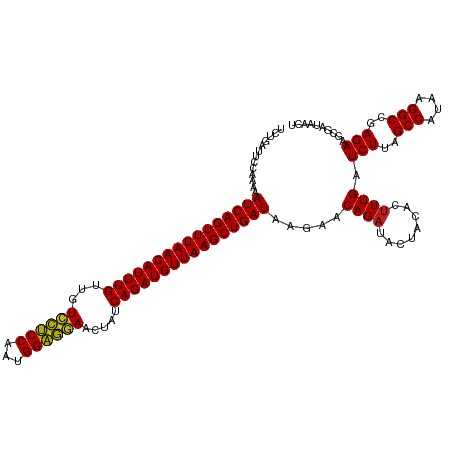
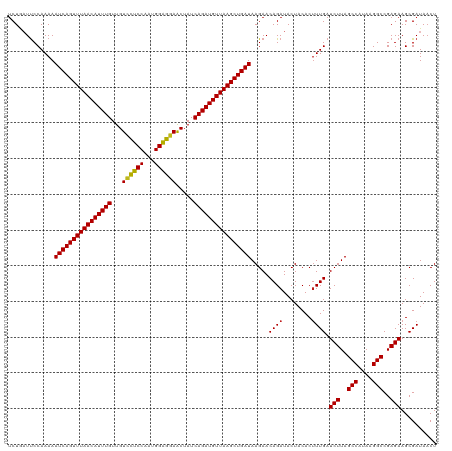
| Location | 16,148,740 – 16,148,860 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.56 |
| Shannon entropy | 0.10188 |
| G+C content | 0.44615 |
| Mean single sequence MFE | -38.91 |
| Consensus MFE | -40.24 |
| Energy contribution | -38.66 |
| Covariance contribution | -1.58 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.68 |
| Structure conservation index | 1.03 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.04 |
| SVM RNA-class probability | 0.997103 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 16148740 120 + 22422827 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGG ..(((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -39.80, z-score = -2.74, R) >droSim1.chrX 12469104 120 + 17042790 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACAGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGG ..(((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -40.50, z-score = -2.96, R) >droSec1.super_47 197106 120 + 216421 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGG ..(((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -39.80, z-score = -2.74, R) >droYak2.chr2R 6278674 120 + 21139217 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGGAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGC ..(((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -39.20, z-score = -2.24, R) >droEre2.scaffold_1444 4816 120 - 5501 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -40.10, z-score = -2.70, R) >droAna3.scaffold_12943 3151897 120 - 5039921 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUUAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...((.(((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -40.10, z-score = -2.88, R) >dp4.chr4_group4 2460421 120 - 6586962 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -37.70, z-score = -2.50, R) >droPer1.super_10 406325 120 - 3432795 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -37.70, z-score = -2.50, R) >droWil1.scaffold_180708 11895437 120 + 12563649 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGAAGUGCUAGGGGCUUGCUCCACCUUUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -35.40, z-score = -2.24, R) >droMoj3.scaffold_6500 17112678 120 - 32352404 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAAUAGACGGAGUGUUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))....(((((((.((..((((....)))))))))))))))))))... ( -39.30, z-score = -3.30, R) >droVir3.scaffold_12963 18754621 120 - 20206255 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGAACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGG ..(((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -37.40, z-score = -2.54, R) >droGri2.scaffold_8087 1399 120 - 4350 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUUCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGG ..(((((((((.(((((.(((((((.((...(((.((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... ( -36.90, z-score = -2.14, R) >anoGam1.chr3L 4334960 120 + 41284009 GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGCUCUCCCAUUGGGAGACGACAAAUGUUAAACUGAUUUUUGGAAAGAGGCGGAUCGUACGGGGCUUGCUCCACAUCCGUCACGGGUUGG ..(((((((((.(((((.(((((((.((...(.((((((...)))))))..)).))))))).)))))....)))....(((((((.((..((((....)))))))))))))))))))... ( -41.90, z-score = -3.27, R) >consensus GUAUCUGUUCUUAUCAGCUUAACAUCUGAUAGUUCCUCCAUUGGAGGACAACAAAUGUUAAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGG ..(((((((((.(((((.(((((((.((...(.((((((...)))))))..)).))))))).)))))....)))....(((((((.(...((((....)))).))))))))))))))... (-40.24 = -38.66 + -1.58)

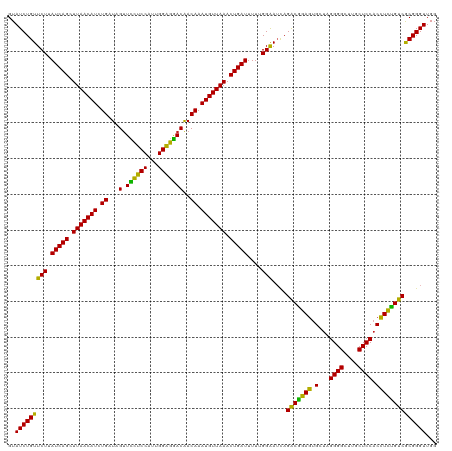
| Location | 16,148,740 – 16,148,860 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.56 |
| Shannon entropy | 0.10188 |
| G+C content | 0.44615 |
| Mean single sequence MFE | -40.29 |
| Consensus MFE | -36.73 |
| Energy contribution | -35.78 |
| Covariance contribution | -0.96 |
| Combinations/Pair | 1.18 |
| Mean z-score | -5.02 |
| Structure conservation index | 0.91 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.57 |
| SVM RNA-class probability | 0.998956 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 16148740 120 - 22422827 CCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(((....(((.((......)))))..)))...(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -43.10, z-score = -5.47, R) >droSim1.chrX 12469104 120 - 17042790 CCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCUGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(((....(((.((......)))))..)))..((((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..))))))))). ( -43.70, z-score = -5.33, R) >droSec1.super_47 197106 120 - 216421 CCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(((....(((.((......)))))..)))...(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -43.10, z-score = -5.47, R) >droYak2.chr2R 6278674 120 - 21139217 GCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUCCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC ...((((......).)))((((...((.....)).))))(((((.((......((((((((((((((((...((((((...)))))).....))))))))))))))))..)).))))).. ( -41.60, z-score = -5.06, R) >droEre2.scaffold_1444 4816 120 + 5501 CCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC ...((((......).)))((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -42.60, z-score = -5.46, R) >droAna3.scaffold_12943 3151897 120 + 5039921 CCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUAAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(.((...((..((((...((.....)).))))..))...)))....((((((((((((((((...((((((...)))))).....))))))))))))))))............ ( -39.40, z-score = -4.89, R) >dp4.chr4_group4 2460421 120 + 6586962 CCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC ...((((......).)))((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -40.20, z-score = -5.30, R) >droPer1.super_10 406325 120 + 3432795 CCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC ...((((......).)))((((...((.....)).))))(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -40.20, z-score = -5.30, R) >droWil1.scaffold_180708 11895437 120 - 12563649 CCAACCCGCGACAAAGGUGGAGCAAGCCCCUAGCACUUCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......((.....(((.((......))))).)).....(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -38.20, z-score = -4.91, R) >droMoj3.scaffold_6500 17112678 120 + 32352404 CCAACCCGCGACAGAGGUGGAGCAAGCCCCUAACACUCCGUCUAUUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .........(((.(((((((.(....).))..)).))).)))...........((((((((((((((((...((((((...)))))).....))))))))))))))))............ ( -35.40, z-score = -4.43, R) >droVir3.scaffold_12963 18754621 120 + 20206255 CCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUUCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(((....(((.((......)))))..)))...(((((.(((.....((((((((((((((((...((((((...)))))).....))))))))))))))))..)))))))).. ( -40.70, z-score = -5.34, R) >droGri2.scaffold_8087 1399 120 + 4350 CCAACCCGUGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGAAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(((....(((.((......)))))..)))...(((((.(((.....((((((((((((((((..((.((((...))))..))...))))))))))))))))..)))))))).. ( -37.80, z-score = -4.51, R) >anoGam1.chr3L 4334960 120 - 41284009 CCAACCCGUGACGGAUGUGGAGCAAGCCCCGUACGAUCCGCCUCUUUCCAAAAAUCAGUUUAACAUUUGUCGUCUCCCAAUGGGAGAGCUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .......(.((.(((.(((((....((...))....))))).))).)))....((((((((((((((((..(((((((...)))))).)...))))))))))))))))............ ( -37.80, z-score = -3.79, R) >consensus CCAACCCGCGACAGAGGUGGAGCAAGCCCCUAGCACUCCGUCUGAUUCCAAAAAUCAGUUUAACAUUUGUUGUCCUCCAAUGGAGGAACUAUCAGAUGUUAAGCUGAUAAGAACAGAUAC .........(((.(((((((.(....).))..)).))).)))...........((((((((((((((((...((((((...)))))).....))))))))))))))))............ (-36.73 = -35.78 + -0.96)

| Location | 16,148,799 – 16,148,915 |
|---|---|
| Length | 116 |
| Sequences | 12 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 90.94 |
| Shannon entropy | 0.19629 |
| G+C content | 0.51690 |
| Mean single sequence MFE | -37.58 |
| Consensus MFE | -35.56 |
| Energy contribution | -35.07 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.22 |
| Structure conservation index | 0.95 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.829499 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
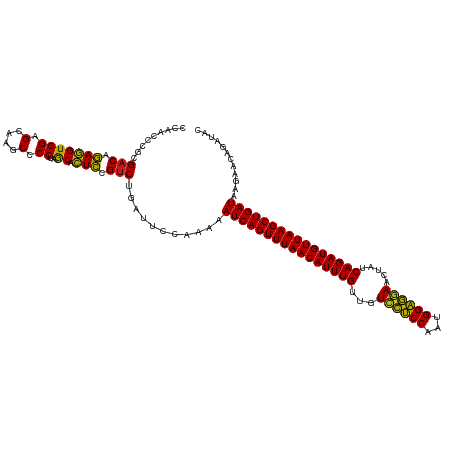
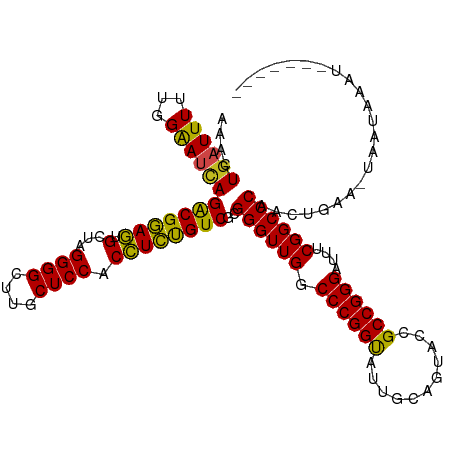
>dm3.chrX 16148799 116 + 22422827 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGACUUCGGCCCAACUGAA-UCAUAAAUAUUUAAU ....((((((((((.....(((((((.(...((((....)))).))))))))..(((....)))(((((....)))))(((((....)))))))))..)))-)))............ ( -38.00, z-score = -1.40, R) >droSec1.super_47 197165 116 + 216421 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAACUGAA-UAAUAAAUGUUUAAU ((((..(((((..(.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..)..))-.)))....))))... ( -37.10, z-score = -1.01, R) >droYak2.chr2R 6278733 109 + 21139217 AAACUGAUUUUUGGGAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGCCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAAUUGAA-UAAUUAAU------- ....(((((.....)))))(((((((.(...((((....)))).))))))))..(((((((((((((((....))))))..)))....)))))).......-........------- ( -36.10, z-score = -0.50, R) >droEre2.scaffold_1444 4875 109 - 5501 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAACUGAA-UAAUGAAU------- ....((((((...))))))(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...))))))).......-........------- ( -36.30, z-score = -0.57, R) >droAna3.scaffold_12943 3151956 109 - 5039921 AAACUGAUUUUUGGAAUUAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCACCUAAA-UAAUAAAU------- .........(((((.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..)))))-........------- ( -36.30, z-score = -0.81, R) >dp4.chr4_group4 2460480 109 - 6586962 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAACUUAA-UAAUAAAU------- ....((((((...))))))(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...))))))).......-........------- ( -36.30, z-score = -0.93, R) >droPer1.super_10 406384 109 - 3432795 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGACUUCGGCCCAACUAAA-CAAUGAAU------- ....((((((...))))))(((((((.(...((((....)))).))))))))(((((....)))(((((....))))))).(((.(....)))).......-........------- ( -36.70, z-score = -0.71, R) >droWil1.scaffold_180708 11895496 109 + 12563649 AAACUGAUUUUUGGAAUCAGACGAAGUGCUAGGGGCUUGCUCCACCUUUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCACUUUAU-UAAUAAAU------- ...(((((((...)))))))..((((((....(((((.(((((((....)).).)))).)))))(((((....)))))(((((....))))).))))))..-........------- ( -34.80, z-score = -0.90, R) >droMoj3.scaffold_6500 17112737 108 - 32352404 AAACUGAUUUUUGGAAAUAGACGGAGUGUUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUU-CGGCCCAACUAAG-AAAUAAAU------- .......(((((((.....(((((((.((..((((....)))))))))))))..((((((.((((((...........))))))...-))))))..)))))-))......------- ( -40.90, z-score = -2.39, R) >droVir3.scaffold_12963 18754680 109 - 20206255 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGCAUUGCAGUAUCGCCGGGACUUCGGCCCAAUUGAA-UAAUACAU------- ....((((((...))))))(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...))))))).......-........------- ( -38.40, z-score = -1.61, R) >droGri2.scaffold_8087 1458 109 - 4350 AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAACUGAA-UAAUCAAU------- ....(((((((..(.....(((((((.(...((((....)))).))))))))..(((((((((((((...........))))))...)))))))..)..))-.)))))..------- ( -39.70, z-score = -1.86, R) >anoGam1.chr3L 4335019 108 + 41284009 AAACUGAUUUUUGGAAAGAGGCGGAUCGUACGGGGCUUGCUCCACAUCCGUCACGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAU--CGGCCCAACUAAUCUCAUAAAU------- ....(((...((((.....(((((((.((..((((....)))))))))))))..(((((((((((((...........)))))).)--))))))..))))..))).....------- ( -40.40, z-score = -1.97, R) >consensus AAACUGAUUUUUGGAAUCAGACGGAGUGCUAGGGGCUUGCUCCACCUCUGUCGCGGGUUGGCCCGGUAUUGCAGUACCGCCGGGAUUUCGGCCCAACUGAA_UAAUAAAU_______ ....((((((...))))))(((((((.(...((((....)))).))))))))..((((((.((((((...........))))))....))))))....................... (-35.56 = -35.07 + -0.48)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:49:07 2011