
| Sequence ID | dm3.chrX |
|---|---|
| Location | 14,992,609 – 14,992,747 |
| Length | 138 |
| Max. P | 0.632399 |

| Location | 14,992,609 – 14,992,747 |
|---|---|
| Length | 138 |
| Sequences | 11 |
| Columns | 138 |
| Reading direction | reverse |
| Mean pairwise identity | 71.06 |
| Shannon entropy | 0.62129 |
| G+C content | 0.59149 |
| Mean single sequence MFE | -51.83 |
| Consensus MFE | -19.08 |
| Energy contribution | -18.04 |
| Covariance contribution | -1.04 |
| Combinations/Pair | 1.57 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.632399 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 14992609 138 - 22422827 UUUCGAACCGGCGGCUAUGGUCUUGCCCGUCGCCGUGGAUACCGUGUCAGAUUCCUGUUCAAUGCUUGCCAUCUUAAUGGUGGCCUCCAGUCGCAUGGCCAACGGAGUGGCCAACUCGAAACUGGGUGCAGCCGCUCC ........(((((((...((.....)).)))))))(((...(((((((((....)))..........((((((.....))))))........)))))))))..((((((((..(((((....)))))...)))))))) ( -50.90, z-score = -0.98, R) >droEre2.scaffold_4690 6729045 132 + 18748788 CUUCGAACCGGCGGCUAUGGUCUUGCC------CGUGGAUCCCGUGUCCGAUUCCUGUUCAAUGCUCGCCAUCUUAAUGGUGGCCUCCAGUCGCAUGGCCAACGGCGUGGCCAACUCGAAACUGGGUGCAGCCGCUCC .........(((((((.(((((.((((------..(((...(((((..(((((...((.....))..((((((.....))))))....)))))))))))))..)))).)))))(((((....)))))..))))))).. ( -47.90, z-score = -0.63, R) >droYak2.chrX 9183684 132 - 21770863 CUUCGAACUGGCGGCUAUGGUCUUUCC------UGUGGAUCCCGUAUCAGAUUCCUGUUCAAUGCUCGCCAUCUUAAUGGUGGCCUCCAGUCGCAUGGCCAACGGAGUGGCCAACUCGAAAUUGGGUGCAGCCGCUCC ...(((.((((.((((((((....(((------...)))..))))).(((....))).........((((((....))))))))).)))))))..........((((((((..(((((....)))))...)))))))) ( -44.60, z-score = -0.79, R) >droSec1.super_32 439244 138 - 562834 UUUCGAACCGGCGGCUAUGGUCUUGGCCGUCGCCGUGGAUACCGUGUCAGAUUCCUGUUCAAUGCUUGCCAUCUUAAUGGUGGCCUCCAGUCGCAUGGCCAACGGAGUGGCCAACUCGAAACUGGGUGCAGCCGCUCC .........((((((((......))))))))(((((((((..(((..(((....)))....)))...((((((.....)))))).....))).))))))....((((((((..(((((....)))))...)))))))) ( -55.30, z-score = -1.84, R) >droPer1.super_28 728537 133 + 1111753 CUUGGCACUUGCGGCUAUGUUCUUGCCAGCUACCGUGGAGCCUGUGUCCGUGUCCUGUUCGACGCUGGCCAUCUUGAUGGUGGCCUCCAGACGCAUGGCCAGCGGCGUGGCCAGCUCAAA---GGCCGC--CUGUCCC ...((.((..((((((.((..((.((((....((((.(.(((((((((.(((((......))))).(((((((.....)))))))....)))))).)))).))))..)))).))..))..---))))))--..)))). ( -59.70, z-score = -1.93, R) >dp4.Unknown_group_716 9170 133 - 19089 CUUGGCACUUGCGGCUAUGUUCUUGCCAGCUAUCGUGGAGCCUGUGUCCGUGUCCUGUUCGACGCUGGCCAUCUUGAUGGUGGCCUCCAGACGCAUGGCCAGCGGCGUGGCCAGCUCAAA---GGCCGC--CUGUCCC ...((.((..((((((.((..((.((((((...(((.(.(((((((((.(((((......))))).(((((((.....)))))))....)))))).)))).))))).)))).))..))..---))))))--..)))). ( -58.60, z-score = -1.78, R) >droWil1.scaffold_180777 2326693 130 + 4753960 ------UUUGUUGGCUUUUGGUGUUACAACUGUGACUGCCGCUGCAUCGGACUCAGCCUCAAUGCUGGCCAUUUUAAUGGUCGCCUCUAAGCGCAUGGCCAGCGGUGUAGCCAUUUCGAA--UGGUUGCUGCUGUGCC ------.......((...(((((((((....))))).))))..))...((.(.((((.....((((((((((.......(.(((......))))))))))))))..((((((((.....)--))))))).)))).))) ( -45.51, z-score = -0.77, R) >droVir3.scaffold_13042 1848536 124 - 5191987 ---UUUGCUGCUGGGUAUGGUCUUGCCUGUGACCGUGGAGGUUGUAUCCGAUUCGGCCUCCAGGCUGGCCAUCUUAAUUGUCGCCUCGAGACGCAUGGCCAGCGGUGUGGCCAGCUCAAAGGCACCC----------- ---..((((..(((((.(((((.((((........(((((((((.........))))))))).(((((((((......((((.......)))).))))))))))))).))))))))))..))))...----------- ( -53.80, z-score = -1.97, R) >droMoj3.scaffold_6359 1759119 124 - 4525533 ---UUUGUUGCUGGCUAUGCUCUUGCCAGUCACCGUGGAUGUUGUAUCCGACUCCGCCUCCAGACUGGCCAUCUUGAUGGUCGCCUCGAGUCGCAUAGCCAGUGGCGUGGCCAGCUCAAAGGCGCCC----------- ---...((..((((((((((....(((((((...(((((.((((....))))))))).....)))))))...(((((.((...)))))))..))))))))))..))..(((..(((....)))))).----------- ( -55.70, z-score = -3.73, R) >droGri2.scaffold_14853 3224935 118 - 10151454 ---CUUGCUGCUGGCUAUAGUCUUGCCGGUU------GAGGUUGUAUCAGAUUCGGCUUCCAGGCUGGCCAUUUUUAUGGUCGCCUCCAGUCGCAUGGCCAGCGGGGUGGCCAAUUCAAAGGGGCCC----------- ---....(((((((((((.(.((.(((((((------((.......))))..)))))....((((.((((((....))))))))))..)).)..)))))))))))...((((..........)))).----------- ( -47.20, z-score = -1.03, R) >droSim1.chrX 11569180 138 - 17042790 UUUCGAACCGGCGGCUAUGGUCUUGCCCGUCGCCGUGGAUACCGUGUCAGAUUCCUGUUCAAUGCUUGCCAUCUUAAUGGUGGCCUCCAGUCGCAUGGCCAACGGAGUGGCCAACUCGAAACUGGGUGCAGCCGCUCC ........(((((((...((.....)).)))))))(((...(((((((((....)))..........((((((.....))))))........)))))))))..((((((((..(((((....)))))...)))))))) ( -50.90, z-score = -0.98, R) >consensus _UUCGAACUGGCGGCUAUGGUCUUGCCAGUCACCGUGGAUCCUGUGUCAGAUUCCUGUUCAAUGCUGGCCAUCUUAAUGGUGGCCUCCAGUCGCAUGGCCAGCGGAGUGGCCAACUCGAAACUGGCUGC_GCCGCUCC ............((((((.((((.............)))).((((.....................((((((....))))))(((...........)))..)))).)))))).......................... (-19.08 = -18.04 + -1.04)

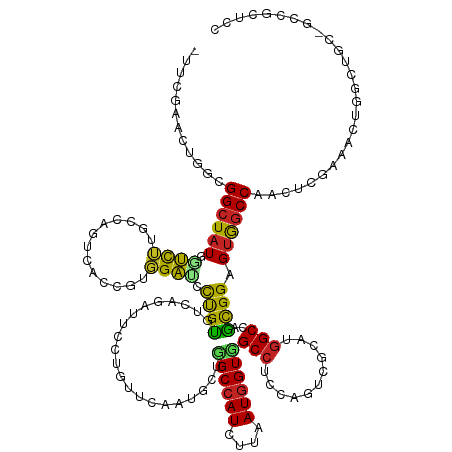

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:45:21 2011