
| Sequence ID | dm3.chrX |
|---|---|
| Location | 14,683,909 – 14,684,029 |
| Length | 120 |
| Max. P | 0.700705 |

| Location | 14,683,909 – 14,684,029 |
|---|---|
| Length | 120 |
| Sequences | 13 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.96 |
| Shannon entropy | 0.30949 |
| G+C content | 0.60962 |
| Mean single sequence MFE | -47.92 |
| Consensus MFE | -34.78 |
| Energy contribution | -34.02 |
| Covariance contribution | -0.75 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.73 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.700705 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 14683909 120 - 22422827 UAUAACGCGCCGGACGUGGACGACACUUCGGUGUCCAGCCGGGCCAGCUCCCGCCUGCUUAGCCUGGACUCGCUGAGCGGCCUCUACGACUGCGAUCUGGACUCCAAGCACGAGCUGGCC ......((((((((.(((.....))))))))))).......(((((((((..(((.(((((((........)))))))))).........(((....(((...))).))).))))))))) ( -50.20, z-score = -1.58, R) >anoGam1.chrX 8916190 120 + 22145176 UACAACGCGCCGGACGUGGACGACACCUCGAUCUCGUCCCGCGCCAGCAGCCGGCUGCUCAGCCUCGACUCGCUGAGCGGGCUGUACGACUGUGAUCUGGACUCGCGCCACGAGAUGGCG ......((((.(((((.((.(((....))).)).))))).))))..((((((...((((((((........))))))))))))))......((((.......))))((((.....)))). ( -50.40, z-score = -0.87, R) >droGri2.scaffold_15203 3482410 120 - 11997470 UACAAUGCGCCCGAUGUGGAUGACACAUCAGUGUCGAGUCGGGCCAGUUCGCGGCUGCUCAGCCUGGAUUCACUCAGCGGCUUAUACGAUUGUGAUUUGGACUCCAAGCAUGAGCUGGCC .....((.(((((((.....((((((....)))))).)))))))))......(((((((((((.(((((((((.(((((.......)).))).)...)))).)))).)).))))).)))) ( -44.50, z-score = -1.20, R) >droMoj3.scaffold_6473 7188215 120 + 16943266 UAUAAUGCGCCCGAUGUGGAUGACACCUCGGUGUCGAGUCGGGCCAGCUCGCGCCUGCUCAGCCUGGACUCGCUCAGCGGUCUUUACGAUUGUGAUUUGGAUUCCAAGCAUGAGCUGGCU .....((.(((((((.....((((((....)))))).)))))))))......(((.(((((((.((((.((.(...((((((.....)))))).....))).)))).)).))))).))). ( -49.90, z-score = -2.17, R) >droVir3.scaffold_12970 11499486 120 - 11907090 UAUAAUGCGCCCGAUGUGGAUGACACCUCGGUGUCGAGUCGGGCCAGCUCGCGCCUGCUCAGCCUGGACUCGCUAAGCGGGCUGUAUGAUUGUGAUUUGGACUCAAAGCAUGAGCUGGCC ......(((((((((.....((((((....)))))).)))))))(((((((((((((((.(((........))).))))))).))......(((.((((....)))).))))))))))). ( -51.40, z-score = -2.54, R) >droWil1.scaffold_181150 854763 120 + 4952429 UACAAUGCACCCGAUGUGGAUGAUACAUCUGUAUCGAGUCGUGCCAGUUCACGUUUGCUAAGCCUCGAUUCAUUGAGCGGUCUCUAUGAUUGUGAUUUGGACUCAAAGCAUGAGUUGGCC ....(((.((.(((((..((((...))))..))))).)))))((((..(((.((((.((((..(.((((.(((.(((....))).))))))).)..)))).....)))).)))..)))). ( -36.10, z-score = -1.21, R) >droPer1.super_17 396709 120 + 1930428 UACAAUGCCCCCGAUGUGGAUGACACCUCAGUGUCGAGUCGCGCCAGCUCUCGUCUGUUAAGCCUGGACUCGCUGAGCGGUCUGUACGAUUGUGAUCUGGACUCCAAGCAUGAGCUGGCC ...............((((.((((((....))))))..))))((((((((..(..((...((.(..((.((((....((.......))...))))))..).)).))..)..)))))))). ( -38.60, z-score = -0.05, R) >dp4.chrXL_group1e 7931599 120 + 12523060 UACAAUGCCCCCGAUGUGGAUGACACCUCAGUGUCGAGUCGCGCCAGCUCUCGUCUGUUAAGCCUGGACUCGCUGAGCGGUCUGUACGAUUGUGAUCUGGACUCCAAGCAUGAGCUGGCC ...............((((.((((((....))))))..))))((((((((..(..((...((.(..((.((((....((.......))...))))))..).)).))..)..)))))))). ( -38.60, z-score = -0.05, R) >droAna3.scaffold_12948 199819 120 + 692136 UACAACGCUCCGGACGUGGACGACACAUCGGUGUCCAGCCGGGCCAGCUCUCGCCUGCUCAGCCUGGACUCGCUGAGCGGCCUCUACGAUUGCGACAUGGACUCCAAGCACGAGCUGGCC .........((((.(.(((((.((......))))))))))))((((((((..(((.(((((((........)))))))))).........(((....(((...))).))).)))))))). ( -51.00, z-score = -2.02, R) >droEre2.scaffold_4690 6439458 120 + 18748788 UAUAACGCGCCGGACGUGGACGACACUUCGGUGUCCAGCCGGGCCAGCUCCCGCCUGCUCAGCCUGGACUCGCUGAGCGGCCUCUACGACUGCGAUCUGGACUCCAAGCAUGAGCUGGCC ......((((((((.(((.....))))))))))).......(((((((((..(((.(((((((........)))))))))).........(((....(((...))).))).))))))))) ( -52.20, z-score = -1.98, R) >droYak2.chrX 8903409 120 - 21770863 UAUAACGCGCCGGACGUGGACGACACUUCGGUGUCCAGUCGGGCCAGCUCCCGCCUGCUCAGCCUGGACUCGCUGAGCGGCCUCUACGAUUGCGAUCUGGACUCCAAGCAUGAGCUGGCC .....((((.....))))((((((((....)))))..))).(((((((((..((..(((((((........)))))))((..((((.(((....)))))))..))..))..))))))))) ( -53.80, z-score = -2.47, R) >droSec1.super_32 131255 120 - 562834 UAUAACGCGCCGGACGUGGACGACACUUCGGUGUCCAGCCGGGCCAGCUCUCGCCUGCUCAGCCUGGACUCGCUGAGCGGCCUCUACGACUGCGAUCUGGACUCCAAGCACGAGCUGGCC ......((((((((.(((.....))))))))))).......(((((((((..(((.(((((((........)))))))))).........(((....(((...))).))).))))))))) ( -52.80, z-score = -2.08, R) >droSim1.chrX 11334872 120 - 17042790 UAUAACGCGCCGGACGUGGACGACACUUCGGUGUCCAGCCGGGCCAGCUCUCGCCUGCUCAGCCUGGACUCGCUGAGCGGUCUCUACGACUGCGAUCUGGACUCCAAGCACGAGCUGGCC ......((((((((.(((.....))))))))))).......(((((((((((((..(((((((........)))))))((((.....))))))))..(((...))).....))))))))) ( -53.40, z-score = -2.19, R) >consensus UAUAACGCGCCGGACGUGGACGACACCUCGGUGUCGAGUCGGGCCAGCUCUCGCCUGCUCAGCCUGGACUCGCUGAGCGGCCUCUACGAUUGUGAUCUGGACUCCAAGCAUGAGCUGGCC ................(((..(((((....)))))...)))(((((((((.....((((.((.(((((.((((....((.......))...))))))))).))...)))).))))))))) (-34.78 = -34.02 + -0.75)

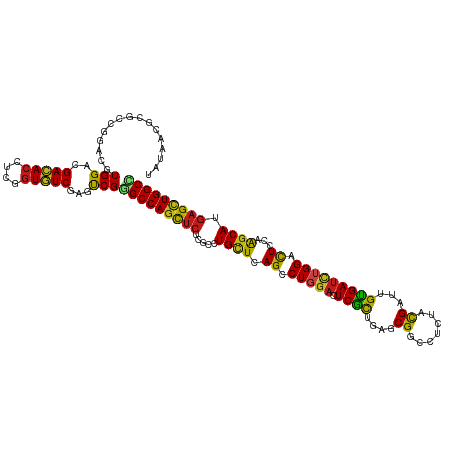
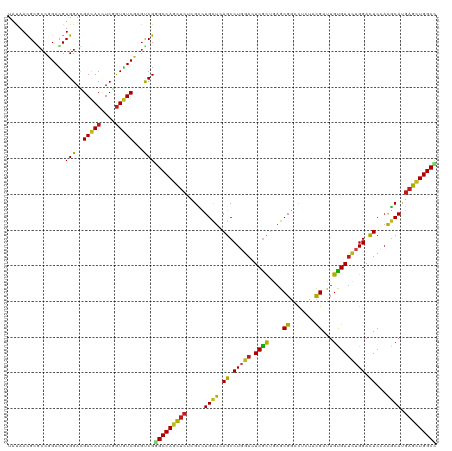
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:44:28 2011