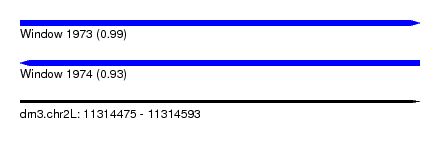
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 11,314,475 – 11,314,593 |
| Length | 118 |
| Max. P | 0.987261 |

| Location | 11,314,475 – 11,314,593 |
|---|---|
| Length | 118 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.19 |
| Shannon entropy | 0.07605 |
| G+C content | 0.26797 |
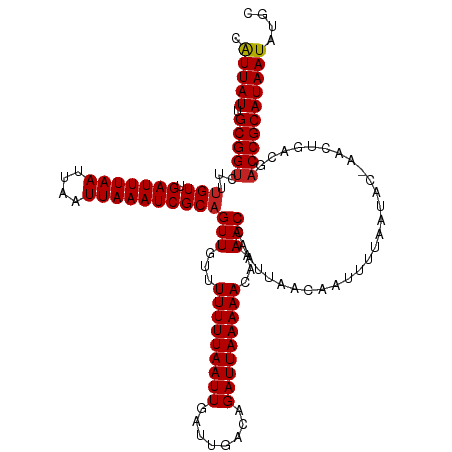
| Mean single sequence MFE | -25.97 |
| Consensus MFE | -22.92 |
| Energy contribution | -22.70 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.88 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.27 |
| SVM RNA-class probability | 0.987261 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 11314475 118 + 23011544 GCAUAUUAUGCGGUCGGACGUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAA-CAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUG ...((((((((((((....(((-((..((((((((.....))))))))((((((((.((.....)))))))))))-))))...(((((((....)))))))......)))))).)))))) ( -27.10, z-score = -3.10, R) >droSim1.chr2L 11118260 119 + 22036055 GCAUAUUAUGCGGUCGGACGUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUG ...((((((((((((....(((-((..((((((((.....))))))))((((((((.((.....)))))))))).)))))...(((((((....)))))))......)))))).)))))) ( -27.10, z-score = -3.11, R) >droSec1.super_3 6711566 119 + 7220098 GCAUAUUAUGCGGUCGGACGUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUG ...((((((((((((....(((-((..((((((((.....))))))))((((((((.((.....)))))))))).)))))...(((((((....)))))))......)))))).)))))) ( -27.10, z-score = -3.11, R) >droYak2.chr2L 7711710 119 + 22324452 GCAUAUUAUGCGGUCGGACGUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUG ...((((((((((((....(((-((..((((((((.....))))))))((((((((.((.....)))))))))).)))))...(((((((....)))))))......)))))).)))))) ( -27.10, z-score = -3.11, R) >droEre2.scaffold_4929 12512942 119 - 26641161 GCAUAUUAUGCGGUCGCACGUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAACG ........((((((((((.(((-((..((((((((.....))))))))((((((((.((.....)))))))))).))))))))(((((((....)))))))......)))))))...... ( -30.60, z-score = -4.30, R) >droAna3.scaffold_12916 12033937 119 - 16180835 GCAUAUUAUGCGGUUGUCAGUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGGCCGCAAUAAUG ...(((((((((((((.(((((-((..((((((((.....))))))))((((((((.((.....)))))))))).))))))))(((((((....)))))))......)))))).)))))) ( -28.10, z-score = -3.51, R) >dp4.chr4_group3 6752543 119 + 11692001 GCAUAUUAUGCGGUCGUCAUUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUC ........((((((((.((.((-((..((((((((.....))))))))((((((((.((.....)))))))))).)))).)))(((((((....)))))))......)))))))...... ( -24.60, z-score = -3.08, R) >droPer1.super_1 8201635 119 + 10282868 GCAUAUUAUGCGGUCGUCAUUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUC ........((((((((.((.((-((..((((((((.....))))))))((((((((.((.....)))))))))).)))).)))(((((((....)))))))......)))))))...... ( -24.60, z-score = -3.08, R) >droWil1.scaffold_181038 225411 119 - 637489 GCAUAUUAUGCGGUUGUCAUUU-GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUC ........(((((((....(((-((................((((...((((((((.((.....))))))))))...))))..(((((((....))))))).))))))))))))...... ( -22.20, z-score = -2.01, R) >droVir3.scaffold_12963 18003293 120 - 20206255 GCAUAUUAUGCGGUUGUCAUUUUGUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAUACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUC ........(((((((.....(((((................((((.((((((((((.((.....)))))))))))).))))..(((((((....))))))).))))))))))))...... ( -24.80, z-score = -2.80, R) >droGri2.scaffold_15126 7120480 120 - 8399593 GCAUAUUAUGCGGUUGUCAUUUUGUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACGGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUC ........((((((((((...((((..((((((((.....))))))))((((((((.((.....)))))))))).)))).)))(((((((....)))))))......)))))))...... ( -22.40, z-score = -1.80, R) >consensus GCAUAUUAUGCGGUCGUCAGUU_GUAUUAAAAUUGUUAAUUGGUUUUGUUUUUAAUCUGUCAAUCAAUUAAAAAAACAACUGCGAUUUAAUUAAUUAAAUCAACAAAGACCGCAAUAAUG ........(((((((........(((.........((((((((((................)))))))))).........)))(((((((....)))))))......)))))))...... (-22.92 = -22.70 + -0.22)

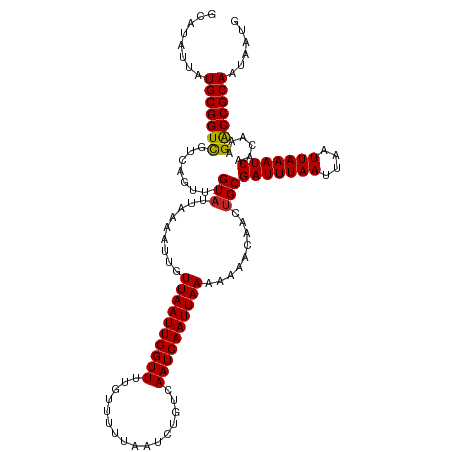
| Location | 11,314,475 – 11,314,593 |
|---|---|
| Length | 118 |
| Sequences | 11 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.19 |
| Shannon entropy | 0.07605 |
| G+C content | 0.26797 |
| Mean single sequence MFE | -25.00 |
| Consensus MFE | -19.43 |
| Energy contribution | -19.53 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.85 |
| Structure conservation index | 0.78 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.929077 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 11314475 118 - 23011544 CAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUG-UUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AACGUCCGACCGCAUAAUAUGC .(((((.((((((..(((((..(((((((((((((..((....)-)..)))))))))))))...(((((((.................))))))).)-))))...))))))))))).... ( -27.83, z-score = -3.82, R) >droSim1.chr2L 11118260 119 - 22036055 CAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AACGUCCGACCGCAUAAUAUGC .(((((.((((((..(((((..(((((((((((((..((....))...)))))))))))))...(((((((.................))))))).)-))))...))))))))))).... ( -26.53, z-score = -3.37, R) >droSec1.super_3 6711566 119 - 7220098 CAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AACGUCCGACCGCAUAAUAUGC .(((((.((((((..(((((..(((((((((((((..((....))...)))))))))))))...(((((((.................))))))).)-))))...))))))))))).... ( -26.53, z-score = -3.37, R) >droYak2.chr2L 7711710 119 - 22324452 CAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AACGUCCGACCGCAUAAUAUGC .(((((.((((((..(((((..(((((((((((((..((....))...)))))))))))))...(((((((.................))))))).)-))))...))))))))))).... ( -26.53, z-score = -3.37, R) >droEre2.scaffold_4929 12512942 119 + 26641161 CGUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AACGUGCGACCGCAUAAUAUGC .(((((.((((((......(((((((....)))))))((((((((.(((((((((........)))))))))........(((((.....)))))))-))).)))))))))))))).... ( -29.30, z-score = -3.35, R) >droAna3.scaffold_12916 12033937 119 + 16180835 CAUUAUUGCGGCCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AACUGACAACCGCAUAAUAUGC .(((((.((((...((((.(((((((....))))))).(((((((.(((((((((........)))))))))........(((((.....)))))))-)))))))))))))))))).... ( -24.10, z-score = -3.04, R) >dp4.chr4_group3 6752543 119 - 11692001 GAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AAAUGACGACCGCAUAAUAUGC .(((((.((((((..(((.(((((((....))))))))))((..(((((((((((........)))))))).........(((((.....)))))..-)))..))))))))))))).... ( -24.80, z-score = -2.82, R) >droPer1.super_1 8201635 119 - 10282868 GAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AAAUGACGACCGCAUAAUAUGC .(((((.((((((..(((.(((((((....))))))))))((..(((((((((((........)))))))).........(((((.....)))))..-)))..))))))))))))).... ( -24.80, z-score = -2.82, R) >droWil1.scaffold_181038 225411 119 + 637489 GAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC-AAAUGACAACCGCAUAAUAUGC .(((((.(((((..(..(((((((((....)))))))..((((((((((((((((........)))))))))...........))))))).......-.))..)..)))))))))).... ( -21.20, z-score = -1.68, R) >droVir3.scaffold_12963 18003293 120 + 20206255 GAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUAUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUACAAAAUGACAACCGCAUAAUAUGC .(((((.(((((..(..(((((((((....)))))))....(((((((...(((((((((..................))))...)))))..)))))))))..)..)))))))))).... ( -22.27, z-score = -2.07, R) >droGri2.scaffold_15126 7120480 120 + 8399593 GAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCCGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUACAAAAUGACAACCGCAUAAUAUGC .(((((.(((((..(..(((((((((....)))))))...(((...(((((((((........)))))))))...))).....................))..)..)))))))))).... ( -21.10, z-score = -1.64, R) >consensus CAUUAUUGCGGUCUUUGUUGAUUUAAUUAAUUAAAUCGCAGUUGUUUUUUUAAUUGAUUGACAGAUUAAAAACAAAACCAAUUAACAAUUUUAAUAC_AACUGACGACCGCAUAAUAUGC .(((((.(((((...(((.(((((((....))))))))))(((...(((((((((........)))))))))...)))............................)))))))))).... (-19.43 = -19.53 + 0.10)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:32:07 2011