| Sequence ID | dm3.chrX |
|---|---|
| Location | 12,530,384 – 12,530,481 |
| Length | 97 |
| Max. P | 0.990369 |

| Location | 12,530,384 – 12,530,481 |
|---|---|
| Length | 97 |
| Sequences | 12 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 72.81 |
| Shannon entropy | 0.56380 |
| G+C content | 0.40033 |
| Mean single sequence MFE | -22.79 |
| Consensus MFE | -16.74 |
| Energy contribution | -16.55 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.73 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.990369 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
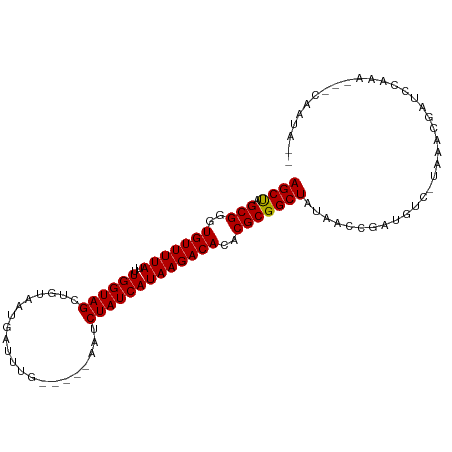
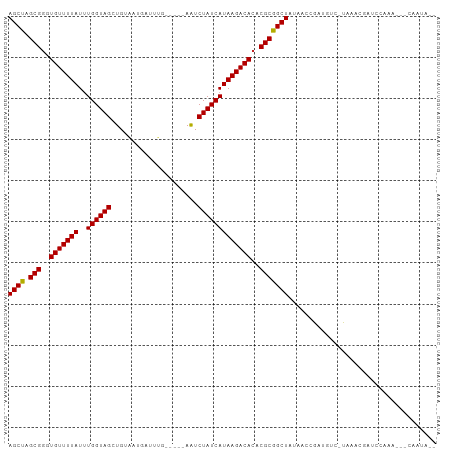
>dm3.chrX 12530384 97 + 22422827 AGCUAGCGGGUGUUUUAUUUGGUAGCUGUAAUGAUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUAACCGUUGUC-UAAGAGCUUCAAAA-UCGAUAAU ((((.(((.((((((((..((((((...(((....)))-----...)))))))))))))).)))))))......((((((-..............-..)))))) ( -23.09, z-score = -1.12, R) >droSim1.chrX_random 3444264 96 + 5698898 AGCUAGCGGGUGUUUUAUUUGGUAGCUGAAUUGAUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUAACCGAUGUC-UAAAUGCUUCAAA--UCGACAUU (((((.((((((...)))))).)))))...((((((((-----((.(.......(((((....(((......))).))))-)....).))))))--)))).... ( -24.50, z-score = -1.89, R) >droSec1.super_21 285109 96 + 1102487 AGCUAGCGGGUGUUUUAUUUGGUAGCUGAAUUGAUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUAACCGAUGUC-UAAAUGCUUCAAA--AUGAUAAU ((((.(((.((((((((..((((((.(..(......).-----.).)))))))))))))).)))))))........((((-.............--..)))).. ( -20.36, z-score = -0.71, R) >droYak2.chrX 6807174 86 + 21770863 AGCUAGCGGGUGUUUUAUUUGGUAGCUGUAAUGAUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUAACCGAUGUCACACAUGCGAC------------- ((((.(((.((((((((..((((((...(((....)))-----...)))))))))))))).))))))).....(((((.....))).))..------------- ( -23.70, z-score = -1.71, R) >droEre2.scaffold_4690 4419736 96 - 18748788 AGCUAGCGGGUGUUUUAUUUGGUAGCUGUAAUGAUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUAACCGAUGUC-UAAACGCUUCAAAGUGCAAUA-- ((((.(((.((((((((..((((((...(((....)))-----...)))))))))))))).)))))))............-.....((........))....-- ( -23.60, z-score = -0.92, R) >droAna3.scaffold_13117 3438255 98 + 5790199 AGCUAGCGGGUGUUUUAUUUGGUAGCUGAUAUCGAAAG-----AAUCUAUCAUAAGACACACGCGGCUAUAAUCGAUGUCCUUAGAGUCUACAAGCUAAAAAU- ((((.(((.((((((((..((((((......((....)-----)..)))))))))))))).)))((((.(((.........))).))))....))))......- ( -25.20, z-score = -1.74, R) >dp4.chrXL_group1a 3316447 96 + 9151740 AGCUAGCGGGUGUUUUAUCUGGUAGCUGUACCCUUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUGACCGAUGCC-CAAACCAUCCAAGAUUCAAUA-- (((((.((((((...)))))).)))))........(((-----(((((.(((((...(......)..)))))..((((..-.....))))..))))))))..-- ( -22.30, z-score = -0.83, R) >droPer1.super_77 64831 96 - 256778 AGCUAGCGGGUGUUUUAUCUGGUAGCUGUACCCUUUUG-----AAUCUAUCAUAAGACACACGCGGCUAUGACCGAUGCC-CAAACCAUCCAAGAUUCAAUA-- (((((.((((((...)))))).)))))........(((-----(((((.(((((...(......)..)))))..((((..-.....))))..))))))))..-- ( -22.30, z-score = -0.83, R) >droWil1.scaffold_181096 1892804 99 + 12416693 AGCUAGCGGGUGUUUUAUUUGGUAGCUGCACUAAAAUUUCAAUAAUCUAUCAUAAGACACACGCGGCUAUGACCGAUGUCUUUCUUAGCCAACAA--UAAU--- .....(((.((((((((..((((((.(.................).)))))))))))))).)))(((((.(((....))).....))))).....--....--- ( -24.33, z-score = -2.09, R) >droMoj3.scaffold_6328 3513362 85 - 4453435 AGCCAGCGGAUGUUUUAUUUGGUAGUUGUAUUUUA--UA---AAGUCUAUCAUAAGACACACGCGGCUAUAACAGAACUGUUCAAUAAGC-------------- ((((.(((..(((((((..(((((((((((...))--))---)...)))))))))))))..)))))))..((((....))))........-------------- ( -21.80, z-score = -2.52, R) >droVir3.scaffold_13042 2658876 85 + 5191987 AGCCAGCGGAUGUUUUAUUUGGUAGUUGCGCUUUA--UU---AAAUCUAUCAUAAGACACACGCGGCUAUAACAGUGCCGUUCAACAAGC-------------- ((((.(((..(((((((..((((((..........--..---....)))))))))))))..)))))))......................-------------- ( -20.99, z-score = -1.14, R) >droGri2.scaffold_15203 5475277 87 + 11997470 AGCCUGCGUAUGUUUUAUUUGGUAGUUGUAACUAAAAUC---GAAUCUAUCAUAAGACACACGCGGCUAUCACAGUUCCGUUCAACAAUC-------------- ((((.((((.(((((((..(((((((((..........)---))..))))))))))))).))))))))......................-------------- ( -21.30, z-score = -2.79, R) >consensus AGCUAGCGGGUGUUUUAUUUGGUAGCUGUAAUGAUUUG_____AAUCUAUCAUAAGACACACGCGGCUAUAACCGAUGUC_UAAACGAUCCAAA___CAAUA__ ((((.(((..(((((((..((((((.....................)))))))))))))..))))))).................................... (-16.74 = -16.55 + -0.19)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:37:48 2011