| Sequence ID | dm3.chrX |
|---|---|
| Location | 12,154,593 – 12,154,724 |
| Length | 131 |
| Max. P | 0.987770 |

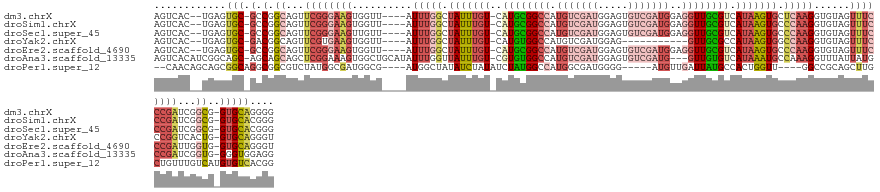
| Location | 12,154,593 – 12,154,724 |
|---|---|
| Length | 131 |
| Sequences | 7 |
| Columns | 140 |
| Reading direction | forward |
| Mean pairwise identity | 74.03 |
| Shannon entropy | 0.49983 |
| G+C content | 0.54488 |
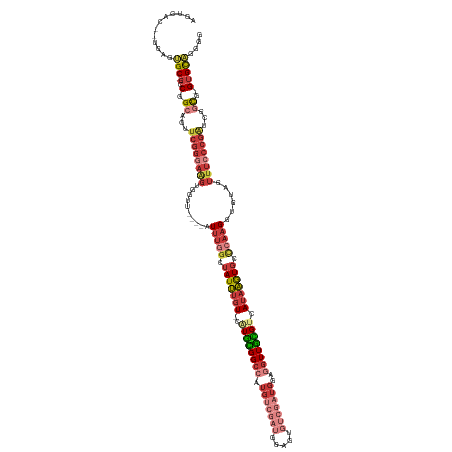
| Mean single sequence MFE | -52.26 |
| Consensus MFE | -20.92 |
| Energy contribution | -23.60 |
| Covariance contribution | 2.68 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.94 |
| Structure conservation index | 0.40 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.29 |
| SVM RNA-class probability | 0.987770 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 12154593 131 + 22422827 AGUCAC--UGAGUGC-GCCGGCAGUUCGGGAAGUGGUU----AUUUGGCUAUUUGU-CAUGCGGCCAUGUCGAUGGAGUGUCGAUGGAGGUUGCGUCAUAAGUGCUCAAGGUGUAGUUUCCCGAUCGGCG-GUGCAGGGG ......--....(((-((((.(...(((((((((....----.((((((.((((((-.((((((((.(((((((.....)))))))..)))))))).)))))))).)))).....)))))))))..).))-))))).... ( -53.00, z-score = -3.11, R) >droSim1.chrX 9360784 131 + 17042790 AGUCAC--UGAGUGC-GCCGGCAGUUCGGGAAGUGGUU----AUUUGGCUAUUUGU-CAUGCGGCCAUGUCGAUGGAGUGUCGAUGGAGGUUGCGUCAUAAGUGCCCAAGGUGUAGUUUCCCGAUCGGCG-GUGCACGGG ......--...((((-((((.(...(((((((((....----.(((((.(((((((-.((((((((.(((((((.....)))))))..)))))))).))))))).))))).....)))))))))..).))-))))))... ( -58.60, z-score = -4.17, R) >droSec1.super_45 172497 131 + 245362 AGUCAC--UGAGUGC-GCCGGCAGUUCGGGAAGUUGUU----AUUUGGCUAUUUGU-CAUGCGGCCAUGUCGAUGGAGUGUCGAUGGAGGUUGCGUCAUAAGUGCCCAAGGUGUAGUUUCCCGAUCGGCG-GUGCACGGG ......--...((((-((((.(...(((((((..((..----.(((((.(((((((-.((((((((.(((((((.....)))))))..)))))))).))))))).)))))...))..)))))))..).))-))))))... ( -59.80, z-score = -4.63, R) >droYak2.chrX 20064878 120 - 21770863 AGUCAC--UGAGUGC-GACGGCAGUUCGUGAAGUGGUU----AUUUGGCUAUUUGU-CAUGUGGCCAUGUCGAUGGAG-----------GUUGCGCCAUAAGUGGCCAAGGUGUAGUUUCCCGGUCACUG-GUGCAGGGU .....(--((.((((-(((..(...(((....((((((----((.((((.....))-)).))))))))..)))..)..-----------)))))))(((.(((((((..((........)).))))))).-))))))... ( -43.90, z-score = -1.74, R) >droEre2.scaffold_4690 4060474 131 - 18748788 AGUCAC--UGAGUGC-GCCGGCAGUUCGGGAAGUGGUU----AUUUGGCUAUUUGU-CAUGCGGCCAUGUCGAUGGAGUGUCGAUGGAGGUUGCGUCAUAAGUGCCCAAGGUGUAGUUUCCCGAUUGGUG-GUGCAGGGU ......--....(((-((((.((((.((((((((....----.(((((.(((((((-.((((((((.(((((((.....)))))))..)))))))).))))))).))))).....)))))))))))).))-))))).... ( -55.50, z-score = -4.16, R) >droAna3.scaffold_13335 2132834 134 - 3335858 AGUCACAUCGGCAGC-AGCAGCAGCUCGGAAAGUGGCUGCAUAUUUGGUUAUUUGU-CGUGUGGCCAUGUCGAUGGAGUGUCGAUG---GUUGUGUCAUAAAUGCCAAAGGUUUAUUAUGCCGAUCGGUG-GGGUGGAGG ..(((((((((((..-.(((((.(((.....))).)))))...((((((.((((((-..((..((((..(((((.....)))))))---))..))..)))))))))))).........)))))))..)))-)........ ( -46.20, z-score = -1.84, R) >droPer1.super_12 1989889 125 - 2414086 --CAACAGCAGCGGCAGGCGGCGUCUAUGGCGAUGGCG----AUGGCUAUAUCUAUAUCUAUGGCCAUGGCGAUGGGG-----AUGUUGAUUAUGCCACUGGUU----GGCCGCAGCUUGCUGUUUGUCAUGUGUCACGG --..(((((((((((((((.((((((((.((.(((((.----((((.(((....))).)))).))))).)).))))).-----...........((((.....)----)))))).)))))))).))))..)))....... ( -48.80, z-score = -0.97, R) >consensus AGUCAC__UGAGUGC_GCCGGCAGUUCGGGAAGUGGUU____AUUUGGCUAUUUGU_CAUGCGGCCAUGUCGAUGGAGUGUCGAUGGAGGUUGCGUCAUAAGUGCCCAAGGUGUAGUUUCCCGAUCGGCG_GUGCAGGGG ............(((.(((((....((((((((..........(((((.(((((((...(((((((..((((((.....))))))...)))))))..))))))).)))))......)))))))))))))....))).... (-20.92 = -23.60 + 2.68)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:36:48 2011