
| Sequence ID | dm3.chrX |
|---|---|
| Location | 12,049,842 – 12,049,962 |
| Length | 120 |
| Max. P | 0.995249 |

| Location | 12,049,842 – 12,049,962 |
|---|---|
| Length | 120 |
| Sequences | 11 |
| Columns | 139 |
| Reading direction | forward |
| Mean pairwise identity | 83.58 |
| Shannon entropy | 0.32571 |
| G+C content | 0.32394 |
| Mean single sequence MFE | -31.46 |
| Consensus MFE | -15.63 |
| Energy contribution | -16.09 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.70 |
| Structure conservation index | 0.50 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.78 |
| SVM RNA-class probability | 0.995249 |
| Prediction | RNA |
| WARNING | Out of training range. z-scores are NOT reliable. |
Download alignment: ClustalW | MAF
>dm3.chrX 12049842 120 + 22422827 UUAUGACAGCCC-AUUUGCAUUUGGA-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCCAAUUAUGCACAAACAC--AAAUUAAUAGACAAAAUAUGCACAUUGCAGCUGUCGCAUCAAUUAAGC------------- ....((((((.(-(..((((((((..-((((((((.....--...(((((((((......))))))))).......--..)))))))).)))...)))))...))..)))))).............------------- ( -28.83, z-score = -3.08, R) >droSec1.super_45 75808 120 + 245362 UUAUGACAGCCC-AUUUGCAUUUGGA-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCCAAUUAUGCACAAACAC--AAAUUAAUAGACAAAAUAUGCACAUUGCAGCUGUCACAUCAAUUAAGC------------- ...(((((((.(-(..((((((((..-((((((((.....--...(((((((((......))))))))).......--..)))))))).)))...)))))...))..)))))))............------------- ( -29.53, z-score = -3.84, R) >droYak2.chrX 19967724 120 - 21770863 UUAUGACAGCAC-AUUUGCAUUUGGA-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCCAAUUAUGCACAAACAC--AAAUUAAUAGACAAAAUAUGCACAUUGCAGCUGUCGCAUCAAUUAAGC------------- ....((((((.(-(..((((((((..-((((((((.....--...(((((((((......))))))))).......--..)))))))).)))...)))))...))..)))))).............------------- ( -28.93, z-score = -3.04, R) >droEre2.scaffold_4690 3965769 120 - 18748788 UUAUGACAGCAC-AUUUGCAUUUGGA-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCCAAUUAUGCACAAACAC--AAAUUAAUAGACAAAAUAUGCACAUUGCAGCUGUCGCAUCAAUUAAGC------------- ....((((((.(-(..((((((((..-((((((((.....--...(((((((((......))))))))).......--..)))))))).)))...)))))...))..)))))).............------------- ( -28.93, z-score = -3.04, R) >droAna3.scaffold_13335 2028123 119 - 3335858 UUAUGACAGGCC-AUUUGCAUUUUGG-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCCAAUUAUGCAUAAACAC--AAAUUAAUAGACAAAAU-UGCACAUUGCAGCUGUCGCAUCAAUUAAGC------------- ....(((((((.-((.(((((((((.-((((((((...(.--((((((((((((......)))))))))))).)..--..)))))))).))))).-)))).)).))..))))).............------------- ( -34.00, z-score = -4.10, R) >droSim1.chrX 9303596 120 + 17042790 UUAUGACAGCCC-AUUUGCAUUUGGA-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCCAAUUAUGCACAAACAC--AAAUUAAUAGACAAAAUAUGCACAUUGCAGCUGUCGCAUCAAUUAAGC------------- ....((((((.(-(..((((((((..-((((((((.....--...(((((((((......))))))))).......--..)))))))).)))...)))))...))..)))))).............------------- ( -28.83, z-score = -3.08, R) >dp4.chrXL_group1a 2779161 122 + 9151740 UUAUGACAGGCC-AUUUGCAUGUUGA-CUGUUAAUCACGA--UUAUGCAUAAUUAAUCCUAAUUAUGCAUAAACACACAAAUUAAUAGACAAAAUAUGCAUAUUGCAGCUGUCGGAUCAAUUAUUC------------- ...((((((((.-((.((((((((..-((((((((.....--(((((((((((((....)))))))))))))........))))))))....)))))))).)).))..))))))............------------- ( -34.72, z-score = -4.54, R) >droPer1.super_12 1883795 122 - 2414086 UUAUGACAGGCC-AUUUGCAUGUUGA-CUGUUAAUCACAA--UUAUGCAUAAUUAAUCCUAAUUAUGCAUAAACACACAAAUUAAUAGACAAAAUAUGCAUAUUGCAGCUGUCGGAUCAAUUAUUC------------- ...((((((((.-((.((((((((..-((((((((.....--(((((((((((((....)))))))))))))........))))))))....)))))))).)).))..))))))............------------- ( -34.72, z-score = -4.72, R) >droWil1.scaffold_180777 3811925 137 + 4753960 UUAUGACAGGCCCAUUUGCAUUUUGGUCUGUUAAUCAUGAGAUUAUGCAUAAUUAAUCCUAAUUAUGCAUAAACAC--AAAUUAAUAGACAAAAUCUGCAUAUUGCUGCCAUUGCAGCUGCCUGUCCCAUCAAUUAGCC ....(((((((.....((((.(((.((((((((((..((.(.(((((((((((((....))))))))))))).).)--).)))))))))).)))..))))....(((((....))))).)))))))............. ( -48.00, z-score = -6.39, R) >droMoj3.scaffold_6359 132224 109 + 4525533 --UUCGAUGCCAAAUUUGCAUUUGG--CUGUUAAUCACGA--UUAUGCAUAAUUAAUUUGCAUUAUGCAUAAACAC--AAAUUAAUAGACAAAAUAUGCAUAUUGCAUCAAUUAGAG---------------------- --...(((((..(((.((((((((.--((((((((...(.--(((((((((((........))))))))))).)..--..)))))))).)))...))))).))))))))........---------------------- ( -29.30, z-score = -3.30, R) >droGri2.scaffold_15081 124559 108 - 4274704 ---UUUAAGCCCAAUUUACAUUUGG--CUGUUAAUCACGA--UUAUGCAUAAUUAAUUCGAUUUAUGCAUAAACAC--AAAUUAAUAGACAAAGUAUGCAUAUUGCGUCAAUUAGUC---------------------- ---.....((.((((.(((.((((.--((((((((...(.--((((((((((..........)))))))))).)..--..)))))))).))))))).....)))).)).........---------------------- ( -20.30, z-score = -1.53, R) >consensus UUAUGACAGCCC_AUUUGCAUUUGGA_CUGUUAAUCACGA__UUAUGCAUAAUUAAUCCCAAUUAUGCAUAAACAC__AAAUUAAUAGACAAAAUAUGCACAUUGCAGCUGUCGCAUCAAUUAAGC_____________ ................(((((......((((((((.......((.(((((((((......))))))))).))........)))))))).......)))))....................................... (-15.63 = -16.09 + 0.45)

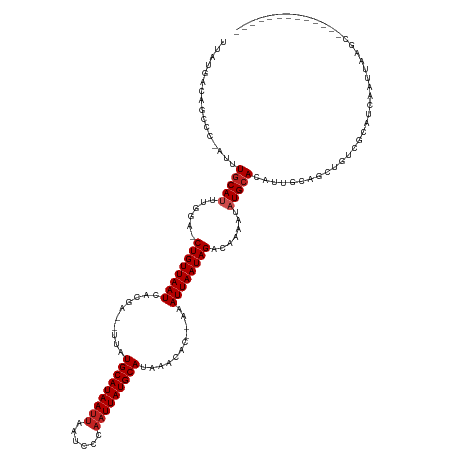

| Location | 12,049,842 – 12,049,962 |
|---|---|
| Length | 120 |
| Sequences | 11 |
| Columns | 139 |
| Reading direction | reverse |
| Mean pairwise identity | 83.58 |
| Shannon entropy | 0.32571 |
| G+C content | 0.32394 |
| Mean single sequence MFE | -35.09 |
| Consensus MFE | -19.56 |
| Energy contribution | -19.80 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.954976 |
| Prediction | RNA |
| WARNING | Out of training range. z-scores are NOT reliable. |
Download alignment: ClustalW | MAF
>dm3.chrX 12049842 120 - 22422827 -------------GCUUAAUUGAUGCGACAGCUGCAAUGUGCAUAUUUUGUCUAUUAAUUU--GUGUUUGUGCAUAAUUGGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-UCCAAAUGCAAAU-GGGCUGUCAUAA -------------((.........))(((((((.((...(((((...(((.((.((((.(.--.((.(((((((((((((....)))))))))))))--.))..))))).))-..))))))))..)-)))))))).... ( -35.30, z-score = -2.29, R) >droSec1.super_45 75808 120 - 245362 -------------GCUUAAUUGAUGUGACAGCUGCAAUGUGCAUAUUUUGUCUAUUAAUUU--GUGUUUGUGCAUAAUUGGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-UCCAAAUGCAAAU-GGGCUGUCAUAA -------------..........((((((((((.((...(((((...(((.((.((((.(.--.((.(((((((((((((....)))))))))))))--.))..))))).))-..))))))))..)-))))))))))). ( -37.70, z-score = -3.28, R) >droYak2.chrX 19967724 120 + 21770863 -------------GCUUAAUUGAUGCGACAGCUGCAAUGUGCAUAUUUUGUCUAUUAAUUU--GUGUUUGUGCAUAAUUGGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-UCCAAAUGCAAAU-GUGCUGUCAUAA -------------((.........))((((((.(((...(((((...(((.((.((((.(.--.((.(((((((((((((....)))))))))))))--.))..))))).))-..))))))))..)-)))))))).... ( -35.60, z-score = -2.44, R) >droEre2.scaffold_4690 3965769 120 + 18748788 -------------GCUUAAUUGAUGCGACAGCUGCAAUGUGCAUAUUUUGUCUAUUAAUUU--GUGUUUGUGCAUAAUUGGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-UCCAAAUGCAAAU-GUGCUGUCAUAA -------------((.........))((((((.(((...(((((...(((.((.((((.(.--.((.(((((((((((((....)))))))))))))--.))..))))).))-..))))))))..)-)))))))).... ( -35.60, z-score = -2.44, R) >droAna3.scaffold_13335 2028123 119 + 3335858 -------------GCUUAAUUGAUGCGACAGCUGCAAUGUGCA-AUUUUGUCUAUUAAUUU--GUGUUUAUGCAUAAUUGGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-CCAAAAUGCAAAU-GGCCUGUCAUAA -------------((.........))(((((..((....((((-.(((((.((.((((.(.--.((.(((((((((((((....)))))))))))))--.))..))))).))-.)))))))))...-.))))))).... ( -35.90, z-score = -2.78, R) >droSim1.chrX 9303596 120 - 17042790 -------------GCUUAAUUGAUGCGACAGCUGCAAUGUGCAUAUUUUGUCUAUUAAUUU--GUGUUUGUGCAUAAUUGGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-UCCAAAUGCAAAU-GGGCUGUCAUAA -------------((.........))(((((((.((...(((((...(((.((.((((.(.--.((.(((((((((((((....)))))))))))))--.))..))))).))-..))))))))..)-)))))))).... ( -35.30, z-score = -2.29, R) >dp4.chrXL_group1a 2779161 122 - 9151740 -------------GAAUAAUUGAUCCGACAGCUGCAAUAUGCAUAUUUUGUCUAUUAAUUUGUGUGUUUAUGCAUAAUUAGGAUUAAUUAUGCAUAA--UCGUGAUUAACAG-UCAACAUGCAAAU-GGCCUGUCAUAA -------------.............(((((..((((((.(((.....))).)))).(((((((((((((((((((((((....)))))))))))))--...((((.....)-)))))))))))))-.))))))).... ( -32.80, z-score = -2.44, R) >droPer1.super_12 1883795 122 + 2414086 -------------GAAUAAUUGAUCCGACAGCUGCAAUAUGCAUAUUUUGUCUAUUAAUUUGUGUGUUUAUGCAUAAUUAGGAUUAAUUAUGCAUAA--UUGUGAUUAACAG-UCAACAUGCAAAU-GGCCUGUCAUAA -------------.............(((((..((((((.(((.....))).)))).(((((((((((((((((((((((....))))))))))))(--((((.....))))-).)))))))))))-.))))))).... ( -33.60, z-score = -2.59, R) >droWil1.scaffold_180777 3811925 137 - 4753960 GGCUAAUUGAUGGGACAGGCAGCUGCAAUGGCAGCAAUAUGCAGAUUUUGUCUAUUAAUUU--GUGUUUAUGCAUAAUUAGGAUUAAUUAUGCAUAAUCUCAUGAUUAACAGACCAAAAUGCAAAUGGGCCUGUCAUAA .............(((((((.(((((....)))))....((((..(((.((((.(((((((--(.(.(((((((((((((....))))))))))))).).)).)))))).)))).))).)))).....))))))).... ( -49.80, z-score = -4.96, R) >droMoj3.scaffold_6359 132224 109 - 4525533 ----------------------CUCUAAUUGAUGCAAUAUGCAUAUUUUGUCUAUUAAUUU--GUGUUUAUGCAUAAUGCAAAUUAAUUAUGCAUAA--UCGUGAUUAACAG--CCAAAUGCAAAUUUGGCAUCGAA-- ----------------------......(((((((.((.(((((...(((.((.((((.(.--.((.(((((((((((........)))))))))))--.))..))))).))--.)))))))).))...))))))).-- ( -31.30, z-score = -3.01, R) >droGri2.scaffold_15081 124559 108 + 4274704 ----------------------GACUAAUUGACGCAAUAUGCAUACUUUGUCUAUUAAUUU--GUGUUUAUGCAUAAAUCGAAUUAAUUAUGCAUAA--UCGUGAUUAACAG--CCAAAUGUAAAUUGGGCUUAAA--- ----------------------......(((((.((((.(((((...(((.((.((((.(.--.((.((((((((((..........))))))))))--.))..))))).))--.)))))))).)))).).)))).--- ( -23.10, z-score = -1.52, R) >consensus _____________GCUUAAUUGAUGCGACAGCUGCAAUGUGCAUAUUUUGUCUAUUAAUUU__GUGUUUAUGCAUAAUUGGGAUUAAUUAUGCAUAA__UCGUGAUUAACAG_UCCAAAUGCAAAU_GGGCUGUCAUAA ...........................((((........(((((.......((.((((((.......(((((((((((((....)))))))))))))......)))))).))......))))).......))))..... (-19.56 = -19.80 + 0.24)



Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:36:19 2011