
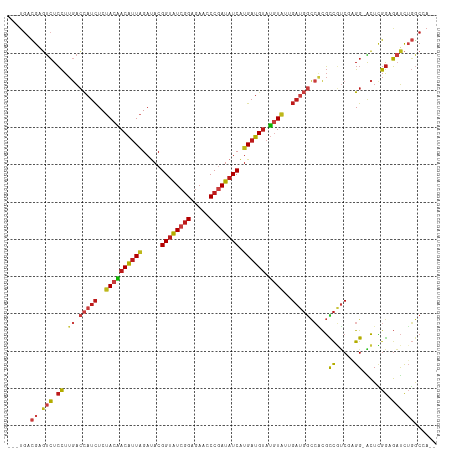
| Sequence ID | dm3.chrX |
|---|---|
| Location | 11,833,765 – 11,833,897 |
| Length | 132 |
| Max. P | 0.925125 |

| Location | 11,833,765 – 11,833,874 |
|---|---|
| Length | 109 |
| Sequences | 14 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 84.09 |
| Shannon entropy | 0.36861 |
| G+C content | 0.49358 |
| Mean single sequence MFE | -37.67 |
| Consensus MFE | -22.65 |
| Energy contribution | -23.41 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.20 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.925125 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 11833765 109 + 22422827 ---UGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGG-ACUCGGAGAUCUGGCCA-- ---...((((..((((((((.(((..((((((((((.....((((((((.....)))))))).)))))).))))..)))(((...))))))))))-)))))............-- ( -38.90, z-score = -2.60, R) >droSim1.chrX 9112017 109 + 17042790 ---UGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGG-ACUCGGAGAUCUGGCCA-- ---...((((..((((((((.(((..((((((((((.....((((((((.....)))))))).)))))).))))..)))(((...))))))))))-)))))............-- ( -38.90, z-score = -2.60, R) >droSec1.super_53 50920 109 + 190754 ---UGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGG-ACUCGGAGAUCUGGCCA-- ---...((((..((((((((.(((..((((((((((.....((((((((.....)))))))).)))))).))))..)))(((...))))))))))-)))))............-- ( -38.90, z-score = -2.60, R) >droYak2.chrX 6904386 109 - 21770863 ---UGGCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGG-ACUCGGAGAUCUGGCCA-- ---.(((.(((((((..(.((.....((((((((((.....((((((((.....)))))))).)))))).))))((((((((...))))))))))-.)..))))).)).))).-- ( -45.20, z-score = -4.05, R) >droEre2.scaffold_4690 15404952 109 - 18748788 ---UGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGG-ACUCGGAGAUCUGGCCA-- ---...((((..((((((((.(((..((((((((((.....((((((((.....)))))))).)))))).))))..)))(((...))))))))))-)))))............-- ( -38.90, z-score = -2.60, R) >droAna3.scaffold_12948 310737 109 + 692136 ---UGACGAGUCUCCUUGACCAUCUCUACGACAUUAGAUACGGUAUCGGAGAACCCUAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCUUCGAGG-GCUUGGCGAUCUGGCGA-- ---.......(((((...(((...((((......))))...)))...)))))....(((((....)))))(((...(((.((((.((((....))-)).)))).)))..))).-- ( -32.40, z-score = -0.77, R) >dp4.chrXL_group1e 2499098 109 - 12523060 ---UGACGUGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGAGG-ACUUGGAGAUCUGGCAA-- ---((.((.((((((..(.(((((..((((((((((.....((((((((.....)))))))).)))))).))))..)))))((.((....)).))-.)..)))))).)).)).-- ( -38.20, z-score = -2.49, R) >droPer1.super_22 208864 109 + 1688296 ---UGACGUGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGAGG-ACUUGGAGAUCUGGCAA-- ---((.((.((((((..(.(((((..((((((((((.....((((((((.....)))))))).)))))).))))..)))))((.((....)).))-.)..)))))).)).)).-- ( -38.20, z-score = -2.49, R) >droWil1.scaffold_181096 10240766 109 + 12416693 ---UGGCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGCGG-ACUCGGAGAUCGGGCUA-- ---((((..((((((....(((((..((((((((((.....((((((((.....)))))))).)))))).))))..)))))((.((....)).))-....))))).)..))))-- ( -39.80, z-score = -2.08, R) >droMoj3.scaffold_6473 12662191 109 + 16943266 ---UGCCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAAUCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUUGG-ACUUGGCGAUCUGGCCA-- ---.(((((((((...((((((((..((((((((((.....((((((((.....)))))))).)))))).))))..))))).....)))....))-)))))))..........-- ( -40.50, z-score = -3.01, R) >droVir3.scaffold_12970 8654635 109 + 11907090 ---UGUCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUAGG-ACUUGGCGAUCUGGCCA-- ---.((((((....)))))).(((..((((((((((.....((((((((.....)))))))).)))))).))))..)))(((((.((((((....-....)))))).))))).-- ( -42.80, z-score = -3.63, R) >droGri2.scaffold_15203 11402485 109 + 11997470 ---UGGCGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGCCGUCUUGG-ACUUGGCGAUCUGGCCA-- ---.(((.((((.((..(.(((....((((((((((.....((((((((.....)))))))).)))))).))))..((((((...)))))).)))-.)..)).)).)).))).-- ( -40.00, z-score = -2.76, R) >anoGam1.chr2R 23167134 103 + 62725911 CGUCUGUGUUCCUCCUUUUGUAUUUCCACCACGUUCGACACGGUGUCGGAGAAACCGAUCUCGUGCUGUUGAUG---------UCGCUUCCGCGG-GCUCGGCGUGCGCACCA-- .....(((((........((......))........)))))((((((((.....)))))...((((((((((..---------((((....))))-..)))))).))))))).-- ( -27.49, z-score = 1.30, R) >triCas2.ChLG10 852862 112 + 8806720 ---UGAUGUUCGUGUUUAACAAUCUCGACCACAUUGGACACGGUAUCAGAAAAGCCAAUAUCGUGAUGAAGAUGAGGAGGAUAUUUUCUACGCGGUGAUGGACUUCUUUGCAACG ---.......(((((((((..............)))))))))((..((((.((((((.(((((((..(((((((.......)))))))..))))))).))).))).))))..)). ( -27.24, z-score = -0.37, R) >consensus ___UGACGAGUCUCCUUGACCAUCUCUACAACAUUAGAUACGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGG_ACUCGGAGAUCUGGCCA__ ......((.(((.((.((.(((((..((((((((((.....((((((((.....)))))))).)))))).))))..))))).))..((.....)).....)).))).))...... (-22.65 = -23.41 + 0.76)


| Location | 11,833,765 – 11,833,874 |
|---|---|
| Length | 109 |
| Sequences | 14 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 84.09 |
| Shannon entropy | 0.36861 |
| G+C content | 0.49358 |
| Mean single sequence MFE | -35.79 |
| Consensus MFE | -23.25 |
| Energy contribution | -24.10 |
| Covariance contribution | 0.85 |
| Combinations/Pair | 1.34 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.65 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.920873 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
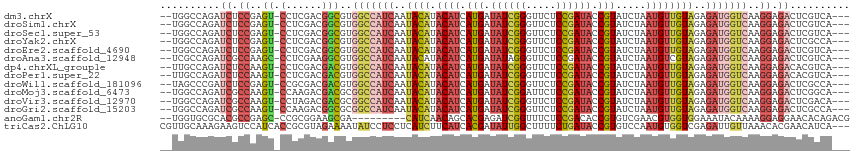
>dm3.chrX 11833765 109 - 22422827 --UGGCCAGAUCUCCGAGU-CCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCA--- --.(((.((.(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))))).))).--- ( -39.30, z-score = -2.38, R) >droSim1.chrX 9112017 109 - 17042790 --UGGCCAGAUCUCCGAGU-CCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCA--- --.(((.((.(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))))).))).--- ( -39.30, z-score = -2.38, R) >droSec1.super_53 50920 109 - 190754 --UGGCCAGAUCUCCGAGU-CCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCA--- --.(((.((.(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))))).))).--- ( -39.30, z-score = -2.38, R) >droYak2.chrX 6904386 109 + 21770863 --UGGCCAGAUCUCCGAGU-CCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCA--- --.(((.((.(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))))).))).--- ( -42.00, z-score = -3.16, R) >droEre2.scaffold_4690 15404952 109 + 18748788 --UGGCCAGAUCUCCGAGU-CCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCA--- --.(((.((.(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))))).))).--- ( -39.30, z-score = -2.38, R) >droAna3.scaffold_12948 310737 109 - 692136 --UCGCCAGAUCGCCAAGC-CCUCGAAGGCGUGGCCAUCAAUACAUACAUCAUGAUAUAGGGUUCUCCGAUACCGUAUCUAAUGUCGUAGAGAUGGUCAAGGAGACUCGUCA--- --......(((.((((.((-(......))).)))).))).............((((...((((.((((...(((((.((((......)))).)))))...))))))))))))--- ( -34.20, z-score = -1.87, R) >dp4.chrXL_group1e 2499098 109 + 12523060 --UUGCCAGAUCUCCAAGU-CCUCGACGACGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACACGUCA--- --........(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))).......--- ( -35.10, z-score = -2.28, R) >droPer1.super_22 208864 109 - 1688296 --UUGCCAGAUCUCCAAGU-CCUCGACGACGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACACGUCA--- --........(((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))).......--- ( -35.10, z-score = -2.28, R) >droWil1.scaffold_181096 10240766 109 - 12416693 --UAGCCCGAUCUCCGAGU-CCGCGACGACGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCA--- --.....((((((((..((-(......))).((((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..)))))))).))))).)))...--- ( -37.60, z-score = -2.37, R) >droMoj3.scaffold_6473 12662191 109 - 16943266 --UGGCCAGAUCGCCAAGU-CCAAGACGACGCGGCCAUCAAUACAUACAUCAUGAUAUCGGAUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGGCA--- --..(((((.((.((..((-(......)))..(((((((..((((.((((.(((.((((((.....)))))).))).....))))))))..)))))))..)).)))).))).--- ( -37.80, z-score = -3.02, R) >droVir3.scaffold_12970 8654635 109 - 11907090 --UGGCCAGAUCGCCAAGU-CCUAGACGACGCGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGACA--- --((((......)))).((-(...........(((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..))))))).((....)).))).--- ( -34.10, z-score = -1.71, R) >droGri2.scaffold_15203 11402485 109 - 11997470 --UGGCCAGAUCGCCAAGU-CCAAGACGGCGCGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGCCA--- --((((......)))).((-(...)))((((.(((((((..((((.((((...((((.(((..((...))..)))))))..))))))))..))))))).((....)))))).--- ( -38.90, z-score = -2.93, R) >anoGam1.chr2R 23167134 103 - 62725911 --UGGUGCGCACGCCGAGC-CCGCGGAAGCGA---------CAUCAACAGCACGAGAUCGGUUUCUCCGACACCGUGUCGAACGUGGUGGAAAUACAAAAGGAGGAACACAGACG --..((((((.......))-.(((....))).---------........))))....((.((((((((..(((((((.....)))))))...........))))))))...)).. ( -31.32, z-score = -0.19, R) >triCas2.ChLG10 852862 112 - 8806720 CGUUGCAAAGAAGUCCAUCACCGCGUAGAAAAUAUCCUCCUCAUCUUCAUCACGAUAUUGGCUUUUCUGAUACCGUGUCCAAUGUGGUCGAGAUUGUUAAACACGAACAUCA--- .(((..((...((((..(((((((((.(((...............)))..((((.(((..(.....)..))).))))....))))))).)))))).)).)))..........--- ( -17.76, z-score = 1.22, R) >consensus __UGGCCAGAUCUCCGAGU_CCUCGACGGCGUGGCCAUCAAUACAUACAUCAUGAUAUCGGGUUCUCCGAUACCGUAUCUAAUGUUGUAGAGAUGGUCAAGGAGACUCGUCA___ ...........................((((.(((((((..((((.((((.(((.((((((.....)))))).))).....))))))))..)))))))..(....).)))).... (-23.25 = -24.10 + 0.85)

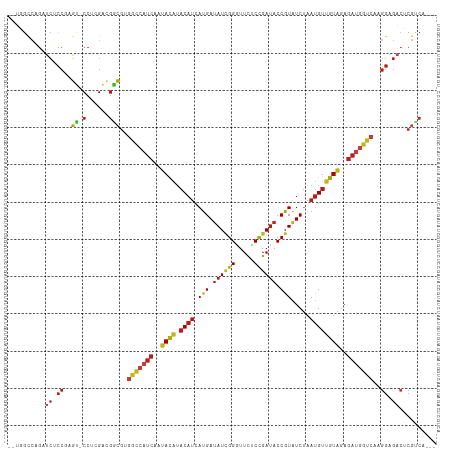
| Location | 11,833,802 – 11,833,897 |
|---|---|
| Length | 95 |
| Sequences | 14 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 76.49 |
| Shannon entropy | 0.52396 |
| G+C content | 0.53471 |
| Mean single sequence MFE | -33.29 |
| Consensus MFE | -18.11 |
| Energy contribution | -18.29 |
| Covariance contribution | 0.18 |
| Combinations/Pair | 1.52 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.54 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.769438 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
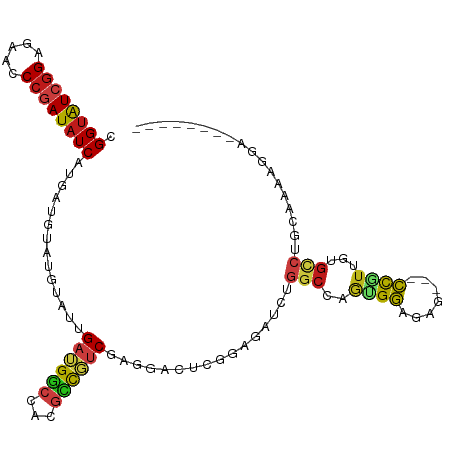
>dm3.chrX 11833802 95 + 22422827 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAG---CCAUUGUGCCUGCAC------------- .((((((((.....))))))))..(((.(..((.((((((((...))))))))..))....).))).(((((((((....---)))))).))).....------------- ( -36.30, z-score = -2.18, R) >droSim1.chrX 9112054 95 + 17042790 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAG---CCGUUGUGCCUGCAG------------- .((((((((.....))))))))..(((.(..((.((((((((...))))))))..))....).))).(((((((((....---)))))).))).....------------- ( -32.90, z-score = -1.04, R) >droSec1.super_53 50957 95 + 190754 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAG---CCGUUGUGCCUGCCG------------- .((((((((.....)))))))).........((.((((((((...))))))))..)).(((......(((((((((....---)))))).)))..)))------------- ( -33.00, z-score = -0.92, R) >droYak2.chrX 6904423 102 - 21770863 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAG---CCGUUGUGCCUGCAGAAAGCAG------ .((((((((.....))))))))..(((.(..((.((((((((...))))))))..))....).))).(((((((((....---)))))).)))(((.....))).------ ( -34.70, z-score = -1.30, R) >droEre2.scaffold_4690 15404989 102 - 18748788 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAG---CCGUUGUGCCUGCAGAAGGGAU------ .((((((((.....))))))))..(((.(..((.((((((((...))))))))..))....).))).(((((((((....---)))))).)))............------ ( -32.90, z-score = -0.37, R) >droAna3.scaffold_12948 310774 102 + 692136 CGGUAUCGGAGAACCCUAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCUUCGAGGGCUUGGCGAUCUGGCGAGCGGAGAG---CCGUUAUGCCUGAAAAUAAACG------ .(((((.((.....)).))))).....((.(((...(((.((((.((((....)))).)))).))).(((((((((....---))))).)))).....))).)).------ ( -34.40, z-score = -1.80, R) >dp4.chrXL_group1e 2499135 100 - 12523060 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGAGGACUUGGAGAUCUGGCAAGUGGCGAG---CCAUUGUGCCUGCAAAAGGA-------- .((((((((.....))))))))........((((..(((..(((.(((......))).)))..))).(((((((((....---))))).))))))))......-------- ( -34.10, z-score = -1.56, R) >droPer1.super_22 208901 100 + 1688296 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGAGGACUUGGAGAUCUGGCAAGUGGCGAG---CCAUUGUGCCUGCAAAAGGA-------- .((((((((.....))))))))........((((..(((..(((.(((......))).)))..))).(((((((((....---))))).))))))))......-------- ( -34.10, z-score = -1.56, R) >droWil1.scaffold_181096 10240803 99 + 12416693 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGUCGUCGCGGACUCGGAGAUCGGGCUAAUGGCGAG---CCGUUAUGCCUAGAAAAAG--------- .((((((((.....)))))))).(((.((.(((..(((((....)))))..))).)))))....((((((((((((....---)))))).)))).)).....--------- ( -32.90, z-score = -1.23, R) >droMoj3.scaffold_6473 12662228 91 + 16943266 CGGUAUCGGAGAAUCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUUGGACUUGGCGAUCUGGCCAAAGGAGAA---CCGUUGUGUCU----------------- .((((((((.....))))))))..((..((........((((((.((((((........)))))).))))))..((....---))..))..)).----------------- ( -34.00, z-score = -2.55, R) >droVir3.scaffold_12970 8654672 100 + 11907090 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGUCGUCUAGGACUUGGCGAUCUGGCCAAAGGAGAA---CCGUUGUGUCUGUGAUGGAA-------- .((((((((.....))))))))................((((((.((((((........)))))).))))))........---((((..(....)..))))..-------- ( -35.60, z-score = -2.29, R) >droGri2.scaffold_15203 11402522 100 + 11997470 CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCGCGCCGUCUUGGACUUGGCGAUCUGGCCAAUGGAGAA---CCGUUGUGUCUGUAACAGAA-------- .((((((((.....))))))))........(((...(((.((((.(((.....)).).)))).))).(((((((((....---)))))).)))....)))...-------- ( -32.50, z-score = -1.33, R) >anoGam1.chr2R 23167174 101 + 62725911 CGGUGUCGGAGAAACCGAUCUCGUGCUGU------UGAUGU---CGCUUCCGCGGGCUCGGCGUGCGCACCAGCGGAGACUC-CCGUCUUGCCGGUCGAUGUAGAAAGGGA ..(..(((((((......))))(((((((------(((..(---(((....))))..)))))).))))(((.(((.((((..-..))))))).))))))..)......... ( -33.70, z-score = 1.70, R) >triCas2.ChLG10 852899 105 + 8806720 CGGUAUCAGAAAAGCCAAUAUCGUGAUGAAGAUGAGGAGGAUAUUUUCUACGCGGUGAUGGACUUCUUUGCAACGAAGGCGAAUUACCACGUCUACACAAAAAAU------ ((....((((.((((((.(((((((..(((((((.......)))))))..))))))).))).))).))))...)).(((((........)))))...........------ ( -25.00, z-score = -1.45, R) >consensus CGGUAUCGGAGAACCCGAUAUCAUGAUGUAUGUAUUGAUGGCCACGCCGUCGAGGACUCGGAGAUCUGGCCAGUGGAGAG___CCGUUGUGCCUGCAAAAGGA________ .((((((((.....))))))))..............((((((...))))))................(((..((((.......))))...))).................. (-18.11 = -18.29 + 0.18)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:35:44 2011