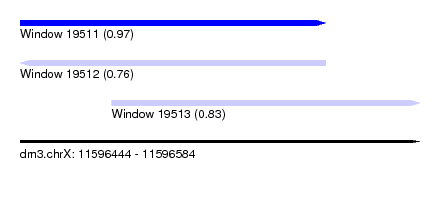
| Sequence ID | dm3.chrX |
|---|---|
| Location | 11,596,444 – 11,596,584 |
| Length | 140 |
| Max. P | 0.973880 |

| Location | 11,596,444 – 11,596,551 |
|---|---|
| Length | 107 |
| Sequences | 11 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 92.06 |
| Shannon entropy | 0.16075 |
| G+C content | 0.39386 |
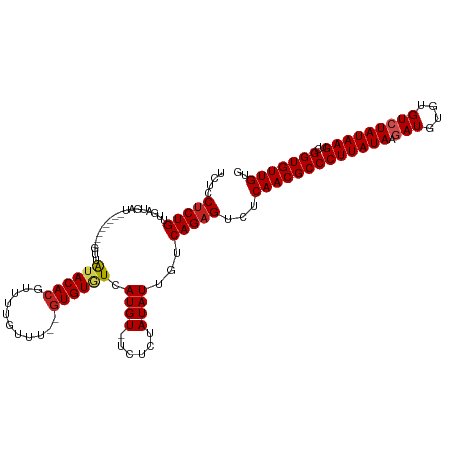
| Mean single sequence MFE | -27.26 |
| Consensus MFE | -18.15 |
| Energy contribution | -18.48 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.30 |
| Structure conservation index | 0.67 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.90 |
| SVM RNA-class probability | 0.973880 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
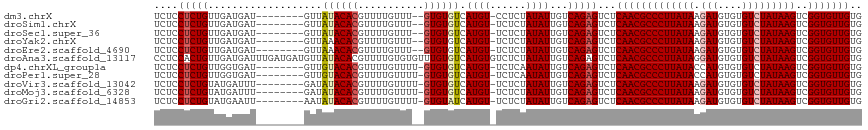
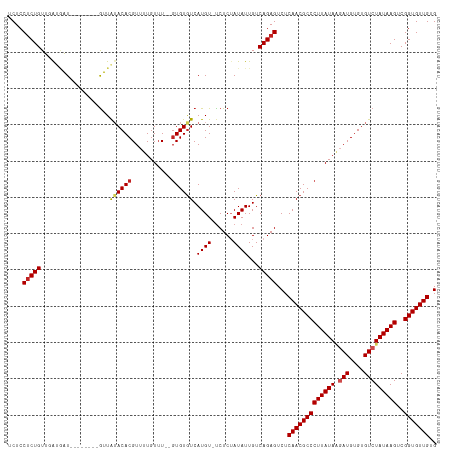
>dm3.chrX 11596444 107 + 22422827 UCUCCUCUGUUGAUGAU--------GUUAUACACGUUUUGUUU--GUGUGUCAUGU-CCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((..((.(((--------((.(((((((.......)--)))))).))))-).))........)))))...(((((((((((((.(((....)))))))))..))))))).. ( -29.70, z-score = -4.42, R) >droSim1.chrX 8925397 107 + 17042790 UCUCCUCUGUUGAUGAU--------GUUAUACACGUUUUGUUU--GUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((..((.(((--------((.(((((((.......)--)))))).))))-).))........)))))...(((((((((((((.(((....)))))))))..))))))).. ( -28.00, z-score = -3.72, R) >droSec1.super_36 285111 107 + 449511 UCUCCUCUGUUGAUGAU--------GUUAUACACGUUUUGUUU--GUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((..((.(((--------((.(((((((.......)--)))))).))))-).))........)))))...(((((((((((((.(((....)))))))))..))))))).. ( -28.00, z-score = -3.72, R) >droYak2.chrX 6681826 107 - 21770863 UCUCCUCUGUUGAUGAU--------GUUAAACACGUUUUGUUU--GUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((..((.(((--------((...(((((.......)--))))...))))-).))........)))))...(((((((((((((.(((....)))))))))..))))))).. ( -25.00, z-score = -2.85, R) >droEre2.scaffold_4690 15158723 107 - 18748788 UCUCCUCUGUUGAUGAU--------GUUAAACACGUUUUGUUU--GUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((..((.(((--------((...(((((.......)--))))...))))-).))........)))))...(((((((((((((.(((....)))))))))..))))))).. ( -25.00, z-score = -2.85, R) >droAna3.scaffold_13117 1091439 118 - 5790199 CCUCCACUGUUGAUGAUUUGAUGAUGUUAUACACGUUUUGUGUGUUUGUGUCAUGUGUCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAGGAUGUGUGUCUAUAAGUCGGUGUUGUG .(((((.(((.((.(((...((((..(.((((((.....))))))..)..))))..))))).))).))...)))...(((((((((((((((.......))))))))..))))))).. ( -29.80, z-score = -1.94, R) >dp4.chrXL_group1a 6832161 108 - 9151740 UCUCCUCUGUUGGUGAU--------GUUGUACACGUUUUGUUUU-GUGUGUCAUGU-UCUCAAUAUUGUCAGAGUCUCAACGCCCUUAUACCAUGUGUGUCUAUAAGUCGGUGUUGUG ....((((((((..(((--------((..((((((........)-)))))..))))-)..)))......)))))...(((((((((((((.((....))..))))))..))))))).. ( -25.90, z-score = -2.57, R) >droPer1.super_28 354937 108 - 1111753 UCUCCUCUGUUGGUGAU--------GUUGUACACGUUUUGUUUU-GUGUGUCAUGU-UCUCAAUAUUGUCAGAGUCUCAACGCCCUUAUACCAUGUGUGUCUAUAAGUCGGUGUUGUG ....((((((((..(((--------((..((((((........)-)))))..))))-)..)))......)))))...(((((((((((((.((....))..))))))..))))))).. ( -25.90, z-score = -2.57, R) >droVir3.scaffold_13042 2806062 108 + 5191987 UCUCCUCUGUAUGAUUU--------GAUAUACACGUUUUGUUUU-GUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((.(..((.(--------((((((((.(......).)-))))))))...-.......))..))))))...(((((((((((((.(((....)))))))))..))))))).. ( -28.00, z-score = -3.87, R) >droMoj3.scaffold_6328 3645061 108 - 4453435 UCUCCUCUGUAUGAUUU--------GAUAUACACGUUUUGUUUU-GUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((.(..((.(--------((((((((.(......).)-))))))))...-.......))..))))))...(((((((((((((.(((....)))))))))..))))))).. ( -28.00, z-score = -3.87, R) >droGri2.scaffold_14853 8815489 108 - 10151454 UCUCCUCUGUAUGAAUU--------AAUAUACACGUUUUGUUUU-GUGUAUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((...((((.--------...(((((((........)-))))))...))-))..........)))))...(((((((((((((.(((....)))))))))..))))))).. ( -26.52, z-score = -3.93, R) >consensus UCUCCUCUGUUGAUGAU________GUUAUACACGUUUUGUUU__GUGUGUCAUGU_UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUG ....(((((...................((((((...........)))))).((((......))))...)))))...(((((((((((((.(((....)))))))))..))))))).. (-18.15 = -18.48 + 0.33)
| Location | 11,596,444 – 11,596,551 |
|---|---|
| Length | 107 |
| Sequences | 11 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 92.06 |
| Shannon entropy | 0.16075 |
| G+C content | 0.39386 |
| Mean single sequence MFE | -18.93 |
| Consensus MFE | -15.39 |
| Energy contribution | -15.51 |
| Covariance contribution | 0.13 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.81 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.762276 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 11596444 107 - 22422827 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGG-ACAUGACACAC--AAACAAAACGUGUAUAAC--------AUCAUCAACAGAGGAGA ..((((.((..((((((((......)).)))))))).))))...(((((............-(((((......--........))))).....--------........))))).... ( -18.05, z-score = -1.59, R) >droSim1.chrX 8925397 107 - 17042790 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGACACAC--AAACAAAACGUGUAUAAC--------AUCAUCAACAGAGGAGA .......((..((((((((......)).))))))...((((((((((((......))))).-.......((((--.........)))).....--------.)).)))))...))... ( -17.80, z-score = -1.81, R) >droSec1.super_36 285111 107 - 449511 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGACACAC--AAACAAAACGUGUAUAAC--------AUCAUCAACAGAGGAGA .......((..((((((((......)).))))))...((((((((((((......))))).-.......((((--.........)))).....--------.)).)))))...))... ( -17.80, z-score = -1.81, R) >droYak2.chrX 6681826 107 + 21770863 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGACACAC--AAACAAAACGUGUUUAAC--------AUCAUCAACAGAGGAGA ..((((.((..((((((((......)).)))))))).))))...(((((..........((-(((((......--........)))))))...--------........))))).... ( -19.34, z-score = -2.31, R) >droEre2.scaffold_4690 15158723 107 + 18748788 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGACACAC--AAACAAAACGUGUUUAAC--------AUCAUCAACAGAGGAGA ..((((.((..((((((((......)).)))))))).))))...(((((..........((-(((((......--........)))))))...--------........))))).... ( -19.34, z-score = -2.31, R) >droAna3.scaffold_13117 1091439 118 + 5790199 CACAACACCGACUUAUAGACACACAUCCUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGACACAUGACACAAACACACAAAACGUGUAUAACAUCAUCAAAUCAUCAACAGUGGAGG (((........(((((((.........)))))))...((((((((((((......)))))....((((.......((((.....))))......))))....)).))))).))).... ( -21.42, z-score = -1.77, R) >dp4.chrXL_group1a 6832161 108 + 9151740 CACAACACCGACUUAUAGACACACAUGGUAUAAGGGCGUUGAGACUCUGACAAUAUUGAGA-ACAUGACACAC-AAAACAAAACGUGUACAAC--------AUCACCAACAGAGGAGA ..((((.((..((((((..((....)).)))))))).))))...(((((.......(((..-.....((((..-..........)))).....--------.)))....))))).... ( -18.42, z-score = -1.13, R) >droPer1.super_28 354937 108 + 1111753 CACAACACCGACUUAUAGACACACAUGGUAUAAGGGCGUUGAGACUCUGACAAUAUUGAGA-ACAUGACACAC-AAAACAAAACGUGUACAAC--------AUCACCAACAGAGGAGA ..((((.((..((((((..((....)).)))))))).))))...(((((.......(((..-.....((((..-..........)))).....--------.)))....))))).... ( -18.42, z-score = -1.13, R) >droVir3.scaffold_13042 2806062 108 - 5191987 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGACACAC-AAAACAAAACGUGUAUAUC--------AAAUCAUACAGAGGAGA ..((((.((..((((((((......)).)))))))).))))...(((((........((..-(((((......-.........)))))...))--------........))))).... ( -18.35, z-score = -2.07, R) >droMoj3.scaffold_6328 3645061 108 + 4453435 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGACACAC-AAAACAAAACGUGUAUAUC--------AAAUCAUACAGAGGAGA ..((((.((..((((((((......)).)))))))).))))...(((((........((..-(((((......-.........)))))...))--------........))))).... ( -18.35, z-score = -2.07, R) >droGri2.scaffold_14853 8815489 108 + 10151454 CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA-ACAUGAUACAC-AAAACAAAACGUGUAUAUU--------AAUUCAUACAGAGGAGA ..((((.((..((((((((......)).)))))))).))))...(((((.(......).((-(....((((((-..........))))))...--------..)))...))))).... ( -20.90, z-score = -3.22, R) >consensus CACAACACCGACUUAUAGACACACAUCUUAUAAGGGCGUUGAGACUCUGACAAUAUAGAGA_ACAUGACACAC__AAACAAAACGUGUAUAAC________AUCAUCAACAGAGGAGA ..((((.((..(((((((.........))))))))).))))...(((((.(......).........((((.............)))).....................))))).... (-15.39 = -15.51 + 0.13)
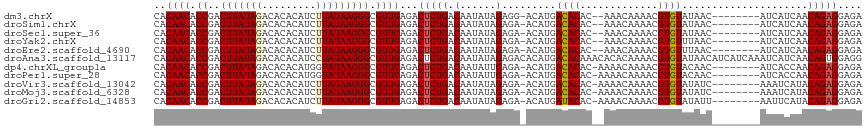
| Location | 11,596,476 – 11,596,584 |
|---|---|
| Length | 108 |
| Sequences | 11 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 93.52 |
| Shannon entropy | 0.13580 |
| G+C content | 0.41317 |
| Mean single sequence MFE | -25.57 |
| Consensus MFE | -22.97 |
| Energy contribution | -23.15 |
| Covariance contribution | 0.18 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.90 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.827722 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 11596476 108 + 22422827 --UUUGUGUGUCAUGU-CCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG---UCAAACUG--- --...((.((.(.((.-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...)))))).....)).)---.)).))..--- ( -24.40, z-score = -1.51, R) >droSim1.chrX 8925429 108 + 17042790 --UUUGUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG---UCAAACUG--- --...((.((.(.((.-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...)))))).....)).)---.)).))..--- ( -25.10, z-score = -1.70, R) >droSec1.super_36 285143 108 + 449511 --UUUGUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG---UCAAACUG--- --...((.((.(.((.-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...)))))).....)).)---.)).))..--- ( -25.10, z-score = -1.70, R) >droYak2.chrX 6681858 108 - 21770863 --UUUGUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG---UCAAAGUG--- --((((..((......-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...)))))).....))..---.))))...--- ( -24.70, z-score = -1.82, R) >droEre2.scaffold_4690 15158755 108 - 18748788 --UUUGUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG---UCAAACUG--- --...((.((.(.((.-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...)))))).....)).)---.)).))..--- ( -25.10, z-score = -1.70, R) >droAna3.scaffold_13117 1091479 113 - 5790199 UGUGUUUGUGUCAUGUGUCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAGGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG---UCAAACUGAU- ...(((((......((..(((.(((......(((((((((((((((((((((.......))))))))..))))))...)))))))...))))))...))....---.)))))....- ( -29.30, z-score = -1.89, R) >dp4.chrXL_group1a 6832193 109 - 9151740 -UUUUGUGUGUCAUGU-UCUCAAUAUUGUCAGAGUCUCAACGCCCUUAUACCAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCAUUGUGAGUAGACCAAA---GAAAACUG--- -((((.((.(((....-.((((....((...(((((((((((((((((((.((....))..))))))..))))))...)))))))))...))))..))))).)---))).....--- ( -26.30, z-score = -1.31, R) >droPer1.super_28 354969 109 - 1111753 -UUUUGUGUGUCAUGU-UCUCAAUAUUGUCAGAGUCUCAACGCCCUUAUACCAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCAUUGUGAGUAGACCAAA---GAAAACUG--- -((((.((.(((....-.((((....((...(((((((((((((((((((.((....))..))))))..))))))...)))))))))...))))..))))).)---))).....--- ( -26.30, z-score = -1.31, R) >droVir3.scaffold_13042 2806094 115 + 5191987 -UUUUGUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAAAAGUCAAAUUGCCA -(((((.((.......-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...))))))...)))))))............. ( -25.40, z-score = -1.75, R) >droMoj3.scaffold_6328 3645093 111 - 4453435 -UUUUGUGUGUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAA--GUCAAAAUGC-- -(((((..........-.(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...)))))).........--..)))))...-- ( -25.55, z-score = -2.12, R) >droGri2.scaffold_14853 8815521 111 - 10151454 -UUUUGUGUAUCAUGU-UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAA--GUAAAACUGC-- -(((((.((.((((..-...(......)...(((((((((((((((((((.(((....)))))))))..))))))...)))))))....))))....))))))--).........-- ( -24.00, z-score = -1.43, R) >consensus __UUUGUGUGUCAUGU_UCUCUAUAUUGUCAGAGUCUCAACGCCCUUAUAAGAUGUGUGUCUAUAAGUCGGUGUUGUGGAGACUCCCUUGUGAGUAAACCAAG___UCAAACUG___ ..............((..(((.(((......(((((((((((((((((((.(((....)))))))))..))))))...)))))))...))))))...)).................. (-22.97 = -23.15 + 0.18)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:35:14 2011