
| Sequence ID | dm3.chrX |
|---|---|
| Location | 11,567,977 – 11,568,082 |
| Length | 105 |
| Max. P | 0.999867 |

| Location | 11,567,977 – 11,568,082 |
|---|---|
| Length | 105 |
| Sequences | 12 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 72.55 |
| Shannon entropy | 0.57607 |
| G+C content | 0.49141 |
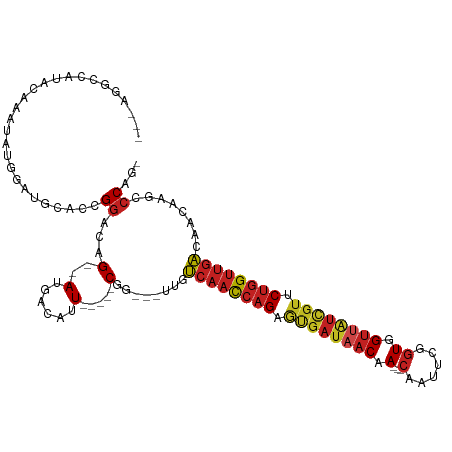
| Mean single sequence MFE | -41.79 |
| Consensus MFE | -14.92 |
| Energy contribution | -15.37 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.33 |
| Mean z-score | -4.71 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.64 |
| SVM RNA-class probability | 0.999867 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
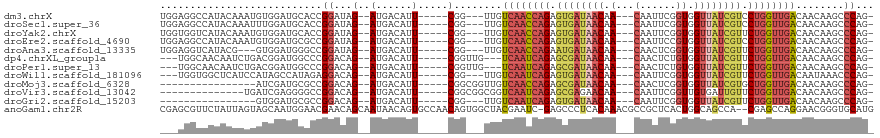
>dm3.chrX 11567977 105 + 22422827 UGGAGGCCAUACAAAUGUGGAUGCACCGGAUAG--AUGACAUU-----CGG---UUGUCAACCAGAGUGAUAACAA---CAAUUCGGUGGUUAUCGUCCUGGUUGACAACAAGCCCAG- (((...(((((....)))))..((..(((((..--.....)))-----))(---(((((((((((..(((((((.(---(......)).)))))))..))))))))))))..))))).- ( -41.10, z-score = -4.17, R) >droSec1.super_36 256789 105 + 449511 UGGAGGCCAUACAAAUUUGGAUGCACCGGAUAG--AUGACAUU-----CGG---UUGUCAACCAGAGUGAUAACAA---CAAUUCGGUGGUUAUCGUCCUGGUUGACAACAAGCCCAG- ..(.(((..........((((((((.(.....)--.)).))))-----))(---(((((((((((..(((((((.(---(......)).)))))))..))))))))))))..))))..- ( -40.60, z-score = -4.42, R) >droYak2.chrX 6652926 105 - 21770863 UGGUGGUCAUACAAAUGUGGAUGCACCGGAUAG--AUGACAUU-----CGG---UUGUCAACCAGAGUGAUAACAA---CAAUUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAG- .(((((((.(((....)))))).))))((...(--(......)-----).(---((((((((((((((((((((.(---(......)).)))))))))))))))))))))....))..- ( -42.90, z-score = -4.66, R) >droEre2.scaffold_4690 15130341 105 - 18748788 UGGAGGCCAUACAAAUGUGGAUGCGCCGGAUAG--AUGACAUU-----CGG---UUGUCAACCAGAGUGAUAACAA---CAAUUCCGUGGUUAUCGUCCUGGUUGACAACAAGCCCAG- (((...(((((....)))))..((..(((((..--.....)))-----))(---(((((((((((..(((((((.(---(......)).)))))))..))))))))))))..))))).- ( -41.20, z-score = -4.25, R) >droAna3.scaffold_13335 2820794 102 - 3335858 UGGAGGUCAUACG---GUGGAUGGGCCGGAUAG--AUGACAUU-----CGG---UUGUCAACCAGAAUGAUAACAA---CAACUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAG- (((..(((((.((---((......)))).....--)))))...-----..(---((((((((((((((((((((.(---(......)).)))))))))))))))))))))....))).- ( -47.90, z-score = -6.20, R) >dp4.chrXL_group1a 7303137 102 + 9151740 ---UGGCAACAAUCUGACGGAUGGCCCGGACAG--AUGACAUU-----CGGUUG---UCAAUCAGAGCGAUAACAA---CAACUCUGUGGUUAUCGUUCUGGUUGACAACAAGCCCAG- ---.(((....(((((.(((.....)))..)))--))......-----..((((---(((((((((((((((((.(---((....))).)))))))))))))))))))))..)))...- ( -49.10, z-score = -7.01, R) >droPer1.super_13 1921154 102 + 2293547 ---UGGCAACAAUCUGACGGAUGGCCCGGACAG--AUGACAUU-----CGGUUG---UCAAUCAGAGCGAUAACAA---CAACUCUGUGGUUAUCGUUCUGGUUGACAACAAGCCCAG- ---.(((....(((((.(((.....)))..)))--))......-----..((((---(((((((((((((((((.(---((....))).)))))))))))))))))))))..)))...- ( -49.10, z-score = -7.01, R) >droWil1.scaffold_181096 5838924 102 - 12416693 ---UGGUGGCUCAUCCAUAGCCAUAGAGGACAG--AUGACAUU-----CGG---UUGUCAAUCAGAGUGAUAACAA---CAAUUCGGUGGUUAUCGUUCUGGUUGACAAUAAACCCAG- ---..((((((.......))))))...((...(--(......)-----).(---((((((((((((((((((((.(---(......)).)))))))))))))))))))))....))..- ( -36.80, z-score = -4.00, R) >droMoj3.scaffold_6328 3613197 92 - 4453435 ----------------AUCGAUGCGCCGGACAG--AUGACAUU-----CGGCGGUUGUCAACCAGAGCGAUAACAA---CAACUCGGUGGUUAUCGUGCUGGUUGACAACAAGCCCAG- ----------------.......(((((((...--......))-----)))))((((((((((((.((((((((.(---(......)).)))))))).))))))))))))........- ( -42.50, z-score = -5.35, R) >droVir3.scaffold_13042 2774384 94 + 5191987 --------------UGAUCGAGGGGCCGGACAG--AUGACAUU-----CGGCGGCGGUCAAUCAGAGCGAGAACAA---CAAUUCGGUUGUGAUUGUUCUGGUUGACAACAAGCCCAG- --------------.......(((((((((...--......))-----)))).(..((((((((((((((..((((---(......)))))..))))))))))))))..)...)))..- ( -40.90, z-score = -4.81, R) >droGri2.scaffold_15203 5556355 89 + 11997470 ----------------GUGGAUGCGCCGGACAG--AUGACAUU-----CGG---UUGUCAAUCAGAGUGAUAACAA---CAAUUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAG- ----------------.((((((((.(.....)--.)).))))-----))(---((((((((((((((((((((.(---(......)).)))))))))))))))))))))........- ( -34.10, z-score = -3.68, R) >anoGam1.chr2R 7791653 116 - 62725911 CGAGCGUUCUAUUAGUAGCAAUGGAACGAACAGCAAUAACAGUGCCAACAGUGGCUACGAAUC-GAGCCCUCACAAACGCCGCUCACUGGCAGCCA--CGAGCCAGGAACGGGUGCAUG .(..(((((((((......)))))))))..).(((......(((.....((.((((.((...)-)))))))))).....(((.((.(((((.....--...))))))).))).)))... ( -35.30, z-score = -0.99, R) >consensus ___AGGCCAUACAAAUAUGGAUGCACCGGACAG__AUGACAUU_____CGG___UUGUCAACCAGAGUGAUAACAA___CAAUUCGGUGGUUAUCGUUCUGGUUGACAACAAGCCCAG_ .................................................((......((((((((.((((((((...............)))))))).))))))))........))... (-14.92 = -15.37 + 0.45)
| Location | 11,567,977 – 11,568,082 |
|---|---|
| Length | 105 |
| Sequences | 12 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 72.55 |
| Shannon entropy | 0.57607 |
| G+C content | 0.49141 |
| Mean single sequence MFE | -35.02 |
| Consensus MFE | -8.97 |
| Energy contribution | -9.43 |
| Covariance contribution | 0.46 |
| Combinations/Pair | 1.42 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.26 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.78 |
| SVM RNA-class probability | 0.967274 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
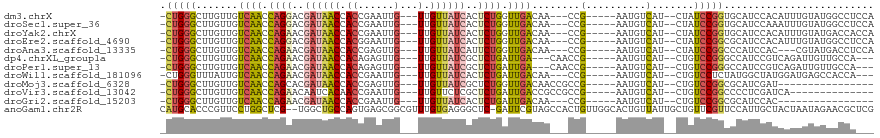
>dm3.chrX 11567977 105 - 22422827 -CUGGGCUUGUUGUCAACCAGGACGAUAACCACCGAAUUG---UUGUUAUCACUCUGGUUGACAA---CCG-----AAUGUCAU--CUAUCCGGUGCAUCCACAUUUGUAUGGCCUCCA -..(((((.(((((((((((((..((((((.((......)---).))))))..))))))))))))---)((-----((((((((--(.....)))).....)))))))...)))))... ( -39.10, z-score = -4.06, R) >droSec1.super_36 256789 105 - 449511 -CUGGGCUUGUUGUCAACCAGGACGAUAACCACCGAAUUG---UUGUUAUCACUCUGGUUGACAA---CCG-----AAUGUCAU--CUAUCCGGUGCAUCCAAAUUUGUAUGGCCUCCA -..(((((.(((((((((((((..((((((.((......)---).))))))..))))))))))))---).(-----.(((.(((--(.....))))))).)..........)))))... ( -37.40, z-score = -3.80, R) >droYak2.chrX 6652926 105 + 21770863 -CUGGGCUUGUUGUCAACCAGAACGAUAACCACCGAAUUG---UUGUUAUCACUCUGGUUGACAA---CCG-----AAUGUCAU--CUAUCCGGUGCAUCCACAUUUGUAUGACCACCA -.(((....(((((((((((((..((((((.((......)---).))))))..))))))))))))---)((-----((((((((--(.....)))).....))))))).....)))... ( -36.80, z-score = -4.09, R) >droEre2.scaffold_4690 15130341 105 + 18748788 -CUGGGCUUGUUGUCAACCAGGACGAUAACCACGGAAUUG---UUGUUAUCACUCUGGUUGACAA---CCG-----AAUGUCAU--CUAUCCGGCGCAUCCACAUUUGUAUGGCCUCCA -..(((((.(((((((((((((..((((((.(((....))---).))))))..))))))))))))---)((-----(((((...--...............)))))))...)))))... ( -36.17, z-score = -2.96, R) >droAna3.scaffold_13335 2820794 102 + 3335858 -CUGGGCUUGUUGUCAACCAGAACGAUAACCACCGAGUUG---UUGUUAUCAUUCUGGUUGACAA---CCG-----AAUGUCAU--CUAUCCGGCCCAUCCAC---CGUAUGACCUCCA -.(((((..((((((((((((((.((((((.((......)---).)))))).)))))))))))))---)((-----.(((....--.))).))))))).....---............. ( -38.50, z-score = -5.09, R) >dp4.chrXL_group1a 7303137 102 - 9151740 -CUGGGCUUGUUGUCAACCAGAACGAUAACCACAGAGUUG---UUGUUAUCGCUCUGAUUGA---CAACCG-----AAUGUCAU--CUGUCCGGGCCAUCCGUCAGAUUGUUGCCA--- -...(((..((((((((.((((.(((((((.((......)---).))))))).)))).))))---))))..-----......((--(((..(((.....))).)))))....))).--- ( -38.00, z-score = -3.66, R) >droPer1.super_13 1921154 102 - 2293547 -CUGGGCUUGUUGUCAACCAGAACGAUAACCACAGAGUUG---UUGUUAUCGCUCUGAUUGA---CAACCG-----AAUGUCAU--CUGUCCGGGCCAUCCGUCAGAUUGUUGCCA--- -...(((..((((((((.((((.(((((((.((......)---).))))))).)))).))))---))))..-----......((--(((..(((.....))).)))))....))).--- ( -38.00, z-score = -3.66, R) >droWil1.scaffold_181096 5838924 102 + 12416693 -CUGGGUUUAUUGUCAACCAGAACGAUAACCACCGAAUUG---UUGUUAUCACUCUGAUUGACAA---CCG-----AAUGUCAU--CUGUCCUCUAUGGCUAUGGAUGAGCCACCA--- -...(((((((((((((.((((..((((((.((......)---).))))))..)))).)))))).---(((-----...(((((--..(....).)))))..))))))))))....--- ( -29.80, z-score = -2.55, R) >droMoj3.scaffold_6328 3613197 92 + 4453435 -CUGGGCUUGUUGUCAACCAGCACGAUAACCACCGAGUUG---UUGUUAUCGCUCUGGUUGACAACCGCCG-----AAUGUCAU--CUGUCCGGCGCAUCGAU---------------- -....((..((((((((((((..(((((((.((......)---).)))))))..)))))))))))).((((-----.((.....--..)).))))))......---------------- ( -37.30, z-score = -3.86, R) >droVir3.scaffold_13042 2774384 94 - 5191987 -CUGGGCUUGUUGUCAACCAGAACAAUCACAACCGAAUUG---UUGUUCUCGCUCUGAUUGACCGCCGCCG-----AAUGUCAU--CUGUCCGGCCCCUCGAUCA-------------- -..(((...((.(((((.((((.(....(((((......)---))))....).)))).))))).)).((((-----.((.....--..)).))))))).......-------------- ( -23.20, z-score = -0.65, R) >droGri2.scaffold_15203 5556355 89 - 11997470 -CUGGGCUUGUUGUCAACCAGAACGAUAACCACCGAAUUG---UUGUUAUCACUCUGAUUGACAA---CCG-----AAUGUCAU--CUGUCCGGCGCAUCCAC---------------- -((((((..((((((((.((((..((((((.((......)---).))))))..)))).)))))))---).(-----(......)--).)))))).........---------------- ( -27.90, z-score = -2.96, R) >anoGam1.chr2R 7791653 116 + 62725911 CAUGCACCCGUUCCUGGCUCG--UGGCUGCCAGUGAGCGGCGUUUGUGAGGGCUC-GAUUCGUAGCCACUGUUGGCACUGUUAUUGCUGUUCGUUCCAUUGCUACUAAUAGAACGCUCG ((((.(((((((((((((...--.....))))).)))))).)).)))).((((..-........((((....)))).((((((..((.............))...))))))...)))). ( -38.02, z-score = -0.37, R) >consensus _CUGGGCUUGUUGUCAACCAGAACGAUAACCACCGAAUUG___UUGUUAUCACUCUGAUUGACAA___CCG_____AAUGUCAU__CUGUCCGGCGCAUCCAUAUUUGUAUGGCCA___ .(((((............((((..((((((...............))))))..))))..((((................))))......)))))......................... ( -8.97 = -9.43 + 0.46)
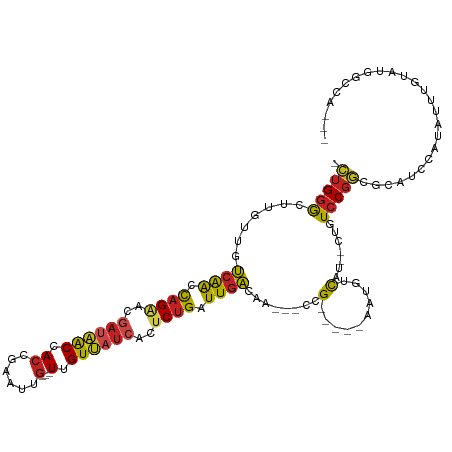
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:35:04 2011