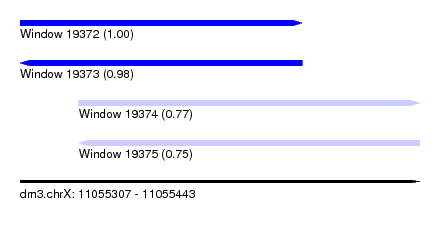
| Sequence ID | dm3.chrX |
|---|---|
| Location | 11,055,307 – 11,055,443 |
| Length | 136 |
| Max. P | 0.999198 |

| Location | 11,055,307 – 11,055,403 |
|---|---|
| Length | 96 |
| Sequences | 13 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 66.65 |
| Shannon entropy | 0.76554 |
| G+C content | 0.43328 |
| Mean single sequence MFE | -26.40 |
| Consensus MFE | -17.07 |
| Energy contribution | -16.95 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.47 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.65 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.70 |
| SVM RNA-class probability | 0.999198 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
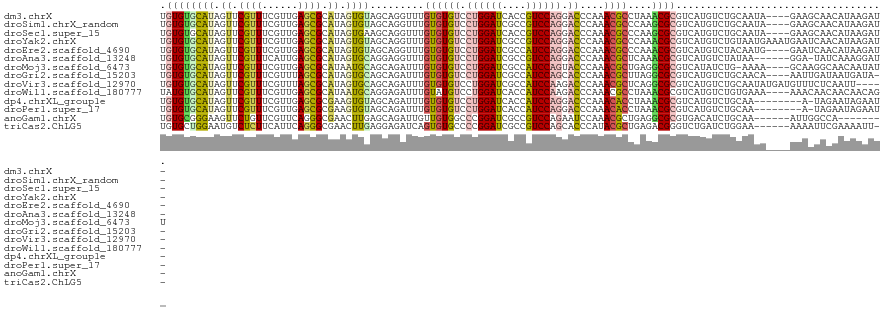
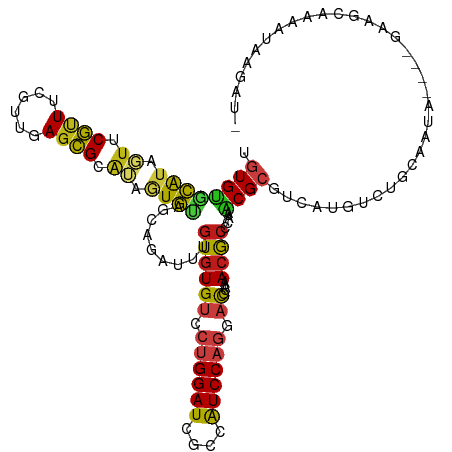
>dm3.chrX 11055307 96 + 22422827 UGUUUUGCAACUAGUAAUUGAUCUUAUGUUGCU-UC----UAUUGCAGACAUGACGCGUUUAGGCGUUUGGGUCCUGGACGGUGAUCCAGGACACACAAAC ...(((((((..((((((.........))))))-..----..)))))))((.(((((......))))))).((((((((......))))))))........ ( -29.60, z-score = -2.20, R) >droSim1.chrX_random 3114018 96 + 5698898 AGUUUUGCAAUUAGUAAUUGAUCUUAUGUUGCU-UC----UAUUGCAGACAUGACGCGCUUGGGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAAC ...((((((((.((((((.........))))))-..----.))))))))((.(((((......))))))).((((((((......))))))))........ ( -31.70, z-score = -2.72, R) >droSec1.super_15 1759170 96 + 1954846 AGUUUUGCAAUUAGUAAUUGAUCUUAUGUUGCU-UC----UAUUGCAGACAUGACGCGCUUGGGCGUUUGGGUCCUGGACGGUGAUCCAGGACACACAAAC ...((((((((.((((((.........))))))-..----.))))))))((.(((((......))))))).((((((((......))))))))........ ( -31.70, z-score = -2.71, R) >droYak2.chrX 10699047 99 - 21770863 -AAAUUACAAAUAGUAUUUAAUCUUAUGUUGAU-UCAUUUCAUUACAGACAUGACGCGUUUGGGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAAC -......(((((.(..((...(((.....(((.-.....)))....)))...))..))))))...(((((.((((((((......))))))))...))))) ( -24.10, z-score = -1.79, R) >droEre2.scaffold_4690 14673137 95 - 18748788 -GUAUUAUAAAUAGUAUUUAAUCUUAUGUUGAU-UC----CAUUGUAGACAUGACGCGUUUGGGCGUUUGGGUCCUGGAUGGCGAUCCAGGACACACAAAC -((((((....))))))......(((((((.((-..----....)).))))))).((......))(((((.(((((((((....)))))))))...))))) ( -26.00, z-score = -1.73, R) >droAna3.scaffold_13248 2600154 99 + 4840945 -AUUCUAUAACUU-UUGUUAUGAUAGUAAUCCUUUGAUAUCCUUAUAGACAUGACGCGUUUGAGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAAC -..(((((((...-.(((((.(((....)))...)))))...)))))))((.(((((......))))))).((((((((......))))))))........ ( -27.30, z-score = -2.41, R) >droMoj3.scaffold_6473 6685961 93 - 16943266 ---GUUAACUAUAAUAAUUGAAUAU-UGUUGCC-UUGCUUUU---CAGAUAUGACGCGCCUCAGCGUUUGGGUACUGGAUGGCGAUCCAGGACACACAAAU ---.....................(-(((((((-(((((.((---(((((..(((((......)))))...)).))))).))))....))).)).)))).. ( -21.80, z-score = -0.82, R) >droVir3.scaffold_12970 11104334 93 + 11907090 ---UUUAAAGGUUUUAUUUGAAUUGAGAAACAU-----CAUAUUGCAGACAUGACGCGCCUGAGCGUUUGGGUCUUGGAUGGCGAUCCAGGACACACAAAU ---.......(((...((((...(((......)-----)).....))))...)))((......))(((((.(((((((((....)))))))))...))))) ( -22.60, z-score = -1.20, R) >droWil1.scaffold_180777 103884 98 - 4753960 -AUGUAGAAUCUU-CAAUUGAUUGC-UGUUGUUGUUGUUUUUUCACAGACAUGACGCGUUUAGGCGUUUGGGUCUUGGAUGGUGAUCCAGGACAUACAAAU -.(((((((....-((((.....((-....)).))))....))).....((.(((((......))))))).(((((((((....))))))))).))))... ( -23.90, z-score = -0.55, R) >dp4.chrXL_group1e 9150800 92 - 12523060 --------GAAUU-CUACUAUUUCUUUAUUAUUCUAUUCUAUUUGCAGACAUGACGCGUUUAGGUGUUUGGGUCCUGGAUGGUGAUCCAGGACACACAAAU --------.....-.......................((((..(((.........)))..)))).(((((.(((((((((....)))))))))...))))) ( -23.00, z-score = -1.91, R) >droPer1.super_17 1620911 92 - 1930428 --------GAAUU-CUACUAUUUCUUUAUUAUUCUAUUCUAUUUGCAGACAUGACGCGUUUAGGUGUUUGGGUCCUGGAUGGUGAUCCAGGACACACAAAU --------.....-.......................((((..(((.........)))..)))).(((((.(((((((((....)))))))))...))))) ( -23.00, z-score = -1.91, R) >anoGam1.chrX 13146613 86 + 22145176 ---------------GGGCGGCGUUUUUUAAAUGUGGCCAAUUUGCAGAUGUCACGCGCCUCAGCGUUUGGAUUCUGGACGGCGAUCCGGGCCACAACAAU ---------------.................(((((((......((((..(((.((((....)))).)))..))))..(((....))))))))))..... ( -28.00, z-score = -0.42, R) >triCas2.ChLG5 3284573 99 - 18847211 -GUGUCGAUUCCGGUCAUUUUUUAUUGAAUUUU-CGAAUUUUUUCCAGAUCAGACCCGUCUCAGCGUAUGGGUGCUGGACGGCGAUCCGGGGCACACUGAU -((((((((((.((..((((......))))..)-))))))....((.((((....(((((.(((((......)))))))))).)))).)))))))...... ( -30.50, z-score = -1.13, R) >consensus __UUUUAAAAAUAGUAAUUGAUUUUAUGUUGCU_UC___UUAUUGCAGACAUGACGCGUUUGGGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAAU .................................................((.(((((......))))))).((((((((......))))))))........ (-17.07 = -16.95 + -0.13)
| Location | 11,055,307 – 11,055,403 |
|---|---|
| Length | 96 |
| Sequences | 13 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 66.65 |
| Shannon entropy | 0.76554 |
| G+C content | 0.43328 |
| Mean single sequence MFE | -23.23 |
| Consensus MFE | -7.59 |
| Energy contribution | -7.75 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.33 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.981068 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 11055307 96 - 22422827 GUUUGUGUGUCCUGGAUCACCGUCCAGGACCCAAACGCCUAAACGCGUCAUGUCUGCAAUA----GA-AGCAACAUAAGAUCAAUUACUAGUUGCAAAACA (((((.(.(((((((((....)))))))))))))))........(((.......)))....----..-.(((((.(((......)))...)))))...... ( -26.70, z-score = -2.84, R) >droSim1.chrX_random 3114018 96 - 5698898 GUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCCCAAGCGCGUCAUGUCUGCAAUA----GA-AGCAACAUAAGAUCAAUUACUAAUUGCAAAACU (((((.(.(((((((((....)))))))))))))))((....))(((..((((.(((....----..-.))))))).((........))...)))...... ( -26.00, z-score = -2.43, R) >droSec1.super_15 1759170 96 - 1954846 GUUUGUGUGUCCUGGAUCACCGUCCAGGACCCAAACGCCCAAGCGCGUCAUGUCUGCAAUA----GA-AGCAACAUAAGAUCAAUUACUAAUUGCAAAACU (((((.(.(((((((((....)))))))))))))))((....))(((..((((.(((....----..-.))))))).((........))...)))...... ( -26.00, z-score = -2.75, R) >droYak2.chrX 10699047 99 + 21770863 GUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCCCAAACGCGUCAUGUCUGUAAUGAAAUGA-AUCAACAUAAGAUUAAAUACUAUUUGUAAUUU- (((((.(.(((((((((....)))))))))))))))((......)).((((.((......)).))))-.................(((.....)))....- ( -25.30, z-score = -2.87, R) >droEre2.scaffold_4690 14673137 95 + 18748788 GUUUGUGUGUCCUGGAUCGCCAUCCAGGACCCAAACGCCCAAACGCGUCAUGUCUACAAUG----GA-AUCAACAUAAGAUUAAAUACUAUUUAUAAUAC- .((((((((((((((((....)))))))))(((.((((......))))..((....)).))----).-....))))))).....................- ( -24.00, z-score = -2.85, R) >droAna3.scaffold_13248 2600154 99 - 4840945 GUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCUCAAACGCGUCAUGUCUAUAAGGAUAUCAAAGGAUUACUAUCAUAACAA-AAGUUAUAGAAU- (((((.(.(((((((((....)))))))))))))))((......))...((((((....)))))).............(((((((..-..))))).))..- ( -27.40, z-score = -3.06, R) >droMoj3.scaffold_6473 6685961 93 + 16943266 AUUUGUGUGUCCUGGAUCGCCAUCCAGUACCCAAACGCUGAGGCGCGUCAUAUCUG---AAAAGCAA-GGCAACA-AUAUUCAAUUAUUAUAGUUAAC--- .((((.(.((.((((((....)))))).))))))).(((...((...(((....))---)...))..-)))....-......................--- ( -18.60, z-score = -0.40, R) >droVir3.scaffold_12970 11104334 93 - 11907090 AUUUGUGUGUCCUGGAUCGCCAUCCAAGACCCAAACGCUCAGGCGCGUCAUGUCUGCAAUAUG-----AUGUUUCUCAAUUCAAAUAAAACCUUUAAA--- (((((((.(((.(((((....))))).))).)).......(((.(((((((((.....)))))-----)))).))).....)))))............--- ( -20.00, z-score = -1.70, R) >droWil1.scaffold_180777 103884 98 + 4753960 AUUUGUAUGUCCUGGAUCACCAUCCAAGACCCAAACGCCUAAACGCGUCAUGUCUGUGAAAAAACAACAACAACA-GCAAUCAAUUG-AAGAUUCUACAU- ...((((.(((.(((((....))))).))).(((((((......))))..((.((((...............)))-))).....)))-.......)))).- ( -16.16, z-score = -0.98, R) >dp4.chrXL_group1e 9150800 92 + 12523060 AUUUGUGUGUCCUGGAUCACCAUCCAGGACCCAAACACCUAAACGCGUCAUGUCUGCAAAUAGAAUAGAAUAAUAAAGAAAUAGUAG-AAUUC-------- .((((.(.(((((((((....)))))))))))))).................(((((..........................))))-)....-------- ( -19.07, z-score = -2.12, R) >droPer1.super_17 1620911 92 + 1930428 AUUUGUGUGUCCUGGAUCACCAUCCAGGACCCAAACACCUAAACGCGUCAUGUCUGCAAAUAGAAUAGAAUAAUAAAGAAAUAGUAG-AAUUC-------- .((((.(.(((((((((....)))))))))))))).................(((((..........................))))-)....-------- ( -19.07, z-score = -2.12, R) >anoGam1.chrX 13146613 86 - 22145176 AUUGUUGUGGCCCGGAUCGCCGUCCAGAAUCCAAACGCUGAGGCGCGUGACAUCUGCAAAUUGGCCACAUUUAAAAAACGCCGCCC--------------- .....((((((((((((((((((((((..........))).)))).))))..))))......))))))).................--------------- ( -25.10, z-score = -0.71, R) >triCas2.ChLG5 3284573 99 + 18847211 AUCAGUGUGCCCCGGAUCGCCGUCCAGCACCCAUACGCUGAGACGGGUCUGAUCUGGAAAAAAUUCG-AAAAUUCAAUAAAAAAUGACCGGAAUCGACAC- ....((((...(((((((((((((((((........)))).)))))...))))))))......((((-...(((........)))...))))....))))- ( -28.60, z-score = -2.12, R) >consensus AUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCCCAAACGCGUCAUGUCUGCAAAAA___GA_AGCAACAUAAAAUCAAUUACUAUUUGCAAAA__ ....((((((.((((((....)))))).))....))))............................................................... ( -7.59 = -7.75 + 0.17)

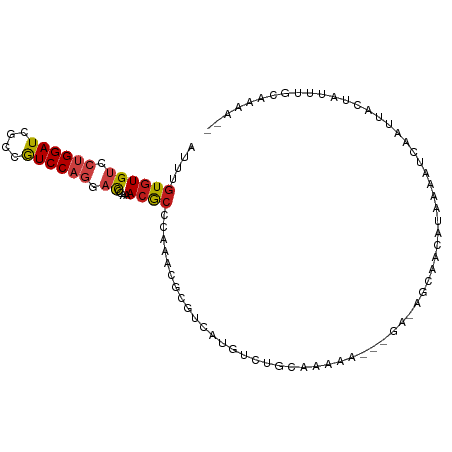
| Location | 11,055,327 – 11,055,443 |
|---|---|
| Length | 116 |
| Sequences | 14 |
| Columns | 121 |
| Reading direction | forward |
| Mean pairwise identity | 78.35 |
| Shannon entropy | 0.48920 |
| G+C content | 0.48474 |
| Mean single sequence MFE | -32.33 |
| Consensus MFE | -16.67 |
| Energy contribution | -16.59 |
| Covariance contribution | -0.09 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.52 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.770552 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
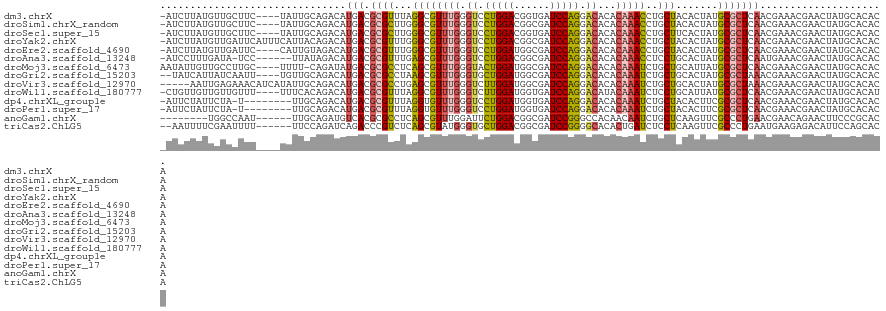
>dm3.chrX 11055327 116 + 22422827 -AUCUUAUGUUGCUUC----UAUUGCAGACAUGACGCGUUUAGGCGUUUGGGUCCUGGACGGUGAUCCAGGACACACAAACCUGCUACACUAUGCGCUCAACGAAACGAACUAUGCACACA -...(((((((((...----....))).))))))..(((((.((((((((.((((((((......))))))))...)))))..((........))))).....)))))............. ( -33.50, z-score = -1.97, R) >droSim1.chrX_random 3114038 116 + 5698898 -AUCUUAUGUUGCUUC----UAUUGCAGACAUGACGCGCUUGGGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAACCUGCUACACUAUGCGCUCAACGAAACGAACUAUGCACACA -...(((((((((...----....))).))))))..((.(((((((((((.((((((((......))))))))...)))))..((........)))))))))).................. ( -36.70, z-score = -2.92, R) >droSec1.super_15 1759190 116 + 1954846 -AUCUUAUGUUGCUUC----UAUUGCAGACAUGACGCGCUUGGGCGUUUGGGUCCUGGACGGUGAUCCAGGACACACAAACCUGCUUCACUAUGCGCUCAACGAAACGAACUAUGCACACA -...(((((((((...----....))).))))))..((.(((((((((((.((((((((......))))))))...)))))..((........)))))))))).................. ( -36.70, z-score = -2.65, R) >droYak2.chrX 10699066 120 - 21770863 -AUCUUAUGUUGAUUCAUUUCAUUACAGACAUGACGCGUUUGGGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAACCUGCUACACUAUGCGCUCAACGAAACGAACUAUGCACACA -......((((((.((((.((......)).))))(((((...((((((((.((((((((......))))))))...)))))..))).....))))).)))))).................. ( -35.20, z-score = -2.83, R) >droEre2.scaffold_4690 14673156 116 - 18748788 -AUCUUAUGUUGAUUC----CAUUGUAGACAUGACGCGUUUGGGCGUUUGGGUCCUGGAUGGCGAUCCAGGACACACAAACCUGCUACACUAUGCGCUCAACGAAACGAACUAUGCACACA -...(((((((.((..----....)).))))))).(((((((((((((((.(((((((((....)))))))))...)))))..((........))))))))...........))))..... ( -36.00, z-score = -2.40, R) >droAna3.scaffold_13248 2600180 113 + 4840945 -AUCCUUUGAUA-UCC------UUAUAGACAUGACGCGUUUGAGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAACCUCCUGCACUAUGCGCUCAAUGAAACGAACUAUGCACACA -...........-...------.....(.((((...((((((((((((((.((((((((......))))))))...))))).....((.....)))))))....))))...)))))..... ( -32.90, z-score = -2.68, R) >droMoj3.scaffold_6473 6685978 116 - 16943266 AAUAUUGUUGCCUUGC----UUUU-CAGAUAUGACGCGCCUCAGCGUUUGGGUACUGGAUGGCGAUCCAGGACACACAAAUCUGCUGCAUUAUGCGCUCAACGAAACGAACUAUGCACACA ....((((((......----...(-((....))).((((..(((((((((.((.((((((....)))))).))...)))))..))))......)))).))))))................. ( -27.30, z-score = 0.36, R) >droGri2.scaffold_15203 3082179 115 + 11997470 --UAUCAUUAUCAAUU----UGUUGCAGACAUGACGCGCCUAAGCGUUUGGGUGCUGGAUGGCGAUCCAGGACACACAAAUCUGCUGCACUAUGCGCUAAACGAAACGAACUAUGCACACA --...(((..((..((----(((((((((((.(((((......))))))).((.((((((....)))))).)).......))))).((.....))....))))))..))...)))...... ( -29.30, z-score = -0.54, R) >droVir3.scaffold_12970 11104351 116 + 11907090 -----AAUUGAGAAACAUCAUAUUGCAGACAUGACGCGCCUGAGCGUUUGGGUCUUGGAUGGCGAUCCAGGACACACAAAUCUGCUGCACUAUGCGCUAAACGAAACGAACUAUGCACACA -----.............((((.(((((.((.(((((......)))((((.(((((((((....)))))))))...))))))))))))).)))).((.................))..... ( -30.13, z-score = -1.24, R) >droWil1.scaffold_180777 103906 116 - 4753960 -CUGUUGUUGUUGUUU----UUUCACAGACAUGACGCGUUUAGGCGUUUGGGUCUUGGAUGGUGAUCCAGGACAUACAAAUCUCCUGCAUUAUGCGCUCAACGAAACGAACUAUGCACAUA -.(((.((((((((((----......)))).((((((((.((((.(((((.(((((((((....)))))))))...)))).).))))....))))).)))....))).)))...))).... ( -31.20, z-score = -0.84, R) >dp4.chrXL_group1e 9150821 111 - 12523060 -AUUCUAUUCUA-U--------UUGCAGACAUGACGCGUUUAGGUGUUUGGGUCCUGGAUGGUGAUCCAGGACACACAAAUCUGCUACACUUCGCGCUCAACGAAACGAACUAUGCACACA -...........-.--------.((((....(((((((...(((((((((.(((((((((....)))))))))...))).......)))))))))).))).((...)).....)))).... ( -30.21, z-score = -1.69, R) >droPer1.super_17 1620932 111 - 1930428 -AUUCUAUUCUA-U--------UUGCAGACAUGACGCGUUUAGGUGUUUGGGUCCUGGAUGGUGAUCCAGGACACACAAAUCUGCUACACUUCGCGCUCAACGAAACGAACUAUGCACACA -...........-.--------.((((....(((((((...(((((((((.(((((((((....)))))))))...))).......)))))))))).))).((...)).....)))).... ( -30.21, z-score = -1.69, R) >anoGam1.chrX 13146632 107 + 22145176 --------UGGCCAAU------UUGCAGAUGUCACGCGCCUCAGCGUUUGGAUUCUGGACGGCGAUCCGGGCCACAACAAUCUGCUCAAGUUCGCCCUGAACGAACAGAACUUCCCGCACA --------(((((...------...((((..(((.((((....)))).)))..))))..(((....))))))))......((((.((..((((.....)))))).))))............ ( -29.80, z-score = 0.19, R) >triCas2.ChLG5 3284599 113 - 18847211 --AAUUUUCGAAUUUU------UUCCAGAUCAGACCCGUCUCAGCGUAUGGGUGCUGGACGGCGAUCCGGGGCACACUGAUCUCCUCAAGUUCGCCCUGAAUGAAGAGACAUUCCAGCACA --.......(((((((------(((..((((....(((((.(((((......)))))))))).))))((((((..(((((.....)).)))..))))))...)))))))..)))....... ( -33.50, z-score = -0.65, R) >consensus _AUCUUAUUGUGAUUC____UAUUGCAGACAUGACGCGUUUGGGCGUUUGGGUCCUGGACGGCGAUCCAGGACACACAAAUCUGCUACACUAUGCGCUCAACGAAACGAACUAUGCACACA ...............................(((.((((...((((((((.((.(((((......))))).))...)))))..))).......)))))))..................... (-16.67 = -16.59 + -0.09)

| Location | 11,055,327 – 11,055,443 |
|---|---|
| Length | 116 |
| Sequences | 14 |
| Columns | 121 |
| Reading direction | reverse |
| Mean pairwise identity | 78.35 |
| Shannon entropy | 0.48920 |
| G+C content | 0.48474 |
| Mean single sequence MFE | -35.51 |
| Consensus MFE | -16.16 |
| Energy contribution | -15.59 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.54 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.46 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.752287 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 11055327 116 - 22422827 UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAGUGUAGCAGGUUUGUGUGUCCUGGAUCACCGUCCAGGACCCAAACGCCUAAACGCGUCAUGUCUGCAAUA----GAAGCAACAUAAGAU- (((((((...(((((......)))))..........(((((((((.(.(((((((((....)))))))))))))))((......)).......))))....----...)).)))))....- ( -38.20, z-score = -2.60, R) >droSim1.chrX_random 3114038 116 - 5698898 UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAGUGUAGCAGGUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCCCAAGCGCGUCAUGUCUGCAAUA----GAAGCAACAUAAGAU- (((((((...(((((......)))))(((..(((..((..(((((.(.(((((((((....)))))))))))))))((....))))..)))...)))....----...)).)))))....- ( -40.10, z-score = -2.87, R) >droSec1.super_15 1759190 116 - 1954846 UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAGUGAAGCAGGUUUGUGUGUCCUGGAUCACCGUCCAGGACCCAAACGCCCAAGCGCGUCAUGUCUGCAAUA----GAAGCAACAUAAGAU- (((((((...(((((......)))))(((..((((.((..(((((.(.(((((((((....)))))))))))))))((....)))).))))...)))....----...)).)))))....- ( -42.30, z-score = -3.81, R) >droYak2.chrX 10699066 120 + 21770863 UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAGUGUAGCAGGUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCCCAAACGCGUCAUGUCUGUAAUGAAAUGAAUCAACAUAAGAU- (((((.....(((((((((((((...((((.((((.((..(((((.(.(((((((((....)))))))))))))))))....))))...))))...)))))))))))))..)))))....- ( -42.00, z-score = -3.86, R) >droEre2.scaffold_4690 14673156 116 + 18748788 UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAGUGUAGCAGGUUUGUGUGUCCUGGAUCGCCAUCCAGGACCCAAACGCCCAAACGCGUCAUGUCUACAAUG----GAAUCAACAUAAGAU- ..........(((.(((((((((((.((((.((((.((..(((((.(.(((((((((....)))))))))))))))))....))))...)))))).)))))----)))).))).......- ( -37.20, z-score = -2.31, R) >droAna3.scaffold_13248 2600180 113 - 4840945 UGUGUGCAUAGUUCGUUUCAUUGAGCGCAUAGUGCAGGAGGUUUGUGUGUCCUGGAUCGCCGUCCAGGACCCAAACGCUCAAACGCGUCAUGUCUAUAA------GGA-UAUCAAAGGAU- .((((((((.((.(((((....))))).)).))))..((((((((.(.(((((((((....))))))))))))))).)))..))))...((((((....------)))-)))........- ( -39.40, z-score = -2.78, R) >droMoj3.scaffold_6473 6685978 116 + 16943266 UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAAUGCAGCAGAUUUGUGUGUCCUGGAUCGCCAUCCAGUACCCAAACGCUGAGGCGCGUCAUAUCUG-AAAA----GCAAGGCAACAAUAUU (((.(((...((...(((((.(((((((......((((.(.((((.(.((.((((((....)))))).))))))))))))..)))).)))....))-))).----))...))))))..... ( -31.40, z-score = -0.07, R) >droGri2.scaffold_15203 3082179 115 - 11997470 UGUGUGCAUAGUUCGUUUCGUUUAGCGCAUAGUGCAGCAGAUUUGUGUGUCCUGGAUCGCCAUCCAGCACCCAAACGCUUAGGCGCGUCAUGUCUGCAACA----AAUUGAUAAUGAUA-- (((.(((((.((.((((......)))).)).)))))((((((.((((((.(((((((....))))(((........))).))))))).)).)))))).)))----..............-- ( -32.30, z-score = -0.43, R) >droVir3.scaffold_12970 11104351 116 - 11907090 UGUGUGCAUAGUUCGUUUCGUUUAGCGCAUAGUGCAGCAGAUUUGUGUGUCCUGGAUCGCCAUCCAAGACCCAAACGCUCAGGCGCGUCAUGUCUGCAAUAUGAUGUUUCUCAAUU----- ....(((((.((.((((......)))).)).)))))((((((.((((.(((.(((((....))))).))).)).((((......)))))).))))))...................----- ( -31.70, z-score = -0.62, R) >droWil1.scaffold_180777 103906 116 + 4753960 UAUGUGCAUAGUUCGUUUCGUUGAGCGCAUAAUGCAGGAGAUUUGUAUGUCCUGGAUCACCAUCCAAGACCCAAACGCCUAAACGCGUCAUGUCUGUGAAA----AAACAACAACAACAG- ..(((((...(((((......))))))))))...(((((.((....)).)))))..((((......((((....((((......))))...))))))))..----...............- ( -25.20, z-score = -0.35, R) >dp4.chrXL_group1e 9150821 111 + 12523060 UGUGUGCAUAGUUCGUUUCGUUGAGCGCGAAGUGUAGCAGAUUUGUGUGUCCUGGAUCACCAUCCAGGACCCAAACACCUAAACGCGUCAUGUCUGCAA--------A-UAGAAUAGAAU- ....(((((.(((((......))))).....)))))((((((.((((((((((((((....)))))))))............)))))....))))))..--------.-...........- ( -31.80, z-score = -1.96, R) >droPer1.super_17 1620932 111 + 1930428 UGUGUGCAUAGUUCGUUUCGUUGAGCGCGAAGUGUAGCAGAUUUGUGUGUCCUGGAUCACCAUCCAGGACCCAAACACCUAAACGCGUCAUGUCUGCAA--------A-UAGAAUAGAAU- ....(((((.(((((......))))).....)))))((((((.((((((((((((((....)))))))))............)))))....))))))..--------.-...........- ( -31.80, z-score = -1.96, R) >anoGam1.chrX 13146632 107 - 22145176 UGUGCGGGAAGUUCUGUUCGUUCAGGGCGAACUUGAGCAGAUUGUUGUGGCCCGGAUCGCCGUCCAGAAUCCAAACGCUGAGGCGCGUGACAUCUGCAA------AUUGGCCA-------- .....((.((((((.((((.....))))))))))..((((((.((..((((((((....)))..(((..........))).))).))..))))))))..------.....)).-------- ( -33.70, z-score = 0.69, R) >triCas2.ChLG5 3284599 113 + 18847211 UGUGCUGGAAUGUCUCUUCAUUCAGGGCGAACUUGAGGAGAUCAGUGUGCCCCGGAUCGCCGUCCAGCACCCAUACGCUGAGACGGGUCUGAUCUGGAA------AAAAUUCGAAAAUU-- .(..(((....((((((((((((.....)))..))))))))))))..)...(((((((((((((((((........)))).)))))...))))))))..------..............-- ( -40.00, z-score = -2.03, R) >consensus UGUGUGCAUAGUUCGUUUCGUUGAGCGCAUAGUGUAGCAGAUUUGUGUGUCCUGGAUCGCCAUCCAGGACCCAAACGCCCAAACGCGUCAUGUCUGCAAUA____GAAGCAAAAUAAGAU_ .((((((((.((.((((......)))).)).)))).........((((((.((((((....)))))).))....))))....))))................................... (-16.16 = -15.59 + -0.58)
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:33:23 2011