
| Sequence ID | dm3.chrX |
|---|---|
| Location | 10,942,020 – 10,942,127 |
| Length | 107 |
| Max. P | 0.997843 |

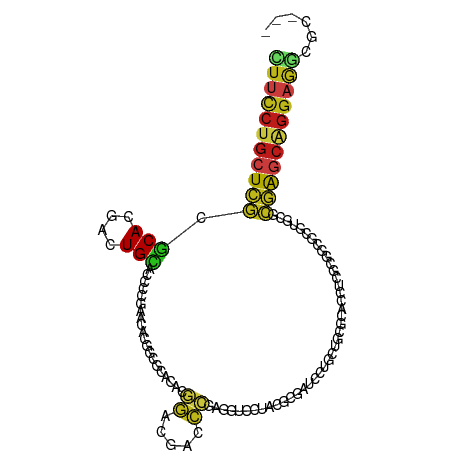
| Location | 10,942,020 – 10,942,127 |
|---|---|
| Length | 107 |
| Sequences | 14 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 72.49 |
| Shannon entropy | 0.64386 |
| G+C content | 0.65502 |
| Mean single sequence MFE | -48.75 |
| Consensus MFE | -16.05 |
| Energy contribution | -15.94 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.75 |
| Mean z-score | -3.22 |
| Structure conservation index | 0.33 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.19 |
| SVM RNA-class probability | 0.997843 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 10942020 107 + 22422827 CUUCCUGCUCGCGCACGACUGCACACCGAACACGGCGCACACGGACGACCCGAGCUCCUUCGAGAUCCUGCUGCGCACCUCGCGGCGCGUGCGCGAGCGGGAAGCGC--- (((((((((((((((((.((((....((....))(((((((.(((...(.((((....)))).).))))).))))).....))))..)))))))))))))))))...--- ( -54.80, z-score = -3.87, R) >droSim1.chrX 8496317 107 + 17042790 CUUCCUGCUCGCGCACGACUGCACACCGAACACGGCGCACACGGACGACCCGAGGUCCUUCGAGAUCCUGCUGCGCACCUCGCGGCGCGUGCGCGAGCGGGAAGCGC--- (((((((((((((((((..(((...(((....))).)))..((((.((((...)))).)))).......((((((.....)))))).)))))))))))))))))...--- ( -56.40, z-score = -3.86, R) >droSec1.super_15 1653775 107 + 1954846 CUUCCUGCUCGCGCACGACUGCACACCGAACACGGCGCACACGGACGACCCGAGGUCCUUCGAGAUCCUGCUGCGCACCUCGCGGCGCGUGCGCGAGCGGGAAGCGC--- (((((((((((((((((..(((...(((....))).)))..((((.((((...)))).)))).......((((((.....)))))).)))))))))))))))))...--- ( -56.40, z-score = -3.86, R) >droYak2.chrX 10594926 107 - 21770863 CUUCCUGCUCGCGCACGACUGCACACCGAACACGGCGCACACGGACGACCCGAGGUCCUUCGAGAUCCUGCUGCGCACCUCGCGGCGCGUGCGCGAGCGGGAAGCGC--- (((((((((((((((((..(((...(((....))).)))..((((.((((...)))).)))).......((((((.....)))))).)))))))))))))))))...--- ( -56.40, z-score = -3.86, R) >droEre2.scaffold_4690 14566802 107 - 18748788 CUUCCUGCUCGCGCACGACUGCACACCGAACACGGCGCACACGGACGACCCGAGGUCCUUCGAGAUCCUGCUGCGCACCUCGCGGCGCGUGCGCGAGCGGGAAGCGC--- (((((((((((((((((..(((...(((....))).)))..((((.((((...)))).)))).......((((((.....)))))).)))))))))))))))))...--- ( -56.40, z-score = -3.86, R) >droAna3.scaffold_13417 5986539 107 - 6960332 CUUCCUGCUCGCGCACGACUGCUCCCCGAAUACGGCGCACACCGAUCACCCGGAGUCCUACGCCAUCAUUUUGAGGACAUCGCGGCGCGUGCGCGAGCGGGAAGCCC--- (((((((((((((((((.(((((((.((((...((((.((.(((......))).))....)))).....)))).)))....))))..)))))))))))))))))...--- ( -54.40, z-score = -5.22, R) >dp4.chrXL_group1e 813316 107 + 12523060 CUUCCUGCUCGCGCACGACUGCAGCCCGAACACUGCGCACACGGACGACCCCCGGUCCUACGCCAUCCUGUUGAGGACAUCGAGGCGCGUGCGCGAGCAGGAGGCUU--- (((((((((((((((((......((((((............(((.......)))(((((..((......))..))))).))).))).)))))))))))))))))...--- ( -53.70, z-score = -4.29, R) >droPer1.super_30 570017 107 - 958394 CUUCCUGCUCGCGCACGACUGCAGCCCGAACACUGCGCACACGGACGACCCCCGGUCCUACGCCAUCCUGUUGAGGACAUCGAGGCGCGUGCGCGAGCAGGAGGCUU--- (((((((((((((((((......((((((............(((.......)))(((((..((......))..))))).))).))).)))))))))))))))))...--- ( -53.70, z-score = -4.29, R) >droWil1.scaffold_180702 997838 107 + 4511350 CUUUCUGCUCGCGCACGACUGCAGUCCGAACACGGCGCACACCGAUGAUCCCGGCACAUAUGCAAUUUUGCUGCGCACUUCGCGGCGCGUGCACGAGCAGGAGGCGU--- (((((((((((.(((((..(((((((((..(((((......))).))....))).))...)))).....((((((.....)))))).))))).)))))))))))...--- ( -46.60, z-score = -2.20, R) >droVir3.scaffold_13042 2012974 107 - 5191987 GUUCCUGCUCGCCCACGACUGCAGCCCCAACACGGCACACACGGACGAUCCACUCAGCUAUGCCAUCCUGUUGCGCACAUCGCGCGCCGUACGCGAACAGGAGACUC--- .((((((.((((..(((...((((((.......)))......((.....)).........))).........((((.....))))..)))..)))).))))))....--- ( -31.80, z-score = -0.93, R) >droMoj3.scaffold_6328 837919 107 - 4453435 CUUUCUGCUCGCGCACGACUGCCGACCAAACACAUCGCACACGGACGAUCCACUGGACUACUCGAUCAUCCUGCGGACGUCCCGUUCGGUGCGCGAGCAGGAGACAC--- .(((((((((((((((((((((.((.........)))))...(((((.(((.(.(((...........))).).)))))))).)))..)))))))))))))))....--- ( -43.90, z-score = -4.05, R) >droGri2.scaffold_15081 3080640 107 + 4274704 GUUCCUGCUCGCGCACGAUUGCACCCCGAACACGGCGCACACGGAUGACCCAGCCACGUUUGCCAUCCUGCUGCGUACGUCGCGACGCGUGCUCGAGCAGGAGCCGU--- (((((((((((.(((((.((((...(((....)))((((((.(((((...(((......))).))))))).))))......))))..))))).)))))))))))...--- ( -48.40, z-score = -2.89, R) >anoGam1.chrX 2429279 105 - 22145176 UGCGAUGCUGUCGCACGACUGUCGCCCGAACACGAAGCACUAUUUUGACGACCGGCUGCAUAUGGUGCUGGUGGCGAC-----GGUCGACAUCCCGGCCGGUGGUGUCAU ((((((...)))))).((((((((((...((....((((((((..((.((......)))).)))))))).))))))))-----))))((((((((....)).)))))).. ( -46.30, z-score = -2.06, R) >triCas2.ChLG8 2199690 107 + 15773733 CUUCCUAAUGUCACACGAUUGUGUCCCUAACACGAAUCACAUUGACGAAGAAAGUACCUUCAGGUUAACAGUUCGGGCCUC--GACUAGGAUAGAACCUGGCGAAAUGA- ..(((...(((((...((((((((.....)))).))))....)))))..(((...(((....)))......))))))..((--(.(((((......)))))))).....- ( -23.30, z-score = 0.20, R) >consensus CUUCCUGCUCGCGCACGACUGCACCCCGAACACGGCGCACACGGACGACCCGAGGUCCUACGCGAUCCUGCUGCGCACCUCGCGGCGCGUGCGCGAGCAGGAGGCGC___ (((((((((((.(((....)))....................((.....))..........................................)))))))))))...... (-16.05 = -15.94 + -0.11)

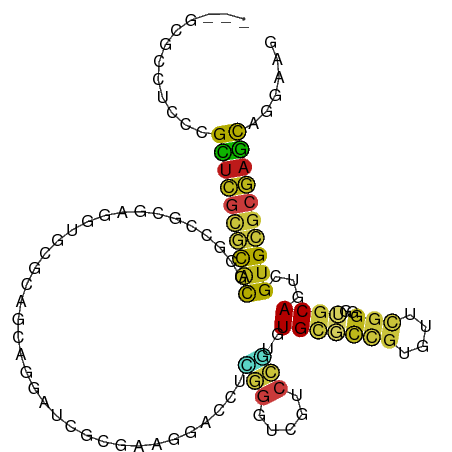
| Location | 10,942,020 – 10,942,127 |
|---|---|
| Length | 107 |
| Sequences | 14 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 72.49 |
| Shannon entropy | 0.64386 |
| G+C content | 0.65502 |
| Mean single sequence MFE | -53.46 |
| Consensus MFE | -20.47 |
| Energy contribution | -20.58 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.90 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.52 |
| SVM RNA-class probability | 0.992137 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 10942020 107 - 22422827 ---GCGCUUCCCGCUCGCGCACGCGCCGCGAGGUGCGCAGCAGGAUCUCGAAGGAGCUCGGGUCGUCCGUGUGCGCCGUGUUCGGUGUGCAGUCGUGCGCGAGCAGGAAG ---...(((((.(((((((((((((((....)))))...((.(((((((((......)))))..)))).((..(((((....)))))..)))).)))))))))).))))) ( -63.30, z-score = -3.52, R) >droSim1.chrX 8496317 107 - 17042790 ---GCGCUUCCCGCUCGCGCACGCGCCGCGAGGUGCGCAGCAGGAUCUCGAAGGACCUCGGGUCGUCCGUGUGCGCCGUGUUCGGUGUGCAGUCGUGCGCGAGCAGGAAG ---...(((((.(((((((((((((((....)))))...((.(((((((((......)))))..)))).((..(((((....)))))..)))).)))))))))).))))) ( -63.30, z-score = -3.64, R) >droSec1.super_15 1653775 107 - 1954846 ---GCGCUUCCCGCUCGCGCACGCGCCGCGAGGUGCGCAGCAGGAUCUCGAAGGACCUCGGGUCGUCCGUGUGCGCCGUGUUCGGUGUGCAGUCGUGCGCGAGCAGGAAG ---...(((((.(((((((((((((((....)))))...((.(((((((((......)))))..)))).((..(((((....)))))..)))).)))))))))).))))) ( -63.30, z-score = -3.64, R) >droYak2.chrX 10594926 107 + 21770863 ---GCGCUUCCCGCUCGCGCACGCGCCGCGAGGUGCGCAGCAGGAUCUCGAAGGACCUCGGGUCGUCCGUGUGCGCCGUGUUCGGUGUGCAGUCGUGCGCGAGCAGGAAG ---...(((((.(((((((((((((((....)))))...((.(((((((((......)))))..)))).((..(((((....)))))..)))).)))))))))).))))) ( -63.30, z-score = -3.64, R) >droEre2.scaffold_4690 14566802 107 + 18748788 ---GCGCUUCCCGCUCGCGCACGCGCCGCGAGGUGCGCAGCAGGAUCUCGAAGGACCUCGGGUCGUCCGUGUGCGCCGUGUUCGGUGUGCAGUCGUGCGCGAGCAGGAAG ---...(((((.(((((((((((((((....)))))...((.(((((((((......)))))..)))).((..(((((....)))))..)))).)))))))))).))))) ( -63.30, z-score = -3.64, R) >droAna3.scaffold_13417 5986539 107 + 6960332 ---GGGCUUCCCGCUCGCGCACGCGCCGCGAUGUCCUCAAAAUGAUGGCGUAGGACUCCGGGUGAUCGGUGUGCGCCGUAUUCGGGGAGCAGUCGUGCGCGAGCAGGAAG ---...(((((.(((((((((((.((.((.(((((.((.....)).)))))....(((((((((..((((....))))))))))))).)).))))))))))))).))))) ( -55.90, z-score = -2.94, R) >dp4.chrXL_group1e 813316 107 - 12523060 ---AAGCCUCCUGCUCGCGCACGCGCCUCGAUGUCCUCAACAGGAUGGCGUAGGACCGGGGGUCGUCCGUGUGCGCAGUGUUCGGGCUGCAGUCGUGCGCGAGCAGGAAG ---.....(((((((((((((((((((.....(((((....)))))))))).(.((((((.....)))).)).)(((((......)))))....)))))))))))))).. ( -55.90, z-score = -2.72, R) >droPer1.super_30 570017 107 + 958394 ---AAGCCUCCUGCUCGCGCACGCGCCUCGAUGUCCUCAACAGGAUGGCGUAGGACCGGGGGUCGUCCGUGUGCGCAGUGUUCGGGCUGCAGUCGUGCGCGAGCAGGAAG ---.....(((((((((((((((((((.....(((((....)))))))))).(.((((((.....)))).)).)(((((......)))))....)))))))))))))).. ( -55.90, z-score = -2.72, R) >droWil1.scaffold_180702 997838 107 - 4511350 ---ACGCCUCCUGCUCGUGCACGCGCCGCGAAGUGCGCAGCAAAAUUGCAUAUGUGCCGGGAUCAUCGGUGUGCGCCGUGUUCGGACUGCAGUCGUGCGCGAGCAGAAAG ---.......((((((((((((.((.((((..((((((.(((....)))......(((((.....)))))))))))))))..))(((....))))))))))))))).... ( -48.30, z-score = -1.33, R) >droVir3.scaffold_13042 2012974 107 + 5191987 ---GAGUCUCCUGUUCGCGUACGGCGCGCGAUGUGCGCAACAGGAUGGCAUAGCUGAGUGGAUCGUCCGUGUGUGCCGUGUUGGGGCUGCAGUCGUGGGCGAGCAGGAAC ---.....((((((((((.((((((((((...))))((..(((.((((((((.(....(((.....))).))))))))).)))..))....)))))).)))))))))).. ( -53.00, z-score = -3.04, R) >droMoj3.scaffold_6328 837919 107 + 4453435 ---GUGUCUCCUGCUCGCGCACCGAACGGGACGUCCGCAGGAUGAUCGAGUAGUCCAGUGGAUCGUCCGUGUGCGAUGUGUUUGGUCGGCAGUCGUGCGCGAGCAGAAAG ---.......(((((((((((((((.(((((((((((((((((((((.(.(.....).).)))))))).)))).))))).)))).))))......))))))))))).... ( -50.90, z-score = -3.35, R) >droGri2.scaffold_15081 3080640 107 - 4274704 ---ACGGCUCCUGCUCGAGCACGCGUCGCGACGUACGCAGCAGGAUGGCAAACGUGGCUGGGUCAUCCGUGUGCGCCGUGUUCGGGGUGCAAUCGUGCGCGAGCAGGAAC ---.....(((((((((.(((((.((.(((.....))).)).(((((((...((....)).))))))).((..(.(((....))).)..))..))))).))))))))).. ( -52.20, z-score = -1.52, R) >anoGam1.chrX 2429279 105 + 22145176 AUGACACCACCGGCCGGGAUGUCGACC-----GUCGCCACCAGCACCAUAUGCAGCCGGUCGUCAAAAUAGUGCUUCGUGUUCGGGCGACAGUCGUGCGACAGCAUCGCA (((((.....((((.((((((.....)-----))).))....(((.....))).))))((((((..((((........))))..)))))).)))))((((.....)))). ( -33.70, z-score = 0.83, R) >triCas2.ChLG8 2199690 107 - 15773733 -UCAUUUCGCCAGGUUCUAUCCUAGUC--GAGGCCCGAACUGUUAACCUGAAGGUACUUUCUUCGUCAAUGUGAUUCGUGUUAGGGACACAAUCGUGUGACAUUAGGAAG -...........(((((...(((....--.)))...))))).....((((((((.....)))))(((.(..(((((.(((((...))))))))))..))))...)))... ( -26.10, z-score = -0.03, R) >consensus ___GCGCCUCCCGCUCGCGCACGCGCCGCGAGGUGCGCAGCAGGAUCGCGAAGGACCUCGGGUCGUCCGUGUGCGCCGUGUUCGGGGUGCAGUCGUGCGCGAGCAGGAAG ............((((((((((....................................(((.....)))..(((((((....)))..))))...))))))))))...... (-20.47 = -20.58 + 0.11)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:33:06 2011