
| Sequence ID | dm3.chrX |
|---|---|
| Location | 10,495,307 – 10,495,427 |
| Length | 120 |
| Max. P | 0.514779 |

| Location | 10,495,307 – 10,495,427 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.02 |
| Shannon entropy | 0.59929 |
| G+C content | 0.55093 |
| Mean single sequence MFE | -45.78 |
| Consensus MFE | -16.45 |
| Energy contribution | -16.09 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.60 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.514779 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 10495307 120 - 22422827 UUUUUGAGCACAUACCGCAAGGAUGGCGUACUGCGCGGCUUGUACGCCGGUUCCGUUCCGGCCGUAUUCGCCAAUGUGGCCGAGAAUUCGGUGCUGUUCGCCGCCUACGGUGGAUGCCAG ....((.(((..(((((..(((.(((((.((.((..(((..(((((((((.......)))).)))))..)))......((((......)))))).)).)))))))).)))))..))))). ( -52.60, z-score = -2.28, R) >droGri2.scaffold_15203 7080445 120 - 11997470 UUCGUUAACACAUAUCGAAAGGAUGGCGUUAUGCAUGGCCUAUAUGCCGGAUCGGUGCCGGCUGUCGUUGCAAAUGUUGCCGAAAAUUCCGUACUCUUUGCCGCCUACGGUGGCUGCCAA ...((((((.(((.((....)))))..)))).)).((((........(((((((((..((..(((....)))..))..)))))....))))........((((((...)))))).)))). ( -35.50, z-score = 0.41, R) >droMoj3.scaffold_6482 2200884 120 - 2735782 UUUUUCAGCACCUACCGAAAAGAUGGGGUCAUGCGGGGUCUGUAUGCCGGCUCAUUGCCGGCGGUAUUUGCGAAUGUGGCAGAAAACUCGGUGCUCUUUGCUGCCUAUGGUGGCUGCCAG ...(((.(((..((((......(((((.((.....)).)))))..((((((.....))))))))))..))))))..((((((...(((.(((((.....)).)))...)))..)))))). ( -43.40, z-score = -0.68, R) >droVir3.scaffold_12970 4374631 120 + 11907090 UUUAUUAGCACCUAUCGCAGAGAUGGCGUCAUGCGGGGCCUGUACGCUGGCUCUGUGCCGGCGGUCUUUGCAAAUGUUGCCGAAAAUUCAGUGCUCUUUGCCGCCUACGGUGGCUGCCAA ......(((((.....(((((((((((.((....)).)))....(((((((.....)))))))))))))))..........((....)).)))))....((((((...))))))...... ( -46.30, z-score = -1.12, R) >droWil1.scaffold_180702 3752720 120 - 4511350 UUUGUAAAGACAUAUCAGAAGGAUGGCUUUAUGCGUGGUCUUUACGCUGGCUCUGUGCCCGCCGUAUUUGCCAAUGUGGCCGAAAAUUCAGUUCUAUUUGCCGCCUAUGGAGGCUGCCAG ...((((((((.((((.....))))((.....))...)))))))).(((((.....(((..(((((...((.((((..((.((....)).))..)))).))....))))).))).))))) ( -34.60, z-score = -0.08, R) >droAna3.scaffold_13117 4498932 120 - 5790199 UUUGUGAAAACAUAUCGAAAGGAUGGUUUCUUUCGGGGAUUAUAUGCCGGAUCGGUGCCGGCUGUUUUUGCCAAUGUCGCUGAGAAUUCCGUAUUAUUUGCGGCAUACGGCGGAUGUCAG ...(..((((((..(((((((((....))))))))).........(((((.......)))))))))))..).....((((((...((.(((((.....))))).)).))))))....... ( -38.00, z-score = -1.35, R) >droEre2.scaffold_4690 14114379 120 + 18748788 UUUGUGAGCACAUACCGCAAGGAUGGCGUACUGCACGGAUUGUACGCCGGAUCCGUUCCGGCCGUAUUCGCCAAUGUGGCCGAGAACUCGGUGCUGUUCGCCGCCUACGGCGGAUGCCAG ......(((((...((....)).((((((((..........)))))))).....((((((((((((((....)))))))))).))))...)))))((((((((....))))))))..... ( -52.00, z-score = -2.16, R) >droYak2.chrX 19064252 120 - 21770863 UUUGUGAGCACAUAUCGCAAGGAUGGCGUCCUCCAUGGCUUGUACGCCGGUUCCGUUCCGGCCGUAUUCGCCAAUGUGGCCGAAAACUCGGUGCUAUUUGCCGCCUACGGUGGAUGCCAG (((((((.......)))))))..((((((((.((.((((..(((((((((.......)))).)))))..))))..((((((((....)))))((.....)))))....)).)))))))). ( -52.40, z-score = -3.33, R) >droSec1.super_15 1199571 120 - 1954846 UUUUUGAGCACAUACCGCAAGGAUGGCGUCCUGCGCGGCUUGUACGCCGGUUCCGUUCCGGCCGUAUUCGCCAAUGUGGCCGAGAACUCGGUGCUGUUCGCCGCCUACGGUGGAUGCCAG ....((.(((..(((((..(((.(((((..(.((..(((..(((((((((.......)))).)))))..)))......(((((....))))))).)..)))))))).)))))..))))). ( -53.50, z-score = -2.35, R) >droSim1.chrX 8140266 120 - 17042790 UUUUUGAGCACAUACCGCAAGGAUGGCGUCCUGCGCGGCUUGUACGCCGGUUCCGUUCCGGCCGUAUUCGCCAAUGUGGCCGAGAACUCGGUGCUGUUCGCCGCCUACGGUGGAUGCCAG ....((.(((..(((((..(((.(((((..(.((..(((..(((((((((.......)))).)))))..)))......(((((....))))))).)..)))))))).)))))..))))). ( -53.50, z-score = -2.35, R) >droPer1.super_21 799543 120 - 1645244 UUCGUGAGCACGUAUCGAAAGGAUGGUAUAUUCCGGGGCCUCUAUGCGGGCUCCCUGCCCGCCGUGGUGGCCAAUGUGGCCGAGAAUUCGGUUCUCUUUGCCGCCUACGGAGGAUGCCAG ..(((....)))..((....)).((((((.(((((.((((.((((((((((.....))))).))))).))))...(((((.((((((...))))))...)))))...))))).)))))). ( -55.40, z-score = -3.01, R) >dp4.chrXL_group1e 5861385 120 - 12523060 UUCGUGAGCACGUAUCGAAAGGAUGGUAUAUUCCGGGGCCUCUAUGCGGGCUCCCUGCCCGCCGUGGUGGCCAAUGUGGCCGAGAAUUCGGUGCUCUUUGCCGCCUACGGAGGAUGCCAG ..(((....)))..((....)).((((((.(((((.((((.((((((((((.....))))).))))).))))...(((((.((((.........)))).)))))...))))).)))))). ( -53.90, z-score = -2.30, R) >anoGam1.chrX 7854839 119 - 22145176 ACGCUGCAAACGUUCCGCCGGGACGGGAUCGUGCGCGGCCUGUACGCCGGCACGCUGCCCGCGGUCGUCGCGAACGUCGCGGAAAACUCAGUAUUAUUCGCCGCGUACGG-GCGUGCCAG (((((((....(((((...)))))(((..(((((.((((......)))))))))...))))))).)))((((..((((((((..((..........))..))))).))).-.)))).... ( -51.90, z-score = 0.63, R) >apiMel3.GroupUn 347024035 120 + 399230636 UUUUUACAAACAUUAAAGACAGAUGGAAUAAUGAAUGGUUUAUAUGCAGGAACUAUACCUGCAUUAGUUGCUAAUGUUGCUGAAAAUUCAGUUUUGUUUGCUGCAUAUGGAGGAUGUCAA ..........(((...((.((((..((((..(((((........((((((.......))))))(((((.((....)).)))))..)))))))))..))))))....)))........... ( -26.40, z-score = -0.83, R) >triCas2.ChLG4 1212561 120 - 13894384 UUCAUGAAAACGUUACGCGCAGAUGGAAUUUAUCGCGGUUUGUAUGCCGGUACAGUCCCGGCUUUGGCGGCAAAUAUUACCGAAAAUUCCGUUUUGUUUCUUUGUUACGGCUUUUGCCAG ........((((....(((.(((((((((((....((((.(((.(((((.((.(((....))).)).))))).)))..)))).)))))))))))))).....))))..(((....))).. ( -37.30, z-score = -1.94, R) >consensus UUUGUGAGCACAUAUCGCAAGGAUGGCGUCAUGCGCGGCUUGUACGCCGGCUCCGUGCCGGCCGUAUUCGCCAAUGUGGCCGAAAAUUCGGUGCUGUUUGCCGCCUACGGUGGAUGCCAG ......(((((..................................(((((.......))))).......(((.....)))..........))))).............((......)).. (-16.45 = -16.09 + -0.36)

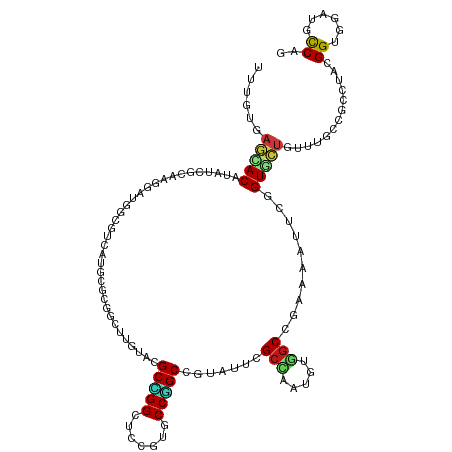

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:31:44 2011