
| Sequence ID | dm3.chrX |
|---|---|
| Location | 10,274,410 – 10,274,588 |
| Length | 178 |
| Max. P | 0.986256 |

| Location | 10,274,410 – 10,274,588 |
|---|---|
| Length | 178 |
| Sequences | 12 |
| Columns | 188 |
| Reading direction | forward |
| Mean pairwise identity | 70.42 |
| Shannon entropy | 0.63352 |
| G+C content | 0.64490 |
| Mean single sequence MFE | -89.43 |
| Consensus MFE | -20.13 |
| Energy contribution | -20.07 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.57 |
| Mean z-score | -2.77 |
| Structure conservation index | 0.23 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.906432 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
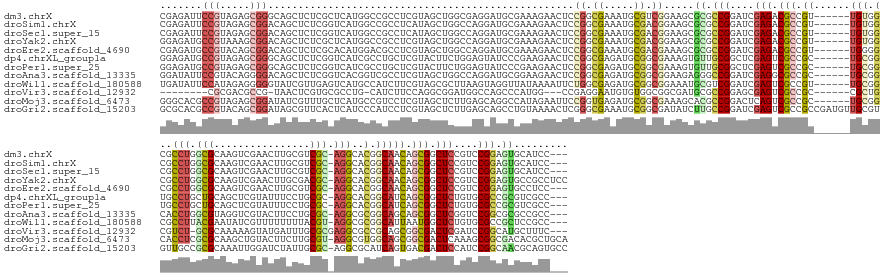
>dm3.chrX 10274410 178 + 22422827 CGAGAUUCCGUAGAGCGGGCAGCUCUCGCUCAUGGCCGCCUCGUAGCUGGCGAGGAUGCGAAAGAACUCCGGCGAAAUGCGUCGGAAGCGCGCCGGAUCGAGACGCCGU------UGUGGCGCCUGGCGCAAGUCGAACUUGCGUCGC-AGGCACGGCAACAGCGGCUCCGUCCGGAGUGCAUCC--- .......((((.(((((((.....))))))))))).((((........)))).((((((........(((((((.....)))))))...((.((((((.(((.(((.((------(((.(.((((((((((((.....)))))))..)-)))).).))))).))).))).)))))).))))))))--- ( -91.50, z-score = -2.48, R) >droSim1.chrX 7998265 178 + 17042790 CGAGAUUCCGUAGAGCGGACAGCUCUCGGUCAUGGCCGCCUCAUAGCUGGCCAGGAUGCGAAAGAACUCCGGCGAAAUGCGACGGAAGCGCGCCGGAUCGAGACGCCGU------UGUGGCGCCUGGCGCAAGUCGAACUUGCGUCGC-AGGCACGGCAACAGCGGCUCCGUCCGGAGUGCAUCC--- ...((..(((.(((((.....)))))))))).(((((((......)).)))))((((((......((((((((.....(((.(....)))))))((((.(((.(((.((------(((.(.((((((((((((.....)))))))..)-)))).).))))).))).))).)))))))))))))))--- ( -90.80, z-score = -3.27, R) >droSec1.super_15 971563 178 + 1954846 CGAGAUUCCGUAGAGCGGACAGCUCUCGGUCAUGGCCGCCUCAUAGCUGGCCAGGAUGCGAAAGAACUCCGGCGAAAUGCGACGGAAGCGCGCCGGAUCGAGACGCCGU------UGUGGCGCCUGGCGCAAGUCGAACUUGCGUCGC-AGGCACGGCAACAGCGGCUCCGUCCGGAGUGCAUCC--- ...((..(((.(((((.....)))))))))).(((((((......)).)))))((((((......((((((((.....(((.(....)))))))((((.(((.(((.((------(((.(.((((((((((((.....)))))))..)-)))).).))))).))).))).)))))))))))))))--- ( -90.80, z-score = -3.27, R) >droYak2.chrX 18848609 181 + 21770863 GGAGAUGCCGUAAAGCGGACAGCUCUCGCUCAUGGCCGCCUCGUAGCUGGCCAGGAUGCGAAAGAACUCCGGCGAAAUGCGACGGAAGCGCGCCGGAUCGAGACGCCGU------UGUGGCGCCUGGCGCAAGUCGAACUUGCGACGC-AGGCACGGCAACAGCGGCUCCGUCCGGAGUGCCGCCUCC ((((.(((((.....))).)).((.((((((.(((((((......)).))))).)).)))).))..))))((((....(((.(....)))).((((((.(((.(((.((------(((.(.((((((((((((.....)))))).).)-)))).).))))).))).))).)))))).....))))... ( -93.20, z-score = -2.86, R) >droEre2.scaffold_4690 13886977 178 - 18748788 CGAGAUGCCGUACAGCGGACAGCUCUCGCACAUGGACGCCUCGUAGCUGGCCAGGAUGCGAAAGAACUCCGGCGAAAUGCGACGAAAGCGCGCCGGAUCGAGACGCCGU------UGGGGCGCCUGGCGCAAGUCGAACUUGCGUCGC-AGGCACGGCAACAGCGGCUCCGUCCGGAGUGCCUCC--- .(((((((((.....))).))..))))((((.((((((.((((...(((((..(((..(....)...)))........(((.(....)))))))))..))))..(((((------((..((((((((((((((.....)))))))..)-))))...))..)))))))..))))))..))))....--- ( -85.30, z-score = -1.87, R) >dp4.chrXL_group1a 2266292 178 - 9151740 GGAGAUGCCGUAGAGCGGGCAGCUCUCGGUCAUCGCCUGCUCGUACUUCUGGAGUAUCCCGAAGAACUCCGGCGAGAUGCGGCGAAAGUGUUGCGGCUCGAGUCGCCGC------UGCGGUGCCUGCUGCAGCUCGUAUUUCCUGCGC-AGGCACGGCAUCAGCGGCUCUGUGCGCCGCGUCGCC--- ((.(((((((.(((((.....)))))))).)))).))(((.((((((((((((((.((.....))))))))).))).)))))))...(((.((((((.((((((((.(.------(((.((((((((.((((..........))))))-)))))).))).).)))))))...).)))))).))).--- ( -92.40, z-score = -2.51, R) >droPer1.super_25 405054 178 + 1448063 GGAGAUGCCGUAGAGCGGGCAGCUCUCGGUCAUCGCCUGCUCGUACUUCUGGAGUAUCCCGAAGAACUCCGGCGAGAUGCGGCGAAAGUGUUGCGGCUCGAGUCGCCGC------UGCGGUGCCUGCUGCAGCUCGUAUUUCCUGCGC-AGGCACGGCAUCAGCGGCUCUGUGCGCCGCGUCGCC--- ((.(((((((.(((((.....)))))))).)))).))(((.((((((((((((((.((.....))))))))).))).)))))))...(((.((((((.((((((((.(.------(((.((((((((.((((..........))))))-)))))).))).).)))))))...).)))))).))).--- ( -92.40, z-score = -2.51, R) >droAna3.scaffold_13335 878423 178 - 3335858 GGAUAUUCCGUACAGGGGACAGCUCUCGGUCACGGUCGCCUCGUAGCUGGCCAGGAUGCGGAAGAACUCCGGCGAGAUGCGGCGGAAGAGGGCCGGAUCGAGGCGCCGC------UGCGGCACCUGGCGUAGGUCGUACUUCCUGCGC-AGGCGCGGCAGCAGCGGCUCGGUCCGGCGCGCCGCC--- ((.....((((((..((((....)))).)).))))...))..(((.((.(((.(((..(....)...)))))).)).)))(((((....(.(((((((((((.(((.((------(((.((.((((.((((((........)))))))-))).)).))))).))).))))))))))).).)))))--- ( -102.50, z-score = -3.30, R) >droWil1.scaffold_180588 1110738 178 + 1294757 UGAUAUUCCAUAGAGGGGGUAUCGUUGAGUCAUGCCAUCUUCGUAGCGCUUAAGUAGGUUAUAAAAUUCUGGCGAGAUGCGGCGGAAAUGCGUCGGAUCGAGUCGCCGU------UGCGGCGCCUUACGAAUAUCGUUUUUUUUACGU-AGGCGCGGCAUUAAUGGCUCUGUGCGCCGCUCCGCC--- .((((((((......))))))))...(.(((.(((((..((..((((.((.....))))))..))....))))).))).)((((((...(((.((.((.((((((....------(((.((((((.(((................)))-)))))).)))....)))))).)).)).)))))))))--- ( -65.59, z-score = -0.88, R) >droVir3.scaffold_12932 89249 165 + 2102469 --------CGCGACGCCG-UAACUCGUGCGCCUG-CAUCUUCCAGGCGGAUGGCCAGCCCAUGG---CCGAGGAAUGUGUGGCGGCGAUGCGCCGGAGCGAGUCGCCGC------CGCUGCGUCU-GCGCAAAAAGUAUGAUUUGCGCGAGGCGCCGCAGCGGCGACUCGAUCCGGCAUGCUUUC--- --------.(((.(((((-(.((.(((.((((((-.......))))))..((((((.....)))---)))....))).)).)))))).)))((((((.(((((((((((------.((.((((((-(((((((........))))))).)))))).)).))))))))))).))))))........--- ( -101.10, z-score = -5.72, R) >droMoj3.scaffold_6473 2210096 181 + 16943266 GGGCACGCCGUAGAGCGGAUAUCGUUUGCUCAUGCCGUCCUCGUAGCUCUUGAGCAGGCCAUAGAAUUCCGGUGAGAUGCGGCGAAAGCACGCCGGACUCAGUCGCCGC------UGCGGCACCUCGCGCAAGCUGUACUUCUUGCGU-AGGCGUGGCAGCGGCGACUCAAAGCGGCGACACGCUGCA .(((.(((((((.(.(((((((.(((((((((.((((....))..))...))))))))).)))....)))).)....)))))))......(((((.....(((((((((------(((.((.(((.(((((((........)))))))-))).)).)))))))))))).....)))))....)))... ( -88.80, z-score = -2.42, R) >droGri2.scaffold_15203 4540816 187 - 11997470 GCGCACGCCGUACAGCGGAUAGCGUUCACUCAUCCCAUCCUCGUAGCUCUUGAGCAGCCUGUAAAACUCGGGCGAAAUGCGGCGAUAUCUUGCCGGAUCGAGUCGCCGCCGAUGUUGCGUGUUGCCGCGCAAAUUGGAUCUAUUGCGC-AGGCGCAUCAGUGACGACUCCAUCCGGCAACGCAGUGCC ((((.(((((((..(.((((((......)).)))))....((...(((....))).(((((.......)))))))..))))))).....(((((((((.((((((.(((.(((((........(((((((((..........))))))-.)))))))).))).)))))).)))))))))....)))). ( -78.80, z-score = -2.20, R) >consensus GGAGAUGCCGUAGAGCGGACAGCUCUCGCUCAUGGCCGCCUCGUAGCUGGCGAGGAUGCGAAAGAACUCCGGCGAAAUGCGGCGGAAGCGCGCCGGAUCGAGUCGCCGC______UGCGGCGCCUGGCGCAAGUCGUACUUGCUGCGC_AGGCACGGCAACAGCGGCUCCGUCCGGCGUGCCGCC___ .......(((.....)))...................................................((.((.....)).)).....((.(((....(((.(((.((......(((.(..(((.(((................))).)))..).))))).))).)))....))).))......... (-20.13 = -20.07 + -0.06)
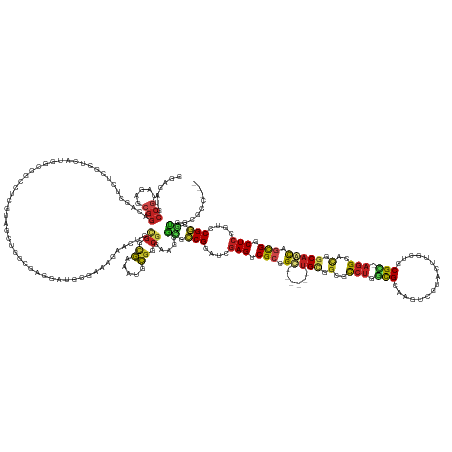
| Location | 10,274,410 – 10,274,588 |
|---|---|
| Length | 178 |
| Sequences | 12 |
| Columns | 188 |
| Reading direction | reverse |
| Mean pairwise identity | 70.42 |
| Shannon entropy | 0.63352 |
| G+C content | 0.64490 |
| Mean single sequence MFE | -85.07 |
| Consensus MFE | -23.58 |
| Energy contribution | -24.41 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.45 |
| Mean z-score | -3.01 |
| Structure conservation index | 0.28 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.23 |
| SVM RNA-class probability | 0.986256 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 10274410 178 - 22422827 ---GGAUGCACUCCGGACGGAGCCGCUGUUGCCGUGCCU-GCGACGCAAGUUCGACUUGCGCCAGGCGCCACA------ACGGCGUCUCGAUCCGGCGCGCUUCCGACGCAUUUCGCCGGAGUUCUUUCGCAUCCUCGCCAGCUACGAGGCGGCCAUGAGCGAGAGCUGCCCGCUCUACGGAAUCUCG ---(((.((.(.(((((..(((.((((((((..((((((-(.(.((((((.....))))))))))))))..))------)))))).)))..))))).).)).)))((.(.((((((.(((.((.(((((((.((...(((.(((....))))))...)))))))))..))))).....))))))))). ( -85.10, z-score = -2.56, R) >droSim1.chrX 7998265 178 - 17042790 ---GGAUGCACUCCGGACGGAGCCGCUGUUGCCGUGCCU-GCGACGCAAGUUCGACUUGCGCCAGGCGCCACA------ACGGCGUCUCGAUCCGGCGCGCUUCCGUCGCAUUUCGCCGGAGUUCUUUCGCAUCCUGGCCAGCUAUGAGGCGGCCAUGACCGAGAGCUGUCCGCUCUACGGAAUCUCG ---(((((((((((((.(((((.((((((((..((((((-(.(.((((((.....))))))))))))))..))------)))))).)))(((.(((((......))))).))).)))))))))......))))))(((((.(((....))))))))...((((((((.....))))).)))....... ( -91.50, z-score = -4.20, R) >droSec1.super_15 971563 178 - 1954846 ---GGAUGCACUCCGGACGGAGCCGCUGUUGCCGUGCCU-GCGACGCAAGUUCGACUUGCGCCAGGCGCCACA------ACGGCGUCUCGAUCCGGCGCGCUUCCGUCGCAUUUCGCCGGAGUUCUUUCGCAUCCUGGCCAGCUAUGAGGCGGCCAUGACCGAGAGCUGUCCGCUCUACGGAAUCUCG ---(((((((((((((.(((((.((((((((..((((((-(.(.((((((.....))))))))))))))..))------)))))).)))(((.(((((......))))).))).)))))))))......))))))(((((.(((....))))))))...((((((((.....))))).)))....... ( -91.50, z-score = -4.20, R) >droYak2.chrX 18848609 181 - 21770863 GGAGGCGGCACUCCGGACGGAGCCGCUGUUGCCGUGCCU-GCGUCGCAAGUUCGACUUGCGCCAGGCGCCACA------ACGGCGUCUCGAUCCGGCGCGCUUCCGUCGCAUUUCGCCGGAGUUCUUUCGCAUCCUGGCCAGCUACGAGGCGGCCAUGAGCGAGAGCUGUCCGCUUUACGGCAUCUCC ((((((((((((((((.(((((.((((((((..((((((-(.(.((((((.....))))))))))))))..))------)))))).)))(((.(((((......))))).))).)))))))))..((((((.((.(((((.(((....)))))))).)))))))))))))..(((....)))..)))) ( -94.30, z-score = -3.49, R) >droEre2.scaffold_4690 13886977 178 + 18748788 ---GGAGGCACUCCGGACGGAGCCGCUGUUGCCGUGCCU-GCGACGCAAGUUCGACUUGCGCCAGGCGCCCCA------ACGGCGUCUCGAUCCGGCGCGCUUUCGUCGCAUUUCGCCGGAGUUCUUUCGCAUCCUGGCCAGCUACGAGGCGUCCAUGUGCGAGAGCUGUCCGCUGUACGGCAUCUCG ---((((((.(.(((((..(((.((((((((..((((((-(.(.((((((.....))))))))))))))..))------)))))).)))..))))).).))))))((((.....((.((((...(((((((((..(((...(((....)))..))).)))))))))...)))).))..))))...... ( -87.00, z-score = -2.54, R) >dp4.chrXL_group1a 2266292 178 + 9151740 ---GGCGACGCGGCGCACAGAGCCGCUGAUGCCGUGCCU-GCGCAGGAAAUACGAGCUGCAGCAGGCACCGCA------GCGGCGACUCGAGCCGCAACACUUUCGCCGCAUCUCGCCGGAGUUCUUCGGGAUACUCCAGAAGUACGAGCAGGCGAUGACCGAGAGCUGCCCGCUCUACGGCAUCUCC ---(((((.(((((.....(((.(((((.(((.((((((-((((((..........)))).)))))))).)))------.))))).)))..))))).......))))).......((((((((((....))..)))))....((....)).)))((((.((((((((.....))))).)))))))... ( -86.30, z-score = -3.07, R) >droPer1.super_25 405054 178 - 1448063 ---GGCGACGCGGCGCACAGAGCCGCUGAUGCCGUGCCU-GCGCAGGAAAUACGAGCUGCAGCAGGCACCGCA------GCGGCGACUCGAGCCGCAACACUUUCGCCGCAUCUCGCCGGAGUUCUUCGGGAUACUCCAGAAGUACGAGCAGGCGAUGACCGAGAGCUGCCCGCUCUACGGCAUCUCC ---(((((.(((((.....(((.(((((.(((.((((((-((((((..........)))).)))))))).)))------.))))).)))..))))).......))))).......((((((((((....))..)))))....((....)).)))((((.((((((((.....))))).)))))))... ( -86.30, z-score = -3.07, R) >droAna3.scaffold_13335 878423 178 + 3335858 ---GGCGGCGCGCCGGACCGAGCCGCUGCUGCCGCGCCU-GCGCAGGAAGUACGACCUACGCCAGGUGCCGCA------GCGGCGCCUCGAUCCGGCCCUCUUCCGCCGCAUCUCGCCGGAGUUCUUCCGCAUCCUGGCCAGCUACGAGGCGACCGUGACCGAGAGCUGUCCCCUGUACGGAAUAUCC ---.((((((.((((((.((((.(((((((((.((((((-(((.(((........))).)).))))))).)))------)))))).)))).)))))).......))))))........(((....(((((.((...((.(((((.(..((((....)).))..)))))).))...)).)))))..))) ( -88.20, z-score = -2.59, R) >droWil1.scaffold_180588 1110738 178 - 1294757 ---GGCGGAGCGGCGCACAGAGCCAUUAAUGCCGCGCCU-ACGUAAAAAAAACGAUAUUCGUAAGGCGCCGCA------ACGGCGACUCGAUCCGACGCAUUUCCGCCGCAUCUCGCCAGAAUUUUAUAACCUACUUAAGCGCUACGAAGAUGGCAUGACUCAACGAUACCCCCUCUAUGGAAUAUCA ---((((((((((((..............(((.((((((-(((................))).)))))).)))------...(((..(((...)))))).....)))))).)).)))).......................((((......))))..........((((..((......))..)))). ( -49.29, z-score = -0.24, R) >droVir3.scaffold_12932 89249 165 - 2102469 ---GAAAGCAUGCCGGAUCGAGUCGCCGCUGCGGCGCCUCGCGCAAAUCAUACUUUUUGCGC-AGACGCAGCG------GCGGCGACUCGCUCCGGCGCAUCGCCGCCACACAUUCCUCGG---CCAUGGGCUGGCCAUCCGCCUGGAAGAUG-CAGGCGCACGAGUUA-CGGCGUCGCG-------- ---........((((((.((((((((((((((.(((.((.(((((((........)))))))-)).))).)))------))))))))))).))))))((..(((((..((.........((---(((.....)))))...((((((.......-)))))).....))..-)))))..)).-------- ( -95.50, z-score = -5.57, R) >droMoj3.scaffold_6473 2210096 181 - 16943266 UGCAGCGUGUCGCCGCUUUGAGUCGCCGCUGCCACGCCU-ACGCAAGAAGUACAGCUUGCGCGAGGUGCCGCA------GCGGCGACUGAGUCCGGCGUGCUUUCGCCGCAUCUCACCGGAAUUCUAUGGCCUGCUCAAGAGCUACGAGGACGGCAUGAGCAAACGAUAUCCGCUCUACGGCGUGCCC .(((..(((((((((..((.((((((((((((..(((((-.((((((........))))))..)))))..)))------))))))))).))..)))).(((((..((((..((((...((....)).((((((.....)).)))).)))).))))..)))))...))))).((((....))))))).. ( -82.50, z-score = -1.96, R) >droGri2.scaffold_15203 4540816 187 + 11997470 GGCACUGCGUUGCCGGAUGGAGUCGUCACUGAUGCGCCU-GCGCAAUAGAUCCAAUUUGCGCGGCAACACGCAACAUCGGCGGCGACUCGAUCCGGCAAGAUAUCGCCGCAUUUCGCCCGAGUUUUACAGGCUGCUCAAGAGCUACGAGGAUGGGAUGAGUGAACGCUAUCCGCUGUACGGCGUGCGC .((((.((((((((((((.((((((((.((((((.((..-((((((..........))))))(....)..))..)))))).)))))))).)))))))).....((((..(((..((((((((((((..((....))..)))))).)).)).))..))).)))))))).....(((....))))))).. ( -83.30, z-score = -2.61, R) >consensus ___GGCGGCACGCCGGACGGAGCCGCUGUUGCCGUGCCU_GCGCAGCAAGUACGACUUGCGCCAGGCGCCGCA______ACGGCGACUCGAUCCGGCGCGCUUCCGCCGCAUUUCGCCGGAGUUCUUUCGCAUCCUCGCCAGCUACGAGGAGGCCAUGACCGAGAGCUGUCCGCUCUACGGCAUCUCC ............(((....(((.((((((((..((((((.(((................))).))))))..))......)))))).)))....)))...(((((.((........)).)))))...............................................(((.....)))....... (-23.58 = -24.41 + 0.84)

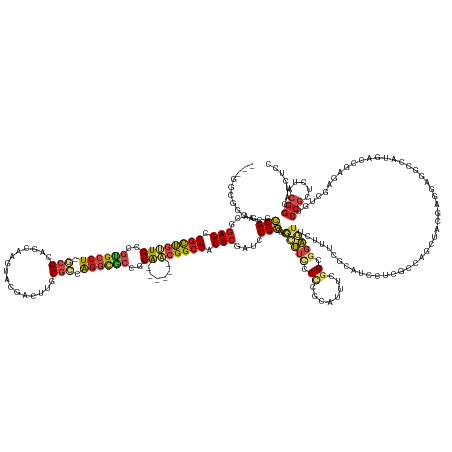
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:31:01 2011