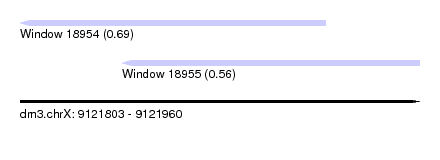
| Sequence ID | dm3.chrX |
|---|---|
| Location | 9,121,803 – 9,121,960 |
| Length | 157 |
| Max. P | 0.687988 |

| Location | 9,121,803 – 9,121,923 |
|---|---|
| Length | 120 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 72.42 |
| Shannon entropy | 0.60139 |
| G+C content | 0.55069 |
| Mean single sequence MFE | -42.47 |
| Consensus MFE | -14.14 |
| Energy contribution | -13.79 |
| Covariance contribution | -0.34 |
| Combinations/Pair | 1.84 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.33 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.687988 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 9121803 120 - 22422827 CCCAUUUCGCGUUCCAAGCCGGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGUACGAACUGCAGAUUGCGCAGAUACGUCAGCCACACGAGCGGCUCAUAGGUGCUCCAAAAGAGCAAC .((.....((((.....(((((((((((((((...)))))))).....((((((....))))))))))).))....)))).((((.(....).))))....))(((((.....))))).. ( -41.50, z-score = -2.08, R) >droSim1.chrX 7279648 120 - 17042790 CCCAUUUCGCGUUCCAAGCCAGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGCACGAACUGCAGAUUGCGCAGGUACGUCAGCCACACGAGCGGCUCAUAGGUGCUCCAAAAGAGCAAC .((.....((((.((..(((((((((((((((...)))))))).....(((((......)))))))))).)).)).)))).((((.(....).))))....))(((((.....))))).. ( -43.60, z-score = -2.48, R) >droSec1.super_68 9823 120 - 137979 CCCAUUUCGCGUUCCAAGCCGGUCAGCUGCUCUCUGAGCAGCUCCAAGUGCAGCACGAACUGCAGAUUGCGCAGGUACGUCAGCCACACGAGCGGCUCAUAGGUGCUCCAAAAGAGCAAC .((.....((((.((..(((((((((((((((...)))))))).....(((((......)))))))))).)).)).)))).((((.(....).))))....))(((((.....))))).. ( -42.80, z-score = -1.53, R) >droYak2.chrX 17718681 120 - 21770863 CCCAUUUCGCGUUCCAAAGCGUUCAGGUGCUCCCGAAGCAUCUCCAGGUGCAGCACAAACUGCAGAUUUCGCAGAUACGUCAGCCACACGAGCGGCUCGUAGGUGCUCCAAAAGAGCAAC .((....(((........)))....))(((((.....(((((....)))))(((((...((((.......)))).((((..((((.(....).)))))))).)))))......))))).. ( -38.20, z-score = -2.04, R) >droEre2.scaffold_4690 12754038 120 + 18748788 CCAAUUUCGCGUUCCAAGGCGGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGCACGAACUGCAGAUUUCGCAGGUACGUCAGCCACACGAGCGGCUCAUAGGUGCUCCAAAAGAGCAAC ((......((((.((...((((((((((((((...)))))))).....(((((......))))))))..))).)).)))).((((.(....).))))....))(((((.....))))).. ( -41.00, z-score = -1.43, R) >droAna3.scaffold_13047 985884 120 - 1816235 CGGAGCUCCUGUUCCAGCUGGGCCAGCUGCUGACGGAGUAGCUCCAUGUGAAGGAUAAACUGGAUGUUUCGGAGGUAGGUCAGGACCACCAGGGGCUCGUAGGUGGUCCAGAAGAGCAGA ........((((((...((((((((.((((.......(.((((((.(.(((((.((.......)).))))).)(((.(((....)))))).))))))))))).))))))))..)))))). ( -46.41, z-score = -0.84, R) >dp4.chrXL_group1e 11343171 120 + 12523060 GAAAGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAUGAGGAAAACGAGCGGCUCGAAUGUGCUCCAGAAGAGUAUC .........(((((((((((((..(((((((.....)))))))))((((((((......))))))))(((.(((....))).))).......)))))))))))(((((.....))))).. ( -46.40, z-score = -1.95, R) >droPer1.super_30 15265 120 - 958394 GAAAGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAUGAGGAAAACGAGCGGCUCGAAUGUGCUCCAGAAGAGUAUC .........(((((((((((((..(((((((.....)))))))))((((((((......))))))))(((.(((....))).))).......)))))))))))(((((.....))))).. ( -46.40, z-score = -1.95, R) >droWil1.scaffold_181096 4922158 120 + 12416693 UUCAACUCGUGCGCUAAUUGCUUCAGUUGUUGACGCAAUAGCUCCACAUGCAAGACGAAUUGUAUGUUGCGGAUAUGAGUCAGCCAAACAAAUGGUUGAAAUGUUGUCCAAAAGAGUACC ....((((..((((((.((((.((((...)))).))))))))...((((((((......)))))))).))(((((....(((((((......))))))).....)))))....))))... ( -39.50, z-score = -3.61, R) >droVir3.scaffold_12970 7541447 120 - 11907090 CCCAGCUCGCAUUCCAAUUGCACCAGCUGUUGCCGCAACAGCUCCAGGUGCAGCAAAAAUUGCGUGUUGCGCAAAUGCACCAGAAACACAAACGGCUGAAAGGUGCUCCAAAAGAGCAGA ..(((((.(((......(((((((((((((((...))))))))...))))))).......)))((((((.((....)).)....)))))....))))).....(((((.....))))).. ( -40.32, z-score = -1.57, R) >droMoj3.scaffold_6482 554292 120 + 2735782 AUCAGCUCGCACUCCAACUGCAGGAGUUUCUGGCGCAACAGAUCCAUGUGUAGCACGAACUGGGUGUUGCGCAGGUGCACCACCAAGAUGAACGGCUGAAAGGUGAUCCAGAAGAGAACA .....(((..(((((.......)))))(((((((((..(((..((.((((((((((.......))))))))))((((...)))).........)))))....)))..))))))))).... ( -38.20, z-score = -0.27, R) >droGri2.scaffold_15203 4927217 120 + 11997470 GCCAGCUCCGAAUCCAACUGGCGGAUUUGCUGGCGGACCACCUCCAGGUGCAGCACAAACUGUGUGUUUCGCAAGUGCACCACAAACACAAACGGCUGAAAAGUGGUCCAAAACAGUAGA ((((((...((((((.......))))))))))))(((((((.....((((((((((.....))))....(....))))))).((..(......)..))....)))))))........... ( -45.30, z-score = -2.71, R) >consensus CCCAGCUCGCGUUCCAACUGGGUCAGCUGCUGCCGGAGCAGCUCCAAGUGCAGCACAAACUGCAGGUUGCGCAGGUACGUCAGCAACACGAGCGGCUCAAAGGUGCUCCAAAAGAGCAAC .......(((..............(((((((.....))))))).....(((((......)))))...........................)))((((...............))))... (-14.14 = -13.79 + -0.34)

| Location | 9,121,843 – 9,121,960 |
|---|---|
| Length | 117 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 71.41 |
| Shannon entropy | 0.62506 |
| G+C content | 0.56595 |
| Mean single sequence MFE | -42.89 |
| Consensus MFE | -14.19 |
| Energy contribution | -14.03 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.70 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.33 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.556863 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 9121843 117 - 22422827 CGUUUCGCU---AGCAAAUCGCGAGUACUCCGCCAGCAGACCCAUUUCGCGUUCCAAGCCGGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGUACGAACUGCAGAUUGCGCAGAUACGU (((.((...---.......((((((...((.(....).)).....))))))......(((((((((((((((...)))))))).....((((((....))))))))))).)).)).))). ( -37.00, z-score = -1.27, R) >droSim1.chrX 7279688 117 - 17042790 CGUUUCGCU---GGCGAAUCGCGAGUACUCCGCCAGCAGACCCAUUUCGCGUUCCAAGCCAGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGCACGAACUGCAGAUUGCGCAGGUACGU .((((.(((---((((...(....).....))))))))))).......((((.((..(((((((((((((((...)))))))).....(((((......)))))))))).)).)).)))) ( -47.80, z-score = -3.26, R) >droSec1.super_68 9863 117 - 137979 CGUUUCGCU---GGCGAAUCGCGAGUACUCCGCCAGCAGACCCAUUUCGCGUUCCAAGCCGGUCAGCUGCUCUCUGAGCAGCUCCAAGUGCAGCACGAACUGCAGAUUGCGCAGGUACGU .((((.(((---((((...(....).....))))))))))).......((((.((..(((((((((((((((...)))))))).....(((((......)))))))))).)).)).)))) ( -47.00, z-score = -2.18, R) >droYak2.chrX 17718721 117 - 21770863 CGUUUCGCU---GGCGAAUCGCGAGUACUCCGCCAGGAGUCCCAUUUCGCGUUCCAAAGCGUUCAGGUGCUCCCGAAGCAUCUCCAGGUGCAGCACAAACUGCAGAUUUCGCAGAUACGU .(...((((---...(((.((((((.(((((....))))).....)))))))))...))))..)..(((((......(((((....))))))))))...((((.......))))...... ( -38.10, z-score = -1.53, R) >droEre2.scaffold_4690 12754078 117 + 18748788 CGUCCCACU---GGCGAAUCGCGAGUACUCCGCCAGGAGACCAAUUUCGCGUUCCAAGGCGGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGCACGAACUGCAGAUUUCGCAGGUACGU .(((.(.((---((((...(....).....))))))).))).......((((.((...((((((((((((((...)))))))).....(((((......))))))))..))).)).)))) ( -43.50, z-score = -1.79, R) >droAna3.scaffold_13047 985924 120 - 1816235 CGUUCCGCUGCAGGCGAAUCUCGAAUACUCGGCCAGGAGACGGAGCUCCUGUUCCAGCUGGGCCAGCUGCUGACGGAGUAGCUCCAUGUGAAGGAUAAACUGGAUGUUUCGGAGGUAGGU ..((((((((.((.((.....))....))))))...(((((((((((((((((.((((((...))))))..))))).).))))))..((..((......))..)))))))))))...... ( -39.30, z-score = 0.84, R) >dp4.chrXL_group1e 11343211 117 + 12523060 CGUUAGGCU---GGCAAAUCGCGAGUACUCUGCCAGUAGGGAAAGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAU .(((.((((---(((.....))((((.((((((((((((((..(((((......)))))...))..))))))))))))..))))......)))).).)))(((((((.....))))))). ( -48.70, z-score = -1.83, R) >droPer1.super_30 15305 117 - 958394 CGUUAGGCU---GGCAAAUCGCGAGUACUCUGCCAGUAGGGAAAGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAU .(((.((((---(((.....))((((.((((((((((((((..(((((......)))))...))..))))))))))))..))))......)))).).)))(((((((.....))))))). ( -48.70, z-score = -1.83, R) >droWil1.scaffold_181096 4922198 114 + 12416693 ----CCGAUU--GGCAAAUUUCGAAUAUUCCGCCAGUAGAUUCAACUCGUGCGCUAAUUGCUUCAGUUGUUGACGCAAUAGCUCCACAUGCAAGACGAAUUGUAUGUUGCGGAUAUGAGU ----...(((--(((................)))))).......(((((((.((((.((((.((((...)))).))))))))(((((((((((......))))))))...)))))))))) ( -31.49, z-score = -1.20, R) >droVir3.scaffold_12970 7541487 117 - 11907090 UGCAACGCU---GGCAAAGCUCGAGUACUCAACGAGCAGCCCCAGCUCGCAUUCCAAUUGCACCAGCUGUUGCCGCAACAGCUCCAGGUGCAGCAAAAAUUGCGUGUUGCGCAAAUGCAC .((((((((---((....(((((.........)))))....))))).((((......(((((((((((((((...))))))))...))))))).......)))).)))))((....)).. ( -47.92, z-score = -3.17, R) >droMoj3.scaffold_6482 554332 117 + 2735782 UGAGCUGUU---AGCAAAGCUCGAGUAUUCGACGAGUAGUAUCAGCUCGCACUCCAACUGCAGGAGUUUCUGGCGCAACAGAUCCAUGUGUAGCACGAACUGGGUGUUGCGCAGGUGCAC .((((((..---.((...(((((.........))))).))..))))))(((((.........(((...((((......))))))).((((((((((.......))))))))))))))).. ( -42.00, z-score = -1.15, R) >droGri2.scaffold_15203 4927257 117 + 11997470 CGCACCGCU---GGCAAAGCCUGAGUACUCGACCAACAGCGCCAGCUCCGAAUCCAACUGGCGGAUUUGCUGGCGGACCACCUCCAGGUGCAGCACAAACUGUGUGUUUCGCAAGUGCAC .(((((((.---.(((((.(((((....))).........(((((............))))))).)))))..))(((.....))).))))).((((....((((.....)))).)))).. ( -43.20, z-score = -1.15, R) >consensus CGUUCCGCU___GGCAAAUCGCGAGUACUCCGCCAGCAGACCCAGCUCGCGUUCCAACUGGGUCAGCUGCUGCCGGAGCAGCUCCAAGUGCAGCACAAACUGCAGGUUGCGCAGGUACGU ......((.....)).........(((((......((((.........................(((((((.....))))))).....(((((......)))))..))))...))))).. (-14.19 = -14.03 + -0.16)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:27:36 2011