
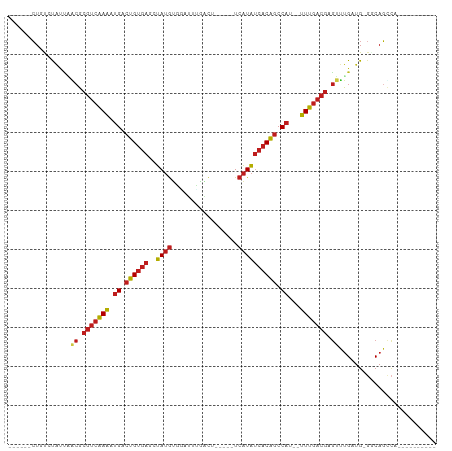
| Sequence ID | dm3.chrX |
|---|---|
| Location | 8,985,257 – 8,985,353 |
| Length | 96 |
| Max. P | 0.999977 |

| Location | 8,985,257 – 8,985,347 |
|---|---|
| Length | 90 |
| Sequences | 13 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 71.72 |
| Shannon entropy | 0.57255 |
| G+C content | 0.45101 |
| Mean single sequence MFE | -35.68 |
| Consensus MFE | -14.93 |
| Energy contribution | -15.34 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.24 |
| Mean z-score | -4.71 |
| Structure conservation index | 0.42 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.94 |
| SVM RNA-class probability | 0.999926 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
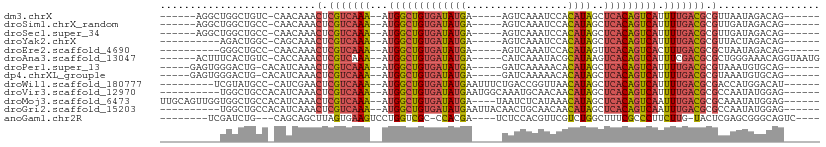
>dm3.chrX 8985257 90 + 22422827 ------CUGUCUAUUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUG-GACAGCCAGCCU------ ------(((((((..(((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).)))...))-))))).......------ ( -36.30, z-score = -5.62, R) >droSim1.chrX_random 2632475 90 + 5698898 ------CUGUCUAUCAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUG-GGCAGCCAGCCU------ ------(((((((.((((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).).))).))-))))).......------ ( -35.30, z-score = -4.43, R) >droSec1.super_34 345612 90 + 457129 ------CUGUCUAUCAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUG-GGCAGCCAGCCU------ ------(((((((.((((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).).))).))-))))).......------ ( -35.30, z-score = -4.43, R) >droYak2.chrX 9440222 86 - 21770863 ------CUGUCUAGUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGCUG-GCCAGUCU---------- ------(((.((((((((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).).))))))-).)))...---------- ( -35.60, z-score = -5.24, R) >droEre2.scaffold_4690 12616408 86 - 18748788 ------CUGUCUAUUAGCGCGUCAAAGUGACUGUGAACUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUG-GGCAGCCC---------- ------(((((((..(((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).)))...))-)))))...---------- ( -35.70, z-score = -5.26, R) >droAna3.scaffold_13047 797475 96 + 1816235 CAUUACCUGUUUCCCAGCGCGUCGAAAUGACUGUGACUUAUGCGUAUUUGAUG-----UCAUAUCACAGCCAU--UUUGACGAGUUUGGUG-GACAGUGAAAGU------ ..((((.((((..(((((.((((((((((.((((((..(((((((.....)))-----.)))))))))).)))--))))))).).))))..-))))))))....------ ( -38.00, z-score = -5.30, R) >droPer1.super_13 1553262 91 + 2293547 ------CUGCACAUUUACGCGUCAAAAUGACUGUGAGCUAUGUGUUUUUGAUC-----UCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUG-CAGUCCCACUC----- ------(((((((((.((.((((((((((.((((((..((((.(........)-----.)))))))))).)))--))))))).))..))))))-)))........----- ( -39.00, z-score = -6.70, R) >dp4.chrXL_group1e 5522185 91 - 12523060 ------CUGCACAUUUACGCGUCAAAAUGACUGUGAGCUAUGUGUUUUUGAUC-----UCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUG-CAGUCCCACUC----- ------(((((((((.((.((((((((((.((((((..((((.(........)-----.)))))))))).)))--))))))).))..))))))-)))........----- ( -39.00, z-score = -6.70, R) >droWil1.scaffold_180777 2053079 92 + 4753960 ------AUGUCCAUGGUCGCGUCAAAAUGACUGUGAGCUAUGUUAACCGGUCAGAAAUUCAUAUCACAGCCAU--UUUGACGAGUUCGAUG-GGCAUACGA--------- ------((((((((.(...((((((((((.((((((..((((.....(.....).....)))))))))).)))--)))))))....).)))-)))))....--------- ( -35.00, z-score = -4.07, R) >droVir3.scaffold_12970 9304330 92 + 11907090 ------CUCCAUAUUGGCGCGUCAAAAUGACUGUGAGCUAUGUUGUUGCAUUUGCCAUUCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGGCAGCCA---------- ------((((((((..((.((((((((((.((((((..((((..((.((....)).)).)))))))))).)))--))))))).).)..)))))).))...---------- ( -35.80, z-score = -4.14, R) >droMoj3.scaffold_6473 3600626 98 - 16943266 ------CUCCAUAUUUGCGCGUCAAAUUGACUGUGAGCUAUGUUUAUGAGAUUA----UCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGGCAGCCACCAACUGCAA ------...((((((.((.(((((((.((.((((((........(((((.....----))))))))))).)).--))))))).))..))))))((((.......)))).. ( -31.90, z-score = -3.58, R) >droGri2.scaffold_15203 307012 92 + 11997470 ------CUCCAUAUUGGCGCGUCAAAUUGACUGUGAGCUAUGUUGUUGCAGUUGUAAUUCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGGCAGCCA---------- ------((((((((..((.(((((((.((.((((((..((((..(((((....))))).)))))))))).)).--))))))).).)..)))))).))...---------- ( -34.70, z-score = -3.91, R) >anoGam1.chr2R 35945408 89 - 62725911 ----GACUGCCCGCUCGAGUA-CAAGAAGGGCGAAAGCCAGACGAACGUGGAGA----UCGUGG-GCGACCAGGACUUCACUAAGCUGCUG---CAGAUCGA-------- ----((((((..((...(((.-.......(((....)))........((((((.----((.(((-....))).))))))))...))))).)---))).))..-------- ( -32.30, z-score = -1.86, R) >consensus ______CUGUCUAUUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU_____UCAUAUCACAGCCAU__UUUGACGAGUUUGAUG_GGCAGCCA__________ ................((.(((((((.((.((((((..((((.................)))))))))).))...))))))).))......................... (-14.93 = -15.34 + 0.40)


| Location | 8,985,257 – 8,985,347 |
|---|---|
| Length | 90 |
| Sequences | 13 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 71.72 |
| Shannon entropy | 0.57255 |
| G+C content | 0.45101 |
| Mean single sequence MFE | -31.59 |
| Consensus MFE | -17.97 |
| Energy contribution | -18.18 |
| Covariance contribution | 0.21 |
| Combinations/Pair | 1.29 |
| Mean z-score | -4.03 |
| Structure conservation index | 0.57 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.97 |
| SVM RNA-class probability | 0.999930 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 8985257 90 - 22422827 ------AGGCUGGCUGUC-CAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUAAUAGACAG------ ------.......(((((-.....(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).)))....)))))------ ( -33.90, z-score = -5.35, R) >droSim1.chrX_random 2632475 90 - 5698898 ------AGGCUGGCUGCC-CAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUGAUAGACAG------ ------.(((.....)))-.....(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).))).........------ ( -33.10, z-score = -4.36, R) >droSec1.super_34 345612 90 - 457129 ------AGGCUGGCUGCC-CAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUGAUAGACAG------ ------.(((.....)))-.....(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).))).........------ ( -33.10, z-score = -4.36, R) >droYak2.chrX 9440222 86 + 21770863 ----------AGACUGGC-CAGCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUACUAGACAG------ ----------...(((.(-.((..(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).))).)).).)))------ ( -30.90, z-score = -4.67, R) >droEre2.scaffold_4690 12616408 86 + 18748788 ----------GGGCUGCC-CAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGUUCACAGUCACUUUGACGCGCUAAUAGACAG------ ----------(((...))-)......(.(((((((--.(((((((((((((.-----.(......)..))))..))))))))).))))))).)...........------ ( -25.20, z-score = -3.02, R) >droAna3.scaffold_13047 797475 96 - 1816235 ------ACUUUCACUGUC-CACCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----CAUCAAAUACGCAUAAGUCACAGUCAUUUCGACGCGCUGGGAAACAGGUAAUG ------.......((((.-..(((..(.((((.((--((((((((((((((.-----(.........))))..))))))))))))).)))).).)))...))))...... ( -30.20, z-score = -3.98, R) >droPer1.super_13 1553262 91 - 2293547 -----GAGUGGGACUG-CACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----GAUCAAAAACACAUAGCUCACAGUCAUUUUGACGCGUAAAUGUGCAG------ -----........(((-(((((...((.(((((((--((((((((((((((.-----(........).))))..))))))))))))))))).))..))))))))------ ( -39.80, z-score = -6.75, R) >dp4.chrXL_group1e 5522185 91 + 12523060 -----GAGUGGGACUG-CACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----GAUCAAAAACACAUAGCUCACAGUCAUUUUGACGCGUAAAUGUGCAG------ -----........(((-(((((...((.(((((((--((((((((((((((.-----(........).))))..))))))))))))))))).))..))))))))------ ( -39.80, z-score = -6.75, R) >droWil1.scaffold_180777 2053079 92 - 4753960 ---------UCGUAUGCC-CAUCGAACUCGUCAAA--AUGGCUGUGAUAUGAAUUUCUGACCGGUUAACAUAGCUCACAGUCAUUUUGACGCGACCAUGGACAU------ ---------....(((.(-(((.(..(.(((((((--((((((((((((((.....((....))....))))..))))))))))))))))).)..))))).)))------ ( -32.20, z-score = -4.10, R) >droVir3.scaffold_12970 9304330 92 - 11907090 ----------UGGCUGCCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGAAUGGCAAAUGCAACAACAUAGCUCACAGUCAUUUUGACGCGCCAAUAUGGAG------ ----------((((((......))....(((((((--((((((((((((((....((....)).....))))..))))))))))))))))).))))........------ ( -32.50, z-score = -3.98, R) >droMoj3.scaffold_6473 3600626 98 + 16943266 UUGCAGUUGGUGGCUGCCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGA----UAAUCUCAUAAACAUAGCUCACAGUCAAUUUGACGCGCAAAUAUGGAG------ ((((((((.((((...))))....))))(((((((--.((((((((((((((----.....)))))........))))))))).))))))).))))........------ ( -31.90, z-score = -3.56, R) >droGri2.scaffold_15203 307012 92 - 11997470 ----------UGGCUGCCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGAAUUACAACUGCAACAACAUAGCUCACAGUCAAUUUGACGCGCCAAUAUGGAG------ ----------((((((......))....(((((((--.(((((((((((((.................))))..))))))))).))))))).))))........------ ( -25.03, z-score = -2.44, R) >anoGam1.chr2R 35945408 89 + 62725911 --------UCGAUCUG---CAGCAGCUUAGUGAAGUCCUGGUCGC-CCACGA----UCUCCACGUUCGUCUGGCUUUCGCCCUUCUUG-UACUCGAGCGGGCAGUC---- --------..((.(((---(....(((((((((((....(((.((-(.((((----.(.....).))))..)))....)))...))).-)))).))))..))))))---- ( -23.00, z-score = 0.92, R) >consensus __________UGGCUGCC_CAUCAAACUCGUCAAA__AUGGCUGUGAUAUGA_____AAUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGCUAAUAGACAG______ ..........................(.(((((((...(((((((((((((.................))))..))))))))).))))))).)................. (-17.97 = -18.18 + 0.21)
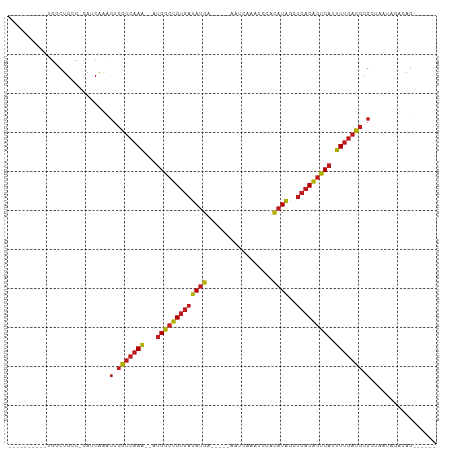
| Location | 8,985,257 – 8,985,353 |
|---|---|
| Length | 96 |
| Sequences | 13 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 69.95 |
| Shannon entropy | 0.66624 |
| G+C content | 0.44808 |
| Mean single sequence MFE | -35.52 |
| Consensus MFE | -19.23 |
| Energy contribution | -19.17 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.42 |
| Mean z-score | -4.34 |
| Structure conservation index | 0.54 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 5.54 |
| SVM RNA-class probability | 0.999977 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
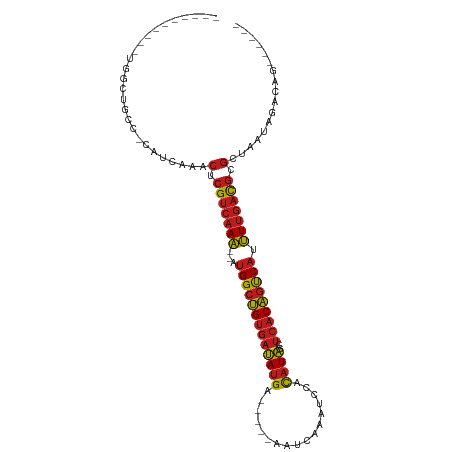
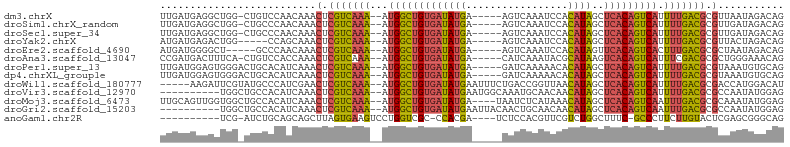
>dm3.chrX 8985257 96 + 22422827 CUGUCUAUUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUGGACAG-CCAGCCUCAUCAA (((((((..(((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).)))...)))))))-............. ( -36.30, z-score = -4.94, R) >droSim1.chrX_random 2632475 96 + 5698898 CUGUCUAUCAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUGGGCAG-CCAGCCUCAUCAA (((((((.((((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).).))).)))))))-............. ( -35.30, z-score = -3.94, R) >droSec1.super_34 345612 96 + 457129 CUGUCUAUCAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUGGGCAG-CCAGCCUCAUCAA (((((((.((((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).).))).)))))))-............. ( -35.30, z-score = -3.94, R) >droYak2.chrX 9440222 92 - 21770863 CUGUCUAGUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGCUGG-----CCAGUCUCAUCAU (((.((((((((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).).)))))))-----.)))......... ( -35.60, z-score = -4.67, R) >droEre2.scaffold_4690 12616408 92 - 18748788 CUGUCUAUUAGCGCGUCAAAGUGACUGUGAACUAUGUGGAUUUGACU-----UCAUAUCACAGCCAU--UUUGACGAGUUUGUUGGGC-----AGCCCCAUCAU (((((((..(((.((((((((((.((((((....((((((......)-----))))))))))).)))--))))))).)))...)))))-----))......... ( -35.70, z-score = -4.68, R) >droAna3.scaffold_13047 797481 96 + 1816235 CUGUUUCCCAGCGCGUCGAAAUGACUGUGACUUAUGCGUAUUUGAUG-----UCAUAUCACAGCCAU--UUUGACGAGUUUGGUGGACAG-UGAAAGUCAUCGG (((((..(((((.((((((((((.((((((..(((((((.....)))-----.)))))))))).)))--))))))).).))))..)))))-............. ( -37.30, z-score = -4.30, R) >droPer1.super_13 1553262 97 + 2293547 CUGCACAUUUACGCGUCAAAAUGACUGUGAGCUAUGUGUUUUUGAUC-----UCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGCAGUCCCACUCCAUCAA (((((((((.((.((((((((((.((((((..((((.(........)-----.)))))))))).)))--))))))).))..))))))))).............. ( -39.00, z-score = -6.47, R) >dp4.chrXL_group1e 5522185 97 - 12523060 CUGCACAUUUACGCGUCAAAAUGACUGUGAGCUAUGUGUUUUUGAUC-----UCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGCAGUCCCACUCCAUCAA (((((((((.((.((((((((((.((((((..((((.(........)-----.)))))))))).)))--))))))).))..))))))))).............. ( -39.00, z-score = -6.47, R) >droWil1.scaffold_180777 2053079 97 + 4753960 AUGUCCAUGGUCGCGUCAAAAUGACUGUGAGCUAUGUUAACCGGUCAGAAAUUCAUAUCACAGCCAU--UUUGACGAGUUCGAUGGGCAUACGAAUCUU----- ((((((((.(...((((((((((.((((((..((((.....(.....).....)))))))))).)))--)))))))....).)))))))).........----- ( -35.00, z-score = -4.00, R) >droVir3.scaffold_12970 9304330 92 + 11907090 CUCCAUAUUGGCGCGUCAAAAUGACUGUGAGCUAUGUUGUUGCAUUUGCCAUUCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGGCAGCCA---------- ((((((((..((.((((((((((.((((((..((((..((.((....)).)).)))))))))).)))--))))))).).)..)))))).))...---------- ( -35.80, z-score = -4.14, R) >droMoj3.scaffold_6473 3600626 98 - 16943266 CUCCAUAUUUGCGCGUCAAAUUGACUGUGAGCUAUGUUUAUGAGAUUA----UCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGGCAGCCACCAACUGCAA ...((((((.((.(((((((.((.((((((........(((((.....----))))))))))).)).--))))))).))..))))))((((.......)))).. ( -31.90, z-score = -3.58, R) >droGri2.scaffold_15203 307012 92 + 11997470 CUCCAUAUUGGCGCGUCAAAUUGACUGUGAGCUAUGUUGUUGCAGUUGUAAUUCAUAUCACAGCCAU--UUUGACGAGUUUGAUGUGGCAGCCA---------- ((((((((..((.(((((((.((.((((((..((((..(((((....))))).)))))))))).)).--))))))).).)..)))))).))...---------- ( -34.70, z-score = -3.91, R) >anoGam1.chr2R 35945410 87 - 62725911 CUGCCCGCUCGAGUACAAGAAGGGC-GAAAGCCAGACGAACGUGGAGA----UCGUGG-GCGACCAGGACUUCACUAAGCUGCUGCAGAU-CGA---------- ((((..((...(((........(((-....)))........((((((.----((.(((-....))).))))))))...))))).))))..-...---------- ( -30.90, z-score = -1.43, R) >consensus CUGUCUAUUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACU_____UCAUAUCACAGCCAU__UUUGACGAGUUUGAUGGGCAG_CCAGCCUCAUCAA ..(((((...((.(((((((.((.((((((..((((.................)))))))))).))...))))))).))....)))))................ (-19.23 = -19.17 + -0.06)


| Location | 8,985,257 – 8,985,353 |
|---|---|
| Length | 96 |
| Sequences | 13 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 69.95 |
| Shannon entropy | 0.66624 |
| G+C content | 0.44808 |
| Mean single sequence MFE | -32.04 |
| Consensus MFE | -17.81 |
| Energy contribution | -17.88 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.66 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 5.21 |
| SVM RNA-class probability | 0.999956 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 8985257 96 - 22422827 UUGAUGAGGCUGG-CUGUCCAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUAAUAGACAG .............-(((((.....(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).)))....))))) ( -33.90, z-score = -4.31, R) >droSim1.chrX_random 2632475 96 - 5698898 UUGAUGAGGCUGG-CUGCCCAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUGAUAGACAG .((.((.(((...-..))))).))(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).)))......... ( -35.20, z-score = -4.26, R) >droSec1.super_34 345612 96 - 457129 UUGAUGAGGCUGG-CUGCCCAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUGAUAGACAG .((.((.(((...-..))))).))(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).)))......... ( -35.20, z-score = -4.26, R) >droYak2.chrX 9440222 92 + 21770863 AUGAUGAGACUGG-----CCAGCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUACUAGACAG .........(((.-----(.((..(((.(((((((--((((((((((((((.-----.(......)..))))..))))))))))))))))).))).)).).))) ( -30.90, z-score = -3.72, R) >droEre2.scaffold_4690 12616408 92 + 18748788 AUGAUGGGGCU-----GCCCAACAAACUCGUCAAA--AUGGCUGUGAUAUGA-----AGUCAAAUCCACAUAGUUCACAGUCACUUUGACGCGCUAAUAGACAG .((.((((...-----.)))).))..(.(((((((--.(((((((((((((.-----.(......)..))))..))))))))).))))))).)........... ( -28.70, z-score = -3.08, R) >droAna3.scaffold_13047 797481 96 - 1816235 CCGAUGACUUUCA-CUGUCCACCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----CAUCAAAUACGCAUAAGUCACAGUCAUUUCGACGCGCUGGGAAACAG ..((......)).-((((...(((..(.((((.((--((((((((((((((.-----(.........))))..))))))))))))).)))).).)))...)))) ( -29.00, z-score = -3.34, R) >droPer1.super_13 1553262 97 - 2293547 UUGAUGGAGUGGGACUGCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----GAUCAAAAACACAUAGCUCACAGUCAUUUUGACGCGUAAAUGUGCAG ..............((((((((...((.(((((((--((((((((((((((.-----(........).))))..))))))))))))))))).))..)))))))) ( -39.80, z-score = -5.94, R) >dp4.chrXL_group1e 5522185 97 + 12523060 UUGAUGGAGUGGGACUGCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGA-----GAUCAAAAACACAUAGCUCACAGUCAUUUUGACGCGUAAAUGUGCAG ..............((((((((...((.(((((((--((((((((((((((.-----(........).))))..))))))))))))))))).))..)))))))) ( -39.80, z-score = -5.94, R) >droWil1.scaffold_180777 2053079 97 - 4753960 -----AAGAUUCGUAUGCCCAUCGAACUCGUCAAA--AUGGCUGUGAUAUGAAUUUCUGACCGGUUAACAUAGCUCACAGUCAUUUUGACGCGACCAUGGACAU -----.........(((.((((.(..(.(((((((--((((((((((((((.....((....))....))))..))))))))))))))))).)..))))).))) ( -32.20, z-score = -3.75, R) >droVir3.scaffold_12970 9304330 92 - 11907090 ----------UGGCUGCCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGAAUGGCAAAUGCAACAACAUAGCUCACAGUCAUUUUGACGCGCCAAUAUGGAG ----------((((((......))....(((((((--((((((((((((((....((....)).....))))..))))))))))))))))).))))........ ( -32.50, z-score = -3.98, R) >droMoj3.scaffold_6473 3600626 98 + 16943266 UUGCAGUUGGUGGCUGCCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGA----UAAUCUCAUAAACAUAGCUCACAGUCAAUUUGACGCGCAAAUAUGGAG ((((((((.((((...))))....))))(((((((--.((((((((((((((----.....)))))........))))))))).))))))).))))........ ( -31.90, z-score = -3.56, R) >droGri2.scaffold_15203 307012 92 - 11997470 ----------UGGCUGCCACAUCAAACUCGUCAAA--AUGGCUGUGAUAUGAAUUACAACUGCAACAACAUAGCUCACAGUCAAUUUGACGCGCCAAUAUGGAG ----------((((((......))....(((((((--.(((((((((((((.................))))..))))))))).))))))).))))........ ( -25.03, z-score = -2.44, R) >anoGam1.chr2R 35945410 87 + 62725911 ----------UCG-AUCUGCAGCAGCUUAGUGAAGUCCUGGUCGC-CCACGA----UCUCCACGUUCGUCUGGCUUUC-GCCCUUCUUGUACUCGAGCGGGCAG ----------...-.........((((.((((((((...(((((.-...)))----))...)).)))).))))))...-((((..((((....)))).)))).. ( -22.40, z-score = 0.94, R) >consensus UUGAUGAGGCUGG_CUGCCCAUCAAACUCGUCAAA__AUGGCUGUGAUAUGA_____AAUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGCUAAUAGACAG ..........................(.(((((((...(((((((((((((.................))))..))))))))).))))))).)........... (-17.81 = -17.88 + 0.06)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:27:18 2011