
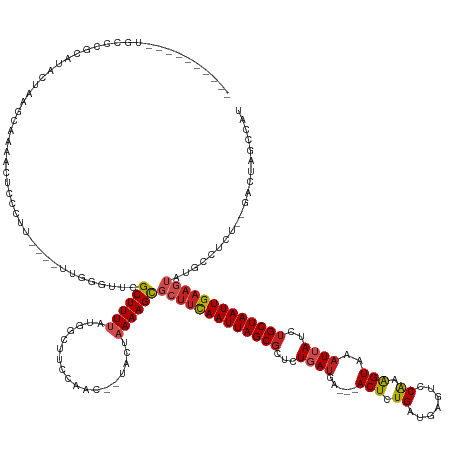
| Sequence ID | dm3.chrX |
|---|---|
| Location | 6,784,049 – 6,784,192 |
| Length | 143 |
| Max. P | 0.995302 |

| Location | 6,784,049 – 6,784,192 |
|---|---|
| Length | 143 |
| Sequences | 11 |
| Columns | 164 |
| Reading direction | forward |
| Mean pairwise identity | 72.22 |
| Shannon entropy | 0.55319 |
| G+C content | 0.41886 |
| Mean single sequence MFE | -36.65 |
| Consensus MFE | -17.54 |
| Energy contribution | -18.23 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.48 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.79 |
| SVM RNA-class probability | 0.995302 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 6784049 143 + 22422827 ----------UGCCCGCAUACUAAGCAAAACUCCCUU----UUGGGUUCGCUUUUAUGGCUUCCAAC--UACUAAAAGCGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCU--GACUAGCCAU ----------.....(((((((.(((......(((..----..)))...((((((((((.......)--)).))))))))))((((((((((....(((((---((((....))))..........))))))))))))))))))))))....--.......... ( -37.80, z-score = -2.90, R) >droSec1.super_4 6168844 122 - 6179234 ----------UGCCCGCAUACUAAGCAAAACUCCCUU----UUGGGUUCGCUUUUAUGGCUUCCAAC--UACUAAAAGCGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGA----------------------- ----------.............(((......(((..----..)))...((((((((((.......)--)).))))))))))((((((((((....(((((---((((....))))..........)))))))))))))))----------------------- ( -27.80, z-score = -1.56, R) >droYak2.chrX 7198957 145 - 21770863 ----------UGCCCGCAUACUAAGCAAAACUCCCUU----UUGGGUUCGCUUUUAUGGCUUCCAAC--UACUAAAAGCGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCUAUGACUAGCCAU ----------.....(((((((.(((......(((..----..)))...((((((((((.......)--)).))))))))))((((((((((....(((((---((((....))))..........))))))))))))))))))))))................ ( -37.80, z-score = -2.84, R) >droEre2.scaffold_4690 15711833 143 + 18748788 ----------UGCCCGCAUACUAAGCAAAACUCCCUU----UUGGGUUCGCUUUUAUGGCUUCCAAC--UACUAAAAGCGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCU--GACUAGCCAU ----------.....(((((((.(((......(((..----..)))...((((((((((.......)--)).))))))))))((((((((((....(((((---((((....))))..........))))))))))))))))))))))....--.......... ( -37.80, z-score = -2.90, R) >droAna3.scaffold_13117 1622680 147 - 5790199 ----------UACCAGGAACCUUGCAGAGUUGAGUUCCCUUUUGGUUUCGCUUUUAUGGCCUCGGCU---ACUAAAAGCGCUUUAAUUAGCGCUCUGAUGA---ACUCUGAUGAGCCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUUA-CUCCUAACCCA ----------....((((.....(((.....(((...((....)).)))(((((((((((....)))---).)))))))((((((((((((((((....(.---...)....))))....((........)))))))))))))).)))....-.))))...... ( -38.70, z-score = -2.11, R) >dp4.Unknown_group_30 84718 150 - 91206 UACUCAAGAGUUCCCGCAUGCUUAG--GAGCUACCUU----UUGGCUUCGCUUU-AUGACUGCCAAC--UACUAAAAGUGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGCCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCC-UGAC-AGCCCU ...............((.((.((((--((((......----(((((.(((....-.)))..))))).--........((((((((((((((((((....(.---...)....))))....((........))))))))))))))))).))))-))))-)))... ( -40.60, z-score = -2.63, R) >droPer1.super_26 725971 150 + 1349181 UACUCAAGAGUUCCCGCAUGCUUAG--GAGCUACCUU----UUGGCUUCGCUUU-AUGACUGCCAAC--UACUAAAAGUGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGCCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCC-UGAC-AGCCCU ...............((.((.((((--((((......----(((((.(((....-.)))..))))).--........((((((((((((((((((....(.---...)....))))....((........))))))))))))))))).))))-))))-)))... ( -40.60, z-score = -2.63, R) >droWil1.scaffold_180777 2856799 152 + 4753960 ----------UGCCCGCAUCCUUGACAAGAUGCCUUUGCCAUUGCUUUUGCUUUUAUGGUUUCAACUGGUACUAAAAGCGCUUCAAUUAGCUCACUGACGACGAACUCUGAUGAGACCAAAGUAAAUUAUGUGCUAAUUGAAGUAUGCGUAU--GACUGACAAC ----------((((.(((((........)))))....(((((.((....))....))))).......))))......((((((((((((((.((.(((..((...(((....)))......))...))))).))))))))))))..))....--.......... ( -35.90, z-score = -1.59, R) >droVir3.scaffold_12928 7510671 134 - 7717345 --------------CACACACAUGAGCACGCACAUUC-----ACCUUUUGCUUUUAUGGCUGCAACU------AAAAGCGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGUGCAAGGUAAAUUAUGUGCUAAUUGAAGUAUGCGUAA--AACUGUCUAU --------------.............(((((.....-----.......(((((((.(.......))------))))))((((((((((((((.((..((.---((((....)))).)).))........)))))))))))))).)))))..--.......... ( -36.10, z-score = -1.55, R) >droMoj3.scaffold_6359 2976152 118 - 4525533 ----------------CACACACAGGCACACACACUU-----ACCUUUGGCUUUUAUGGCUUCAACU------AAAAGCGCUUCAAUUAGCGCUCUGAUGA---ACUCUGAUGAGUCCGAAGUAAAUGAUGUGCUAAUUGCACUGUGC---------------- ----------------....((((((((((((((.((-----((.((((((((....(..((((.(.------...((((((......))))))..).)))---)..)....))).))))))))).)).)))).....))).))))).---------------- ( -36.10, z-score = -3.22, R) >droGri2.scaffold_15203 10125947 127 - 11997470 --------------CACACUAAAAACCAAGCACACUC-----GCCCU-AGCUUUUAUGACUUAAAGU------AAAAGCUCUUCAAUUAGCGCUCUGAUGA---ACUUUGAUGAGACCAAAGUAAAUGAUGUGCUAAUUGAAGUAUGCGUAC--AACU------ --------------...............(((.....-----.....-(((((((((........))------)))))))(((((((((((((..(.((..---((((((.......))))))..)).).)))))))))))))..)))....--....------ ( -34.00, z-score = -4.24, R) >consensus __________UGCCCGCAUACUAAGCAAAACUCCCUU____UUGGGUUCGCUUUUAUGGCUUCCAAC__UACUAAAAGCGCUUCAAUUAGCGCUCUGAUGA___ACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCU__GACUAGCCAU .................................................((((((..................))))))(((((((((((((...((((.....(((.((.......)).)))..))))..))))))))))))).................... (-17.54 = -18.23 + 0.70)

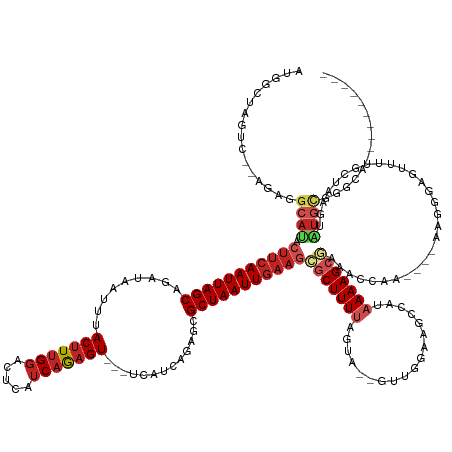
| Location | 6,784,049 – 6,784,192 |
|---|---|
| Length | 143 |
| Sequences | 11 |
| Columns | 164 |
| Reading direction | reverse |
| Mean pairwise identity | 72.22 |
| Shannon entropy | 0.55319 |
| G+C content | 0.41886 |
| Mean single sequence MFE | -40.89 |
| Consensus MFE | -18.82 |
| Energy contribution | -20.39 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.46 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.961454 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 6784049 143 - 22422827 AUGGCUAGUC--AGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUA--GUUGGAAGCCAUAAAAGCGAACCCAA----AAGGGAGUUUUGCUUAGUAUGCGGGCA---------- ..(((((.(.--((((((...(((((((((((.((....)).((.(((((((....))))---)))..))...))))))))))).))))))).))--)))....(((....((((((((((..----..)))...))))))).(....).))).---------- ( -43.40, z-score = -2.32, R) >droSec1.super_4 6168844 122 + 6179234 -----------------------UCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUA--GUUGGAAGCCAUAAAAGCGAACCCAA----AAGGGAGUUUUGCUUAGUAUGCGGGCA---------- -----------------------(((((((...............(((((((....))))---)))..((((((((......))))))))...))--)))))..(((....((((((((((..----..)))...))))))).(....).))).---------- ( -35.60, z-score = -2.31, R) >droYak2.chrX 7198957 145 + 21770863 AUGGCUAGUCAUAGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUA--GUUGGAAGCCAUAAAAGCGAACCCAA----AAGGGAGUUUUGCUUAGUAUGCGGGCA---------- ...(((.(.((((.(((((..(((((((((((.((....)).((.(((((((....))))---)))..))...)))))))))))((((((((...--((.....))..))))))))..(((..----..))).....)))))..))))).))).---------- ( -45.10, z-score = -2.75, R) >droEre2.scaffold_4690 15711833 143 - 18748788 AUGGCUAGUC--AGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUA--GUUGGAAGCCAUAAAAGCGAACCCAA----AAGGGAGUUUUGCUUAGUAUGCGGGCA---------- ..(((((.(.--((((((...(((((((((((.((....)).((.(((((((....))))---)))..))...))))))))))).))))))).))--)))....(((....((((((((((..----..)))...))))))).(....).))).---------- ( -43.40, z-score = -2.32, R) >droAna3.scaffold_13117 1622680 147 + 5790199 UGGGUUAGGAG-UAAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGGCUCAUCAGAGU---UCAUCAGAGCGCUAAUUAAAGCGCUUUUAGU---AGCCGAGGCCAUAAAAGCGAAACCAAAAGGGAACUCAACUCUGCAAGGUUCCUGGUA---------- ..((((.((((-((.....))))))(((((((.((....)).((.(((((((....))))---)))..))...)))))))....((((((((..---.((....))..)))))))).))))....(((((((...........)))))))....---------- ( -40.10, z-score = -1.10, R) >dp4.Unknown_group_30 84718 150 + 91206 AGGGCU-GUCA-GGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGGCUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCACUUUUAGUA--GUUGGCAGUCAU-AAAGCGAAGCCAA----AAGGUAGCUC--CUAAGCAUGCGGGAACUCUUGAGUA ......-.(((-((((((...(((((((((((.((....)).((.(((((((....))))---)))..))...))))))))))).((((((....--..((((.((...-...))...)))))----))))).))((--((........)))).)))))))... ( -40.80, z-score = -0.21, R) >droPer1.super_26 725971 150 - 1349181 AGGGCU-GUCA-GGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGGCUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCACUUUUAGUA--GUUGGCAGUCAU-AAAGCGAAGCCAA----AAGGUAGCUC--CUAAGCAUGCGGGAACUCUUGAGUA ......-.(((-((((((...(((((((((((.((....)).((.(((((((....))))---)))..))...))))))))))).((((((....--..((((.((...-...))...)))))----))))).))((--((........)))).)))))))... ( -40.80, z-score = -0.21, R) >droWil1.scaffold_180777 2856799 152 - 4753960 GUUGUCAGUC--AUACGCAUACUUCAAUUAGCACAUAAUUUACUUUGGUCUCAUCAGAGUUCGUCGUCAGUGAGCUAAUUGAAGCGCUUUUAGUACCAGUUGAAACCAUAAAAGCAAAAGCAAUGGCAAAGGCAUCUUGUCAAGGAUGCGGGCA---------- ..((((.(((--((..((...(((((((((((.(((.....((((((((...)))))))).........))).))))))))))).(((((((................)))))))....)).)))))....(((((((....))))))).))))---------- ( -45.13, z-score = -3.27, R) >droVir3.scaffold_12928 7510671 134 + 7717345 AUAGACAGUU--UUACGCAUACUUCAAUUAGCACAUAAUUUACCUUGCACUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCGCUUUU------AGUUGCAGCCAUAAAAGCAAAAGGU-----GAAUGUGCGUGCUCAUGUGUGUG-------------- ..........--...(((((((........((((((...(((((((((((((....))))---.....((((((((......)))))))).------....)).((.......))..)))))-----))))))))........)))))))-------------- ( -38.69, z-score = -1.87, R) >droMoj3.scaffold_6359 2976152 118 + 4525533 ----------------GCACAGUGCAAUUAGCACAUCAUUUACUUCGGACUCAUCAGAGU---UCAUCAGAGCGCUAAUUGAAGCGCUUUU------AGUUGAAGCCAUAAAAGCCAAAGGU-----AAGUGUGUGUGCCUGUGUGUG---------------- ----------------((((((.((.....(((((.(((((((((.((((((....))))---))...((((((((......)))))))).------.....................))))-----))))))))))))))))))...---------------- ( -41.10, z-score = -3.47, R) >droGri2.scaffold_15203 10125947 127 + 11997470 ------AGUU--GUACGCAUACUUCAAUUAGCACAUCAUUUACUUUGGUCUCAUCAAAGU---UCAUCAGAGCGCUAAUUGAAGAGCUUUU------ACUUUAAGUCAUAAAAGCU-AGGGC-----GAGUGUGCUUGGUUUUUAGUGUG-------------- ------....--((((((...(((((((((((.(.((....((((((((...))))))))---......))).))))))))))).(((((.------.((((........))))..-)))))-----..))))))...............-------------- ( -35.70, z-score = -2.54, R) >consensus AUGGCUAGUC__AGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGU___UCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUA__GUUGGAAGCCAUAAAAGCGAAACCAA____AAGGGAGUUUUGCUAAGUAUGCGGGCA__________ ................((((.(((((((((((.........(((((((.....)))))))...((....))..)))))))))))(((((((..................))))))).............................))))............... (-18.82 = -20.39 + 1.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:22:13 2011