
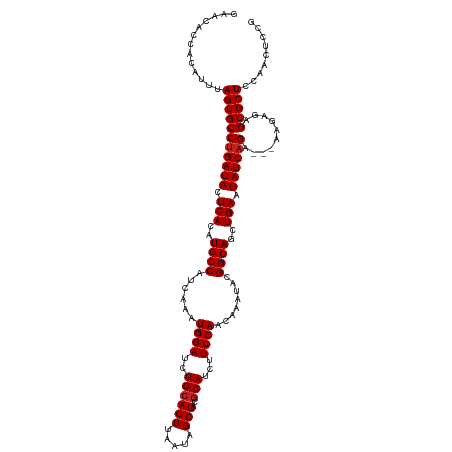
| Sequence ID | dm3.chrX |
|---|---|
| Location | 5,293,641 – 5,293,758 |
| Length | 117 |
| Max. P | 0.951160 |

| Location | 5,293,641 – 5,293,758 |
|---|---|
| Length | 117 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.62 |
| Shannon entropy | 0.05179 |
| G+C content | 0.45178 |
| Mean single sequence MFE | -25.23 |
| Consensus MFE | -23.90 |
| Energy contribution | -24.07 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.95 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.699894 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
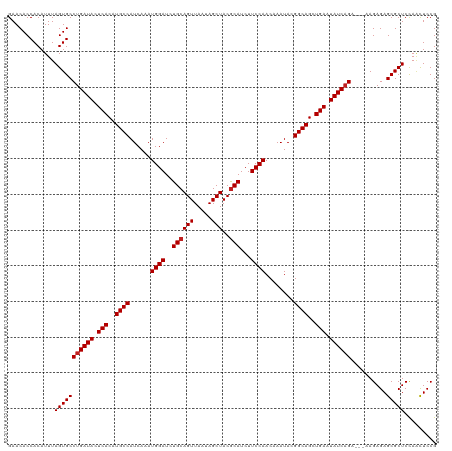
>dm3.chrX 5293641 117 + 22422827 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.42, R) >droSim1.chrX 4070958 117 + 17042790 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.42, R) >droSec1.super_4 4717787 117 - 6179234 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.42, R) >droYak2.chrX 18594694 117 - 21770863 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.42, R) >droEre2.scaffold_4690 2647718 117 + 18748788 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.42, R) >droAna3.scaffold_13117 2744390 117 + 5790199 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCACUUCCG .............((((.((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---..(((....))))))).... ( -25.34, z-score = -1.44, R) >dp4.chrXL_group1e 10885277 120 + 12523060 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGUUGAAUAUCAGAGAGAAGAGAGCGCUCCAGUUCCG ((((..(((.....))).((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).))))))......(((....)))..)))).. ( -27.14, z-score = -1.18, R) >droPer1.super_14 327270 120 + 2168203 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGUUGAAUAUCAGAGAGAAGAGAGCGCUCCAGUUCCG ((((..(((.....))).((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).))))))......(((....)))..)))).. ( -27.14, z-score = -1.18, R) >droWil1.scaffold_181096 9003027 117 + 12416693 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACACAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCUAAUUCCG ......(((.....))).((((((.(((..((((......((((..((.(((.....))).)).....))))........))))..))).)))))).---.((((....))))....... ( -22.94, z-score = -1.07, R) >droVir3.scaffold_12932 1831568 117 - 2102469 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCCGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).))))(..---..))).)))))......... ( -24.84, z-score = -1.49, R) >droMoj3.scaffold_6308 298430 117 + 3356042 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---CAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.35, R) >droGri2.scaffold_14853 2508446 117 + 10151454 GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA---AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).)))))).---......)))))......... ( -25.05, z-score = -1.42, R) >consensus GAACACCACAUUUAGUGCCUGAUACUCACAUGCCAUCAAAUGGAUCUGCAGUUAAUAGCUACGCAUCUUCCAACAAAUACGGCAGCUGAAUAUCAGA___AAGAGAGCGCUCCAACUCCG .............(((((((((((.(((..((((......((((..((((((.....)))..)))...))))........))))..))).))))))..........)))))......... (-23.90 = -24.07 + 0.17)

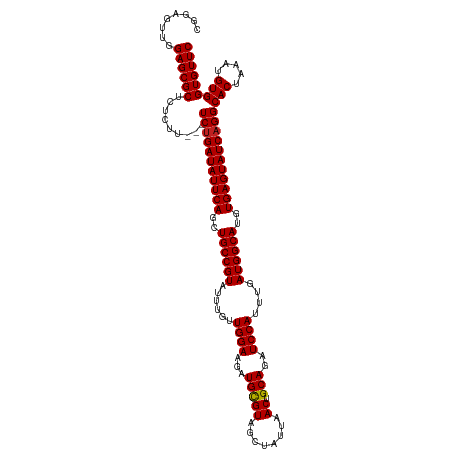
| Location | 5,293,641 – 5,293,758 |
|---|---|
| Length | 117 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.62 |
| Shannon entropy | 0.05179 |
| G+C content | 0.45178 |
| Mean single sequence MFE | -37.83 |
| Consensus MFE | -37.25 |
| Energy contribution | -37.26 |
| Covariance contribution | 0.01 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.98 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.57 |
| SVM RNA-class probability | 0.951160 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
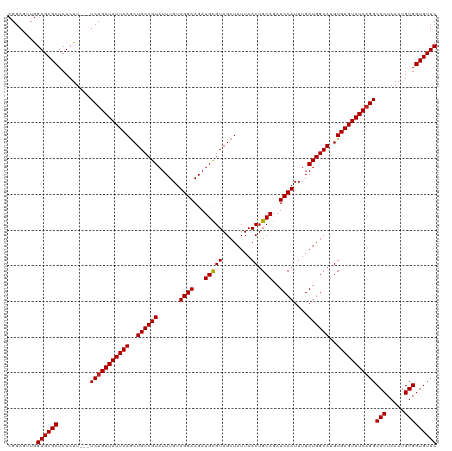
>dm3.chrX 5293641 117 - 22422827 CGGAGUUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -37.90, z-score = -1.80, R) >droSim1.chrX 4070958 117 - 17042790 CGGAGUUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -37.90, z-score = -1.80, R) >droSec1.super_4 4717787 117 + 6179234 CGGAGUUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -37.90, z-score = -1.80, R) >droYak2.chrX 18594694 117 + 21770863 CGGAGUUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -37.90, z-score = -1.80, R) >droEre2.scaffold_4690 2647718 117 - 18748788 CGGAGUUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -37.90, z-score = -1.80, R) >droAna3.scaffold_13117 2744390 117 - 5790199 CGGAAGUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC (....)..((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -38.10, z-score = -1.91, R) >dp4.chrXL_group1e 10885277 120 - 12523060 CGGAACUGGAGCGCUCUCUUCUCUCUGAUAUUCAACUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((.........(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -38.00, z-score = -2.12, R) >droPer1.super_14 327270 120 - 2168203 CGGAACUGGAGCGCUCUCUUCUCUCUGAUAUUCAACUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((.........(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -38.00, z-score = -2.12, R) >droWil1.scaffold_181096 9003027 117 - 12416693 CGGAAUUAGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGUGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(.(((((.......))))).)))))....))))))..)))))))))))(((.....))))))))) ( -38.60, z-score = -2.68, R) >droVir3.scaffold_12932 1831568 117 + 2102469 CGGAGUUGGAGCGCUCUCUU---UCGGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ((((...((((....)))))---)))((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))).(((((((....))))))). ( -34.30, z-score = -0.66, R) >droMoj3.scaffold_6308 298430 117 - 3356042 CGGAGUUGGAGCGCUCUCUG---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........(((((((....(---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..))))))))))))((.....))))))))) ( -39.50, z-score = -2.10, R) >droGri2.scaffold_14853 2508446 117 - 10151454 CGGAGUUGGAGCGCUCUCUU---UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((......---(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) ( -37.90, z-score = -1.80, R) >consensus CGGAGUUGGAGCGCUCUCUU___UCUGAUAUUCAGCUGCCGUAUUUGUUGGAAGAUGCGUAGCUAUUAACUGCAGAUCCAUUUGAUGGCAUGUGAGUAUCAGGCACUAAAUGUGGUGUUC ........((((((.........(((((((((((..((((((......((((...(((((........)).)))..))))....))))))..)))))))))))(((.....))))))))) (-37.25 = -37.26 + 0.01)

Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:17:37 2011