
| Sequence ID | dm3.chrX |
|---|---|
| Location | 4,006,788 – 4,006,905 |
| Length | 117 |
| Max. P | 0.763195 |

| Location | 4,006,788 – 4,006,905 |
|---|---|
| Length | 117 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.41 |
| Shannon entropy | 0.61637 |
| G+C content | 0.53466 |
| Mean single sequence MFE | -43.00 |
| Consensus MFE | -16.20 |
| Energy contribution | -16.13 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.61 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.763195 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 4006788 117 + 22422827 ---AGCCGUCCGGCUUCGUGGUCACGAGAUUGGGAAAGACACUGCUGUCGUGGUUGGUCACCAACAGGCAGGGCGAGUUUAGACGCUUGGCUUGGUGAACCAGCAUGUAGCACCGAUUGU ---((((....))))((((....))))((((((.........(((((.((((.((((((((((...(((..((((........))))..)))))))).)))))))))))))))))))).. ( -43.60, z-score = -0.94, R) >droWil1.scaffold_181150 2540757 117 - 4952429 ---AGCCAUCUGGCUGCGUUCUUAUCAAAUUGGGAAAAACGCUGCUGUCAUGAUUGGUCACCAGCAGACAGGGUGAGUUUAGUCUUUUGGCCUGAUGGACCAACAUAUAACAGCGAUUAU ---((((....))))(((((((((......)))))...))))((((((.(((.((((((.((((.((((..((....))..)))).)))).(....))))))))))...))))))..... ( -34.00, z-score = -0.94, R) >droPer1.super_60 32795 117 + 401158 ---CACCGUCCUCGUUGCUCUGCAACAGGUUCGGAAAGAUGCUGCUGUCGUGGUUGGUCACCAGCAGGCAGGGCGAGUUCAGACGCUUUGCCUGGUGAACCAGCAUGUAGCAGCGAUUAU ---..(((.(((.(((((...))))))))..))).....((((((((.((((.((((((((((...(((((((((........)))))))))))))).)))))))))))))))))..... ( -57.20, z-score = -4.63, R) >dp4.chrXL_group1e 12224402 117 - 12523060 ---CACCGUCCUCGUUGCUCUGCAACAGGUUCGGAAAGAUGCUGCUGUCGUGGUUUGUCACCAGCAGGCAGGGUGAAUUCAGACGCUUUGCCUGGUGAACCAGCAUGUAGCAGCGAUUAU ---..(((.(((.(((((...))))))))..))).....((((((((.((((.(...((((((...(((((((((........)))))))))))))))...).))))))))))))..... ( -50.90, z-score = -3.52, R) >droAna3.scaffold_13117 177085 117 - 5790199 ---AGUUGUCCGGAUUAGUGACCAGAAGGUUGGGGAAGACGCUGCUGUCGUGAUUCGUGACCAGAAGGCACGGGGAGUUGAGCCGCUUGGCCUGGUGGACCAGCAUGUAGCAGCGAUUGU ---..((.(((........((((....))))))).))..((((((((.(((..((((((.((....))))))))..((((..(((((......)))))..)))))))))))))))..... ( -39.70, z-score = -0.34, R) >droEre2.scaffold_4690 1383556 117 + 18748788 ---AGCCGUCAGGCUUUGUGGUCAGGAGAUUGGGAAAGACACUGCUGUCGUGAUUGGUCACCAACAGGCAGGGCGAGUUAAGGCGCUUGGCCUGGUGAACCAGCAUGUAGCAGCGGUUGU ---.(((....)))(((.(((((....))))).)))..((((((((((((((.((((((((((...(((..((((........))))..)))))))).)))))))))..))))))).))) ( -47.20, z-score = -2.02, R) >droYak2.chrX 17348705 117 - 21770863 ---AGCCGUCAGGCAUUGUGGUCAAGAGAUUGGGAAAGACACUGCUGUCGUGGUUGGUCACCAACAGGCAGGGCGAGUUAAGGCGCUUGGCCUGGUGAACCAGCAUGUAGCAGCGGUUGU ---.(((....))).((.(((((....))))).))...((((((((((..(((((((((((((...(((..((((........))))..)))))))).)))))).))..))))))).))) ( -47.30, z-score = -2.23, R) >droSec1.super_4 3464086 117 - 6179234 ---AGCCGUCCGGCUUCGUGGUCAGGAGAUUGGGAAAGGCACUGCUGUCGUGGUUGGUCACCAACAAGCAGGGCGAGUUUAGGCGUUUGGCUUGGUGAACCAGCAUGUAGCAGCGGUUGU ---.(((.(((((((((.......)))).)))))...)))((((((((..(((((((((((((...(((..((((........))))..)))))))).)))))).))..))))))))... ( -49.20, z-score = -2.66, R) >droSim1.chrX 2979147 117 + 17042790 ---AGCCGUCCGGCUUCGUGGUCAGGAGAUUGGGAAAGGCACUGCUGUCGUGGUUGGUCACCAACAGGCAGGGCGAGUUUAGGCGCUUGGCUUGGUGAACCAGCAUGUAGCAGCGGUUGU ---.(((.(((((((((.......)))).)))))...)))((((((((..(((((((((((((...(((..((((........))))..)))))))).)))))).))..))))))))... ( -51.70, z-score = -2.74, R) >droGri2.scaffold_14853 2922233 114 - 10151454 ---UGCCAUCCACC---GUUUGGACCAGAUUGGGAAAGACGCUGCUGUCAUGGUUGGUCACCAGCAGGCAUGGCGAAUUGAGGCGCUUGGCCUGAUGCACCAGCAUAUAGCAGCGAUUGU ---.....(((.(.---(((((...))))).))))....(((((((((....((((((((....(((((..((((........))))..))))).)).))))))..)))))))))..... ( -42.30, z-score = -0.93, R) >droVir3.scaffold_12928 6643045 120 + 7717345 UGCCGGUGGCGGCAUCGGUCUGCAGCAGAUUGGGAAAGAAACUGCUGUCGUGAUUGGUCACCAGCAGACAGGGCGAAUUGAGCCGUUUGGCCUGAUGCACCAGCAUAUAGCAGCGAUUGU (((((....)))))((((((((...))))))))........(((((((.(((.(((((.....(((..((((.(((((......))))).)))).))))))))))))))))))....... ( -42.50, z-score = -0.20, R) >droMoj3.scaffold_6328 2322261 120 + 4453435 CGUUGGUGGCUACAUCCGUUUGCAGCAGAUUGGGAAAGAAGCUGCUGUCGUGGUUGGUCACCAGCAGACAGGGGGAAUUCAGGCGUUUGGCCUGGUGGACCAGCAUAUAGCAGCGAUUGU .(((((((((((...(((...(((((((.((........)))))))))..))).)))))))))))..((((.((....((((((.....))))))....)).((.....)).....)))) ( -43.80, z-score = -1.27, R) >anoGam1.chr2R 22068152 117 + 62725911 ---UACCGUCGUCCUUCGUGUACUGCAUCGAGGGAAAGGUCGAGCUGUCCCCGUUCGUUACCACAAAGCACGGCGAGUUGAGGCGCUUCGCCUGGUGCACCAGCAUGUAGCACCGGUUGU ---.((((((((((((((((.....)).)))))))..((((((((.......)))))..)))........))))......(((((...)))))(((((((......)).))))))))... ( -40.20, z-score = -0.15, R) >apiMel3.Group4 9291843 117 - 10796202 ---UACCAUCUGGUUUAGUAGUAGUAAAAUUAGGCAAUAUUGUGGCAUCAUGAUUUGUAAUUAAAACAAUUGGACUGUUUAAUCUUUUCGAUUGAUGUACAAGCAUGUAACAUCGAUUAU ---........((((((((.((..(((......((((.((..((....))..))))))..)))..)).)))))))).............((((((((((((....))).))))))))).. ( -20.50, z-score = 0.16, R) >triCas2.ChLG7 16079035 117 - 17478683 ---ACCCAUCAGGCAGAGACGCCGAUAGGUUGGGUAAAAUUGCGCUAUCAUGGUUGAUUACAGCCACACACGGCGAAUUCAGCCUUUUAGCUUGGUGCACCAACAUAUAACACCGCUUGU ---(((((.(.(((......))).....).)))))......((((((....((((((.....(((......)))....))))))...))))..((((.............)))))).... ( -34.92, z-score = -1.68, R) >consensus ___AGCCGUCCGGCUUCGUGGUCAGCAGAUUGGGAAAGACACUGCUGUCGUGGUUGGUCACCAGCAGGCAGGGCGAGUUUAGGCGCUUGGCCUGGUGAACCAGCAUGUAGCAGCGAUUGU ...........................................(((((.(((.(((((((((((.((((...............))))...)))))).))))))))...)))))...... (-16.20 = -16.13 + -0.08)

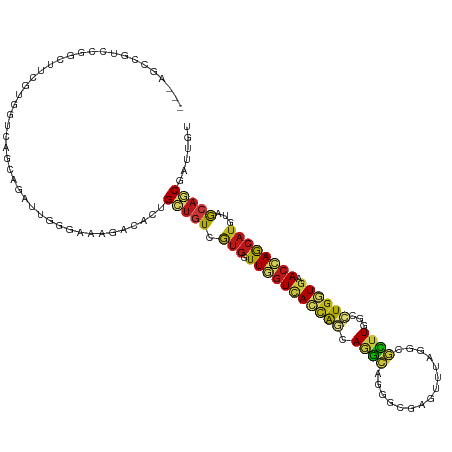
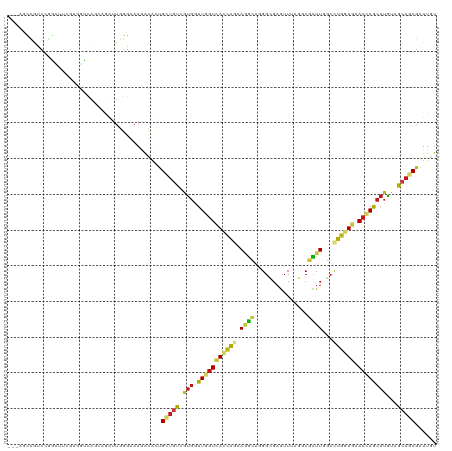
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:13:28 2011