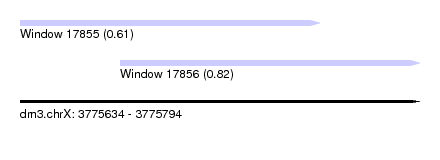
| Sequence ID | dm3.chrX |
|---|---|
| Location | 3,775,634 – 3,775,794 |
| Length | 160 |
| Max. P | 0.815538 |

| Location | 3,775,634 – 3,775,754 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.35 |
| Shannon entropy | 0.37112 |
| G+C content | 0.49889 |
| Mean single sequence MFE | -32.24 |
| Consensus MFE | -17.95 |
| Energy contribution | -17.46 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.58 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.609606 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 3775634 120 + 22422827 GUCCAACUUUCUGGACUUCAUUACCUACGGUUAUAUGAUCGACAACAUCAUUCUGCUGAUCACUGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCACCCUCUGGG (((((......))))).......((((((((...(((((.......)))))...)))).....((((((((((...))))..........(((.....)))....)))))).....)))) ( -31.90, z-score = -1.69, R) >droEre2.scaffold_4690 1159540 120 + 18748788 GUCCAACUUCCUGGACUUCAUUACCUACGGCUAUAUGAUCGACAACAUCAUUCUGCUGAUCACUGGCACGCUGCAUCAGCGACCCAUUUCGGAGCUGAUUCCUAAGUGCCACCCUCUGGG (((((......))))).......((((((((...(((((.......)))))...)))).....((((((...(.((((((..((......)).)))))).)....)))))).....)))) ( -36.00, z-score = -3.26, R) >droYak2.chrX 17131424 120 - 21770863 GUCCAACUUCCUGGACUUCAUUACCUACGGCUAUAUGAUCGACAACAUCAUUCUGCUGAUCACUGGAACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCAUCCGCUGGG (((((......)))))...........(((((...((((((.((.........)).)))))).(((..(((((...)))))..)))......)))))...((((.(((.....))))))) ( -32.70, z-score = -1.71, R) >droSec1.super_4 3247094 120 - 6179234 GUCCAACUUUCUGGACUUCAUUACCUACGGCUAUAUGAUCGACAACAUCAUUCUGCUGAUCACUGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCACCCUCUGGG (((((......))))).......((((((((...(((((.......)))))...)))).....((((((((((...))))..........(((.....)))....)))))).....)))) ( -33.50, z-score = -2.31, R) >droSim1.chrX 2781355 120 + 17042790 GUCCAACUUUCUGGACUUCAUUACCUACGGCUAUAUGAUCGACAACAUCAUUCUGCUGAUCACUGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCACCCUCUGGG (((((......))))).......((((((((...(((((.......)))))...)))).....((((((((((...))))..........(((.....)))....)))))).....)))) ( -33.50, z-score = -2.31, R) >droWil1.scaffold_181096 11868718 120 + 12416693 GUCCAAUUUCCUAGACUUUAUCACCUAUGGUUAUAUGAUUGAUAAUAUCAUUUUGCUGAUCACCGGAACUCUGCAUCAGCGUCCCAUUUCGGAGCUAAUUCCCAAAUGCCAUCCAUUGGG ..(((((......(((.((((((..((((....))))..)))))).........((((((...(((....))).))))))))).(((((.((((....)))).)))))......))))). ( -25.60, z-score = -0.98, R) >droMoj3.scaffold_6328 3926820 120 + 4453435 GUCGAACUUCCUCGACUUCAUCACGUAUGGCUAUAUGAUUGACAACAUCAUAUUGUUGAUCACGGGCACACUCCAUCAGCGCCCCAUCUCGGAGCUGAUACCCAAGUGUCAUCCGUUGGG (((((......)))))....(((((.((((((...((((..((((.......))))..))))..)))(((((..((((((.((.......)).)))))).....)))))))).)).))). ( -35.00, z-score = -1.85, R) >droGri2.scaffold_15081 1436192 120 - 4274704 AUCCAAUUUUUUGGACUUCAUCACGUAUGGUUAUAUGAUUGAUAAUAUCAUAUUGCUGAUAACGGGCACACUGCAUCAGCGACCCAUCUCGGAGCUGAUACCCAAGUGCCAUCCGCUGGG .(((((....)))))..(((.(.((((((((.(((((((.......))))))).))).)).)))(((((...(.((((((..((......)).)))))).)....)))))....).))). ( -31.60, z-score = -0.94, R) >droVir3.scaffold_13042 4662847 120 + 5191987 GUCCAAUUUCUUGGACUUUAUCACGUACGGCUAUAUGAUCGAUAAUAUUAUAUUGUUGAUCACGGGCACGCUGCACCAGCGUCCCAUUUCGGAGCUGAUACCCAAAUGCCAUCCGCUGGG ((((((....))))))...........(((((...((((((((((((...)))))))))))).(((.((((((...))))))))).......)))))...((((...((.....)))))) ( -41.80, z-score = -4.02, R) >droPer1.super_14 1711553 120 + 2168203 AUCCAACUUCCUGGACUUCAUCACCUAUGGCUAUAUGAUCGACAACAUCAUCCUGCUCAUUACGGGUACGCUGCACCAGCGUCCCAUUUCGGAGCUGAUACCCAAGUGCCAUCCGCUGGG .((((......))))..(((.(.((.(((((...(((((.......)))))...)).)))...(((.((((((...))))))))).....)).).)))..((((.(((.....))))))) ( -31.70, z-score = -1.09, R) >dp4.chrXL_group1e 621745 120 - 12523060 AUCCAACUUCCUGGACUUCAUCACCUAUGGCUAUAUGAUCGACAACAUCAUCCUGCUCAUUACGGGCACGCUGCACCAGCGUCCCAUUUCGGAGCUGAUACCCAAGUGCCAUCCGCUGGG .((((......))))..(((.(.((.(((((...(((((.......)))))...)).)))...(((.((((((...))))))))).....)).).)))..((((.(((.....))))))) ( -31.70, z-score = -1.02, R) >droAna3.scaffold_13117 714207 120 + 5790199 GUCUAACUUCCUGGACUUCAUCACCUACGGGUACAUGAUCGACAACAUCAUCCUGCUGAUCACCGGCACCCUGCACCAGCGCCCCAUCUCGGAGCUGAUCCCCAAGUGCCAUCCCCUGGG (((((......))))).......((((.(((....((((((.((.........)).))))))..(((((.......((((.((.......)).))))........)))))..))).)))) ( -32.86, z-score = -1.23, R) >anoGam1.chrX 13134904 120 + 22145176 GUCCACCUUCCUGGACUACAUCACGUACAGCUACAUGAUCGACAACAUCAUCCUGCUGAUCACGGGCACGCUGCACCAGCGCCCGAUCUCGGAGCUCAUCCCGAAGUGCCACCCGCUCGG (((((......))))).......(((.((((...(((((.......)))))...))))...)))(((((((((...))))(((((....))).))..........))))).......... ( -36.00, z-score = -1.97, R) >apiMel3.GroupUn 397709700 120 - 399230636 AUCACAAUUUUUAGAUUUUAUUACUUAUAGUUAUAUGAUUGAUAAUAUUAUCUUACUAAUUACUGGUACUUUGCAUCAAAGACCAAUUUCAGAAUUGAUUCCUAAAUGUCACCCGCUUGG ....(((((((.((((.((((((.(((((....))))).))))))....))))..........((((.(((((...))))))))).....)))))))....................... ( -16.90, z-score = -0.75, R) >triCas2.ChLG5 7104736 120 - 18847211 GUCUACGUUUUUGGAUUUUAUCACUUACAGUUACAUGAUUGAUAAUAUUAUUUUACUGAUUACGGGGACGUUGCACCAGCGCCCCAUUGGGGAGCUGAUCCCGAAAUGCCACCCCUUGGG .....((((((.(((((...((.((((((((...(((((.......)))))...)))).....((((.(((((...)))))))))..))))))...))))).))))))(((.....))). ( -32.90, z-score = -1.68, R) >consensus GUCCAACUUCCUGGACUUCAUCACCUACGGCUAUAUGAUCGACAACAUCAUUCUGCUGAUCACGGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCAUCCGCUGGG (((((......)))))...........((((...(((((.......)))))...))))......((((........((((...((.....)).)))).........)))).......... (-17.95 = -17.46 + -0.48)


| Location | 3,775,674 – 3,775,794 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.16 |
| Shannon entropy | 0.36002 |
| G+C content | 0.55556 |
| Mean single sequence MFE | -42.27 |
| Consensus MFE | -24.98 |
| Energy contribution | -24.25 |
| Covariance contribution | -0.73 |
| Combinations/Pair | 1.73 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.59 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.815538 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 3775674 120 + 22422827 GACAACAUCAUUCUGCUGAUCACUGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCACCCUCUGGGCAGCUUUGAGCAGAUGGAAGCCAUCCAUGUGGCGUCCACU ...........(((((.......(((..(((((...)))))..)))..(((((((((...((((((.......)).))))))))))))))))))((((.(((((....))))).)))).. ( -42.30, z-score = -1.42, R) >droEre2.scaffold_4690 1159580 120 + 18748788 GACAACAUCAUUCUGCUGAUCACUGGCACGCUGCAUCAGCGACCCAUUUCGGAGCUGAUUCCUAAGUGCCACCCUCUGGGCAGCUUUGAGCAAAUGGAAGCCAUCCAUGUGGCGUCCACU .......(((....((((.....((((((...(.((((((..((......)).)))))).)....))))))(((...)))))))..))).....((((.(((((....))))).)))).. ( -39.70, z-score = -1.38, R) >droYak2.chrX 17131464 120 - 21770863 GACAACAUCAUUCUGCUGAUCACUGGAACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCAUCCGCUGGGCAGCUUCGAGCAGAUGGAAGCCAUCCAUGUGGCGUCCACU ...........(((((.......(((..(((((...)))))..)))..(((((((((...((((.(((.....)))))))))))))))))))))((((.(((((....))))).)))).. ( -47.90, z-score = -2.90, R) >droSec1.super_4 3247134 120 - 6179234 GACAACAUCAUUCUGCUGAUCACUGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCACCCUCUGGGCAGCUUUGAGCAGAUGGAAGCCAUCCAUGUGGCGUCCACU ...........(((((.......(((..(((((...)))))..)))..(((((((((...((((((.......)).))))))))))))))))))((((.(((((....))))).)))).. ( -42.30, z-score = -1.42, R) >droSim1.chrX 2781395 120 + 17042790 GACAACAUCAUUCUGCUGAUCACUGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCACCCUCUGGGCAGCUUUGAGCAGAUGGAAGCCAUCCAUGUGGCGUCCACU ...........(((((.......(((..(((((...)))))..)))..(((((((((...((((((.......)).))))))))))))))))))((((.(((((....))))).)))).. ( -42.30, z-score = -1.42, R) >droWil1.scaffold_181096 11868758 120 + 12416693 GAUAAUAUCAUUUUGCUGAUCACCGGAACUCUGCAUCAGCGUCCCAUUUCGGAGCUAAUUCCCAAAUGCCAUCCAUUGGGCAGUUUCGAGCAAAUGGAAGCCAUCCAUGUUGCCUCGACC ..............((((((...(((....))).))))))(((.....((((((((....(((((.((.....))))))).))))))))(((((((((.....))))).))))...))). ( -33.10, z-score = -1.45, R) >droMoj3.scaffold_6328 3926860 120 + 4453435 GACAACAUCAUAUUGUUGAUCACGGGCACACUCCAUCAGCGCCCCAUCUCGGAGCUGAUACCCAAGUGUCAUCCGUUGGGUAGCUUCGAGCAGAUGGAGGCCAUCCACGUGGCCUCAACG (((((((......))))).))..((((.(.........).))))((((((((((((...((((((.((.....))))))))))))))))...))))((((((((....)))))))).... ( -47.00, z-score = -3.58, R) >droGri2.scaffold_15081 1436232 120 - 4274704 GAUAAUAUCAUAUUGCUGAUAACGGGCACACUGCAUCAGCGACCCAUCUCGGAGCUGAUACCCAAGUGCCAUCCGCUGGGCAGUUUCGAGCAGAUGGAGGCCAUCCAUGUCGCUUCGACG ............((((((((..(((.....))).)))))))).....((((((((((...((((.(((.....)))))))))))))))))...(((((.....)))))((((...)))). ( -39.60, z-score = -1.26, R) >droVir3.scaffold_13042 4662887 120 + 5191987 GAUAAUAUUAUAUUGUUGAUCACGGGCACGCUGCACCAGCGUCCCAUUUCGGAGCUGAUACCCAAAUGCCAUCCGCUGGGCAGCUUCGAGCAGAUGGAGGCGAUACAUGUGGCAUCCACG ..........(((((((.(((..(((.((((((...)))))))))..((((((((((...((((...((.....))))))))))))))))..)))...)))))))...((((...)))). ( -42.10, z-score = -1.78, R) >droPer1.super_14 1711593 120 + 2168203 GACAACAUCAUCCUGCUCAUUACGGGUACGCUGCACCAGCGUCCCAUUUCGGAGCUGAUACCCAAGUGCCAUCCGCUGGGCAGCUUCGAGCAGAUGGAGGCUAUCCAUGUGGCGUCGACG (((..(..((((.(((((.....(((.((((((...))))))))).....(((((((...((((.(((.....)))))))))))))))))))))))..)(((((....)))))))).... ( -52.30, z-score = -3.88, R) >dp4.chrXL_group1e 621785 120 - 12523060 GACAACAUCAUCCUGCUCAUUACGGGCACGCUGCACCAGCGUCCCAUUUCGGAGCUGAUACCCAAGUGCCAUCCGCUGGGCAGCUUCGAGCAGAUGGAGGCUAUCCAUGUGGCGUCGACG (((..(..((((.(((((.....(((.((((((...))))))))).....(((((((...((((.(((.....)))))))))))))))))))))))..)(((((....)))))))).... ( -52.30, z-score = -3.74, R) >droAna3.scaffold_13117 714247 120 + 5790199 GACAACAUCAUCCUGCUGAUCACCGGCACCCUGCACCAGCGCCCCAUCUCGGAGCUGAUCCCCAAGUGCCAUCCCCUGGGCAGCUUCGAGCAGAUGGAGGCCAUCCACGUCGCCUCCACG (((..........(((((.....)))))....((....))(((((((((((((((((...((((.(........).)))))))))))))...))))).))).......)))......... ( -38.90, z-score = -1.26, R) >anoGam1.chrX 13134944 120 + 22145176 GACAACAUCAUCCUGCUGAUCACGGGCACGCUGCACCAGCGCCCGAUCUCGGAGCUCAUCCCGAAGUGCCACCCGCUCGGCAGCUUCGAGCAGAUGGAGGCGAUCCACGUCGCGGCCACA ............((((.((((..((((..((((...))))))))))))((((((((....((((.(((.....))))))).)))))))))))).(((..(((((....)))))..))).. ( -46.20, z-score = -1.20, R) >apiMel3.GroupUn 397709740 120 - 399230636 GAUAAUAUUAUCUUACUAAUUACUGGUACUUUGCAUCAAAGACCAAUUUCAGAAUUGAUUCCUAAAUGUCACCCGCUUGGUAGUUUUGAACAAAUGGAAGCUAUUCAUGUAGCAGCUACA ((((....))))...........((((.(((((...)))))))))..((((((((((((........)))(((.....))))))))))))....(((..(((((....)))))..))).. ( -22.10, z-score = -0.54, R) >triCas2.ChLG5 7104776 120 - 18847211 GAUAAUAUUAUUUUACUGAUUACGGGGACGUUGCACCAGCGCCCCAUUGGGGAGCUGAUCCCGAAAUGCCACCCCUUGGGGAGCUUUGAGCAGAUGGAGGCUAUUCAUGUGGCCUCCACG .....................(((....)))(((.(.(((.(((((..(((..((............))..)))..))))).)))..).)))..((((((((((....)))))))))).. ( -46.00, z-score = -2.73, R) >consensus GACAACAUCAUUCUGCUGAUCACGGGCACGCUGCACCAGCGACCCAUCUCGGAGCUGAUCCCCAAGUGCCAUCCGCUGGGCAGCUUCGAGCAGAUGGAGGCCAUCCAUGUGGCGUCCACG .....((((....(((((.....)))))...................((((((((((...((((............))))))))))))))..))))((.(((((....))))).)).... (-24.98 = -24.25 + -0.73)


Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:12:35 2011