
| Sequence ID | dm3.chrX |
|---|---|
| Location | 3,155,847 – 3,155,981 |
| Length | 134 |
| Max. P | 0.901211 |

| Location | 3,155,847 – 3,155,981 |
|---|---|
| Length | 134 |
| Sequences | 12 |
| Columns | 154 |
| Reading direction | forward |
| Mean pairwise identity | 79.49 |
| Shannon entropy | 0.42766 |
| G+C content | 0.39954 |
| Mean single sequence MFE | -34.27 |
| Consensus MFE | -17.44 |
| Energy contribution | -17.68 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.51 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.901211 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chrX 3155847 134 + 22422827 ----------ACACACAGUUAUCCACGAUACAAGUCGCAGCUGU--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-UUUGCCACAAAAUAUUUACACAAUUGACAGCUGCUU-CUGUUCAGUG ----------.............(((...(((.(..((((((((--(((((((((..((-----(....(((((((((....))-)).)))))..))))))))..(-((((...)))))..........))))))))))))..-))))...))) ( -34.60, z-score = -2.36, R) >droPer1.super_18 1575838 124 + 1952607 --------------------ACCGACCCGCAGACUAGCAGCUGC--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-CUUGCCACAAAAUAUUUACACAACUGACAGCUGCUU-CUGUUCGGUG --------------------(((((...(((((..(((((((((--....))..(((.(-----((((.(((((((((....))-)).))))).......)).)))-...)))......................))))))))-))))))))). ( -37.80, z-score = -3.25, R) >dp4.chrXL_group1e 2675118 124 + 12523060 --------------------ACCGACCCGCAGACUCGCAGCUGC--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-CUUGCCACAAAAUAUUUACACAACUGACAGCUGCUU-CUGUUCGGUG --------------------(((((...(((((...((((((((--....))..(((.(-----((((.(((((((((....))-)).))))).......)).)))-...)))......................)))))).)-))))))))). ( -36.10, z-score = -2.91, R) >droAna3.scaffold_12613 263559 143 - 519072 AUACUCAGAUAUAUCUAGAUACUUG-AAUACAAGUCGCAGCUGU--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-UUUGCCGCAAAAUAUUUACACAAUUGACAGCUGCUU-CUUUUCAGUA ...((.(((....))))).((((.(-((...(((..((((((((--((((((..(((.(-----((((.(((((((((....))-)).))))).......)).)))-...))))))..............)))))))))))..-)))))))))) ( -35.23, z-score = -2.12, R) >droEre2.scaffold_4690 561475 134 + 18748788 ----------ACACACAGAUAUCCACGAUACAAGUCGCAGCUGU--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GAAAGCAUUUCUAUUUGUACU-UUUGCCACAAAAUAUUUACACAAUUGACAGCUGCUU-CUGCUCAGUG ----------.(((.((((.......(((....)))((((((((--(((((((((..((-----(....(((((((((....))-).))))))..))))))))..(-((((...)))))..........)))))))))))).)-)))....))) ( -34.70, z-score = -2.30, R) >droYak2.chrX 16541616 134 - 21770863 ----------ACCCACAGAUAUCCACGAUACAAGUCGCAGCUGU--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-UUUGCCACAAAAUAUUUACACAAUUGACAGCUGCUU-CUGUUCAGUG ----------....(((((.......(((....)))((((((((--(((((((((..((-----(....(((((((((....))-)).)))))..))))))))..(-((((...)))))..........)))))))))))).)-))))...... ( -35.30, z-score = -2.81, R) >droSec1.super_10 2863683 134 + 3036183 ----------ACACACAGAUAUCCACGAUACAAGUCGCAGCUGU--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-UUUGCCACAAAAUAUUUACACAAUUGACAGCUGCUU-CUGUUCAGUG ----------.((((((((.......(((....)))((((((((--(((((((((..((-----(....(((((((((....))-)).)))))..))))))))..(-((((...)))))..........)))))))))))).)-))))...))) ( -36.50, z-score = -2.93, R) >droSim1.chrX 2244175 134 + 17042790 ----------ACACACAGAUAUCCACGAUACAAGUCGCAGCUGU--UAAUGCAAGGCUA-----GUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-UUUGCCACAAAAUAUUUACACAAUUGACAGCUGCUU-CUGUUCAGUG ----------.((((((((.......(((....)))((((((((--(((((((((..((-----(....(((((((((....))-)).)))))..))))))))..(-((((...)))))..........)))))))))))).)-))))...))) ( -36.50, z-score = -2.93, R) >droVir3.scaffold_12726 2267897 136 - 2840439 ----------GUGAACAAGAUACAAGAUACAAGUUCGCAGCUGU--UAAUGCAAGGCU-----AGUCAUGUGCCUCGAUUUAUCGAAUGGCAUUUCUAUUUGUACU-UUUGGCACAAAAUAUUUACACAAUUGACAGCUGUUAGAUGUUAGAUG ----------..((((................))))((((((((--((((........-----.....((((((.........(((((((.....)))))))....-...)))))).............))))))))))))............. ( -31.75, z-score = -0.63, R) >droMoj3.scaffold_6308 1080290 128 - 3356042 ------------------GAUACAAGAUACAAGU-CGCAGCUGU--UAAUGCACGUCUGU---AGUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUACU-UUUGCCACCAAAUAUUUACACAAUUGACAGCUGCUCGCUGCGCGUUG ------------------.......(((....))-)((((((((--((((........((---((....(((((((((....))-)).)))))..)))).((((..-((((....))))....))))..)))))))))))).(((...)))... ( -31.90, z-score = -0.79, R) >droGri2.scaffold_14853 5825029 138 - 10151454 ----------AUAUACAAUCUUCAACAUACAAGA-CCCAGCUGU--UAAUGCAAGGCGGUU-GGGUCAUGUGCUUCGAUUUAUC-AACAGUAUUUCUAUUUGUACU-UUUGGCACAAAAUAUUUACACAAUUGACAGCUGCUCGAUGCCCGCUG ----------...........((((.......((-(((((((((--(.......)))))))-))))).(((((..........(-((.(((((........)))))-.))))))))..............))))((((.((.....))..)))) ( -35.40, z-score = -2.17, R) >droWil1.scaffold_180588 995453 116 + 1294757 --------------------------GAAACAAGUCACAGCUGUGUUAAUGCAAGGCUAAAUAAGUCAUGUGCUUCGAUUUAUC-GACAGCAUUUCUAUUUGUUCAAUUUGGCACAAAAUAUUUACACAAUUGACAGCUGUUG----------- --------------------------..........((((((((((....))...(((((((((((...(((((((((....))-)).)))))....)))))).....)))))....................))))))))..----------- ( -25.50, z-score = -0.76, R) >consensus __________ACACACAGAUAUCCACGAUACAAGUCGCAGCUGU__UAAUGCAAGGCUA_____GUCAUGUGCUUCGAUUUAUC_GACAGCAUUUCUAUUUGUACU_UUUGCCACAAAAUAUUUACACAAUUGACAGCUGCUU_CUGUUCAGUG ....................................((((((((.....((((((((.......)))..(((((((((....)).)).)))))......))))).....................((....))))))))))............. (-17.44 = -17.68 + 0.24)

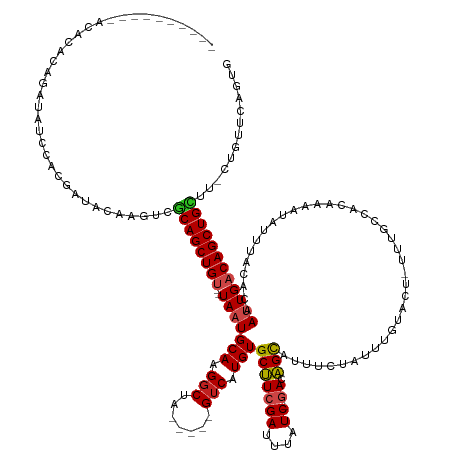
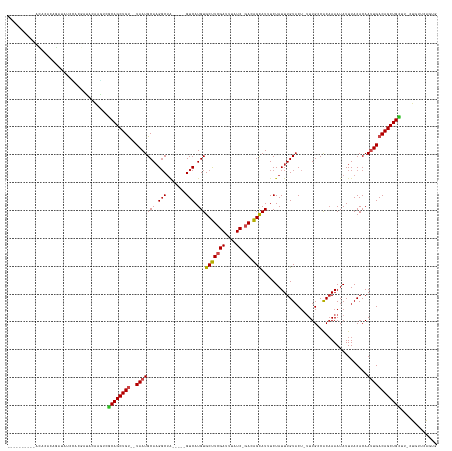
Generated by rnazCluster.pl (part of RNAz 1.0) on Wed Apr 20 01:11:02 2011